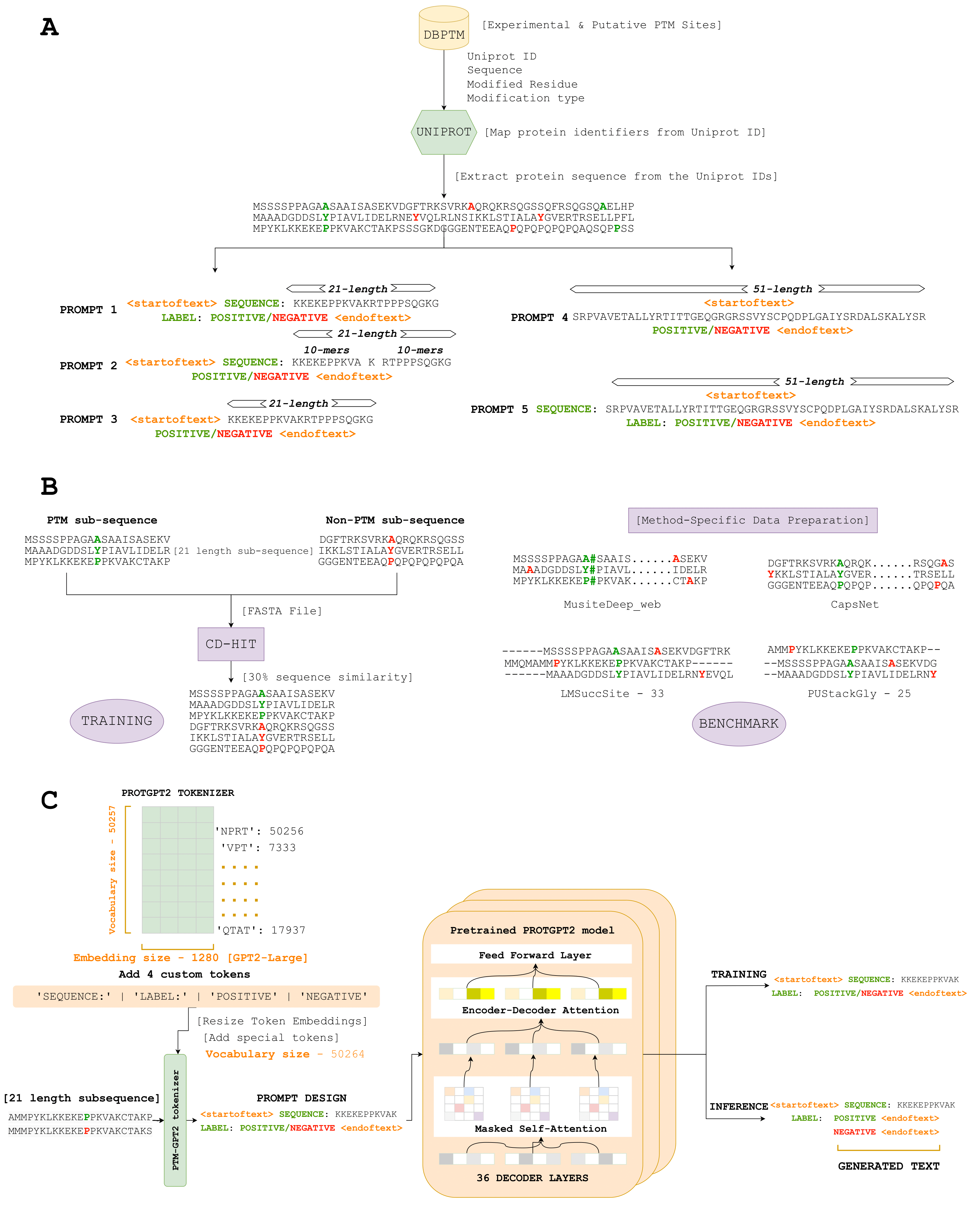
هنا ، نقدم PTMGPT2 ، مجموعة من النماذج القادرة على توليد الرموز التي تشير إلى تسلسل البروتين المعدل ، حاسم لتحديد مواقع PTM. في صميم هذا النظام الأساسي هو Protgpt2 ، نموذج محول التلقائي. لقد قمنا بتكييف protgpt2 ، واستخدامه كنموذج مدرب مسبقًا ، وقمنا بزيادة ضبطه لمهمة spe cific المتمثلة في إنشاء ملصقات تصنيف لنوع PTM معين. بشكل فريد ، يستخدم PTMGPT2 بنية فك التشفير فقط ، مما يلغي الحاجة إلى رأس التحويل الخاص بالمهمة أثناء التدريب. بدلاً من ذلك ، تعمل الطبقة النهائية من وحدة فك الترميز كإسقاط مرة أخرى إلى مساحة المفردات ، مما يولد بشكل فعال الرمز الممكنة التالية بناءً على الأنماط المستفادة بين الرموز في موجه الإدخال.
الرابط - (https://nsclbio.jbnu.ac.kr/gpt_model/)
اتصل بنا مباشرة على [email protected] للتنبؤات السائبة والنماذج المدربة
الرابط - (https://nsclbio.jbnu.ac.kr/tools/ptmgpt2/)
رابط - (https://doi.org/10.5281/zenodo.11371883)
رابط - (https://zenodo.org/records/11362322)
الرابط - (https://doi.org/10.5281/zenodo.11377398)
بيثون 3.11.3
المحولات 4.29.2
Scikit-Learn 1.2.2
Pytorch 2.0.1
Pytorch-Cuda 11.7
• النموذج: يستضيف هذا المجلد نموذج عينة مصمم للتنبؤ بمواقع PTM من تسلسل البروتين المعطى ، مما يوضح تطبيق PTMGPT2.
• Tokenizer: يحتوي هذا المجلد على عينة رمزية مسؤولة عن تسلسل البروتين الرمزي ، بما في ذلك الرموز المميزة المصنوعة يدويًا للأحماض الأمينية المحددة أو الزخارف.
• Interference.ipynb: يوفر هذا الملف رمزًا قابلًا للتنفيذ لتطبيق نموذج PTMPPT2 و Tokenizer للتنبؤ بمواقع PTM ، ويكون بمثابة دليل عملي للمستخدمين لتطبيق النموذج على مجموعات البيانات الخاصة بهم.