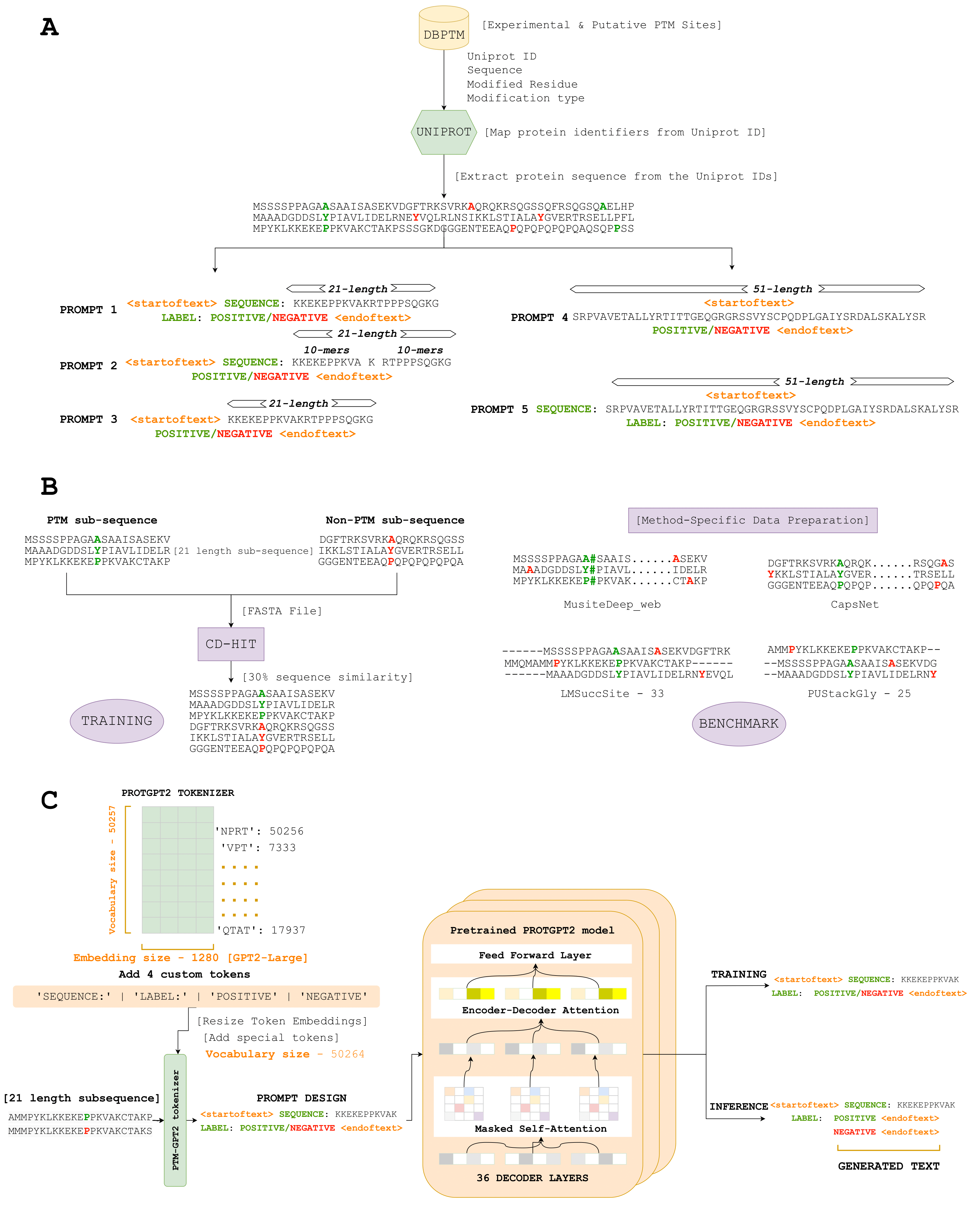
Hier führen wir PTMGPT2 ein, eine Reihe von Modellen, die Token erzeugen können, die modifizierte Proteinsequenzen bedeuten, die für die Identifizierung von PTM -Stellen entscheidend sind. Im Kern dieser Plattform steht ProtGPT2, ein autoregressives Transformatormodell. Wir haben ProGGPT2 angepasst, indem wir es als vorgebildetes Modell verwendet und es für die Spe-Cific-Aufgabe, Klassifizierungsbezeichnungen für einen bestimmten PTM-Typ zu generieren, weiter abgestimmt. Eindeutig verwendet PTMGPT2 eine Nur-Decoder-Architektur, wodurch die Notwendigkeit eines aufgabenspezifischen Klassifizierungskopfs während des Trainings beseitigt wird. Stattdessen fungiert die endgültige Schicht des Decoders als Projektion zurück zum Wortschatz und erzeugt das nächste mögliche Token, das auf den gelernten Mustern unter Token in der Eingabeaufforderung basiert.
Link - (https://nsclbio.jbnu.ac.kr/gpt_model/)
Kontaktieren Sie uns direkt unter [email protected] für Massenvorhersagen und geschulte Modelle
Link - (https://nsclbio.jbnu.ac.kr/tools/ptmgpt2/)
Link - (https://doi.org/10.5281/zenodo.11371883)
Link - (https://zenodo.org/records/11362322)
Link - (https://doi.org/10.5281/zenodo.11377398)
Python 3.11.3
Transformatoren 4.29.2
Scikit-Learn 1.2.2
Pytorch 2.0.1
Pytorch-Cuda 11.7
• Modell: In diesem Ordner wird ein Beispielmodell veranstaltet, das PTM -Stellen aus angegebenen Proteinsequenzen vorhergesagt hat, die die Anwendung von PTMGPT2 veranschaulicht.
• Tokenizer: Dieser Ordner enthält einen Proben -Tokenizer, der für die Tokenisierungsproteinsequenzen verantwortlich ist, einschließlich handgefertigter Token für bestimmte Aminosäuren oder Motive.
• Inference.IPYNB: Diese Datei bietet ausführbaren Code für die Anwendung von PTMGPT2 -Modell und Tokenizer zur Vorhersage von PTM -Sites und dient als praktische Leitfaden für Benutzer, um das Modell auf ihre Datensätze anzuwenden.