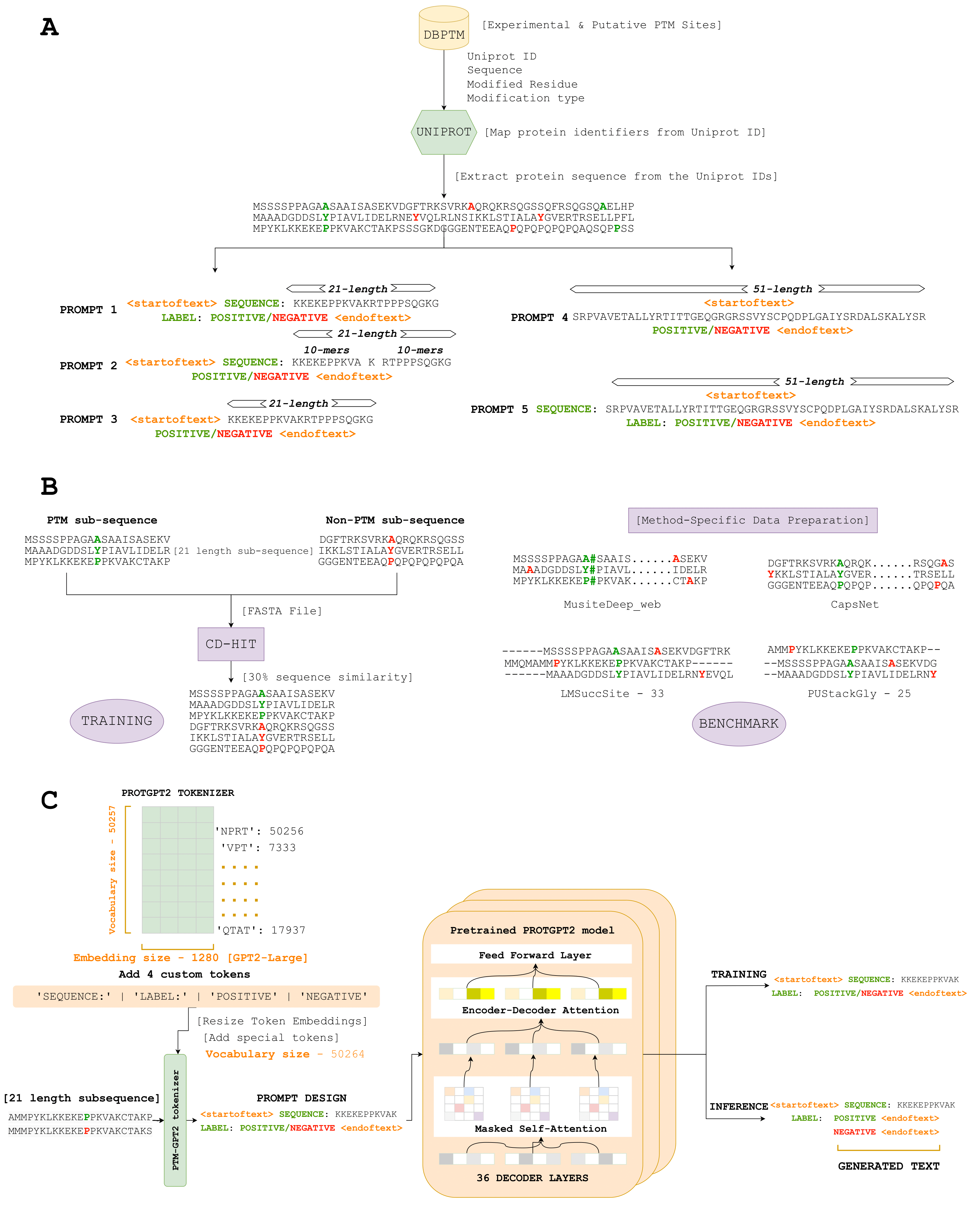
Aqui, introduzimos o PTMGPT2, um conjunto de modelos capazes de gerar tokens que significam sequências de proteínas modificadas, cruciais para identificar locais de PTM. No centro desta plataforma está o ProtGPT2, um modelo de transformador autoregressivo. Adaptamos o ProtGPT2, utilizando-o como um modelo pré-treinado e mais ajustado para a tarefa específica de gerar rótulos de classificação para um determinado tipo PTM. Singularmente, o PTMGPT2 utiliza uma arquitetura somente para decodificador, que elimina a necessidade de uma cabeça de clasificação específica da tarefa durante o treinamento. Em vez disso, a camada final do decodificador funciona como uma projeção de volta ao espaço do vocabulário, gerando efetivamente o próximo token possível com base nos padrões aprendidos entre os tokens no prompt de entrada.
Link - (https://nsclbio.jbnu.ac.kr/gpt_model/)
Entre em contato conosco diretamente em [email protected] para previsões em massa e modelos treinados
Link - (https://nsclbio.jbnu.ac.kr/tools/ptmgpt2/)
Link - (https://doi.org/10.5281/zenodo.11371883)
Link - (https://zenodo.org/records/11362322)
Link - (https://doi.org/10.5281/zenodo.11377398)
Python 3.11.3
Transformers 4.29.2
Scikit-Learn 1.2.2
Pytorch 2.0.1
Pytorch-Cuda 11.7
• Modelo: Esta pasta hospeda um modelo de amostra projetado para prever sites PTM de determinadas sequências de proteínas, ilustrando a aplicação do PTMGPT2.
• Tokenizer: Esta pasta contém um tokenizador de amostra responsável por sequências de proteínas de tokenização, incluindo tokens artesanais para aminoácidos ou motivos específicos.
• Inferência.ipynb: Este arquivo fornece código executável para aplicar o modelo PTMGPT2 e o tokenizador para prever sites PTM, servindo como um guia prático para os usuários aplicarem o modelo aos seus conjuntos de dados.