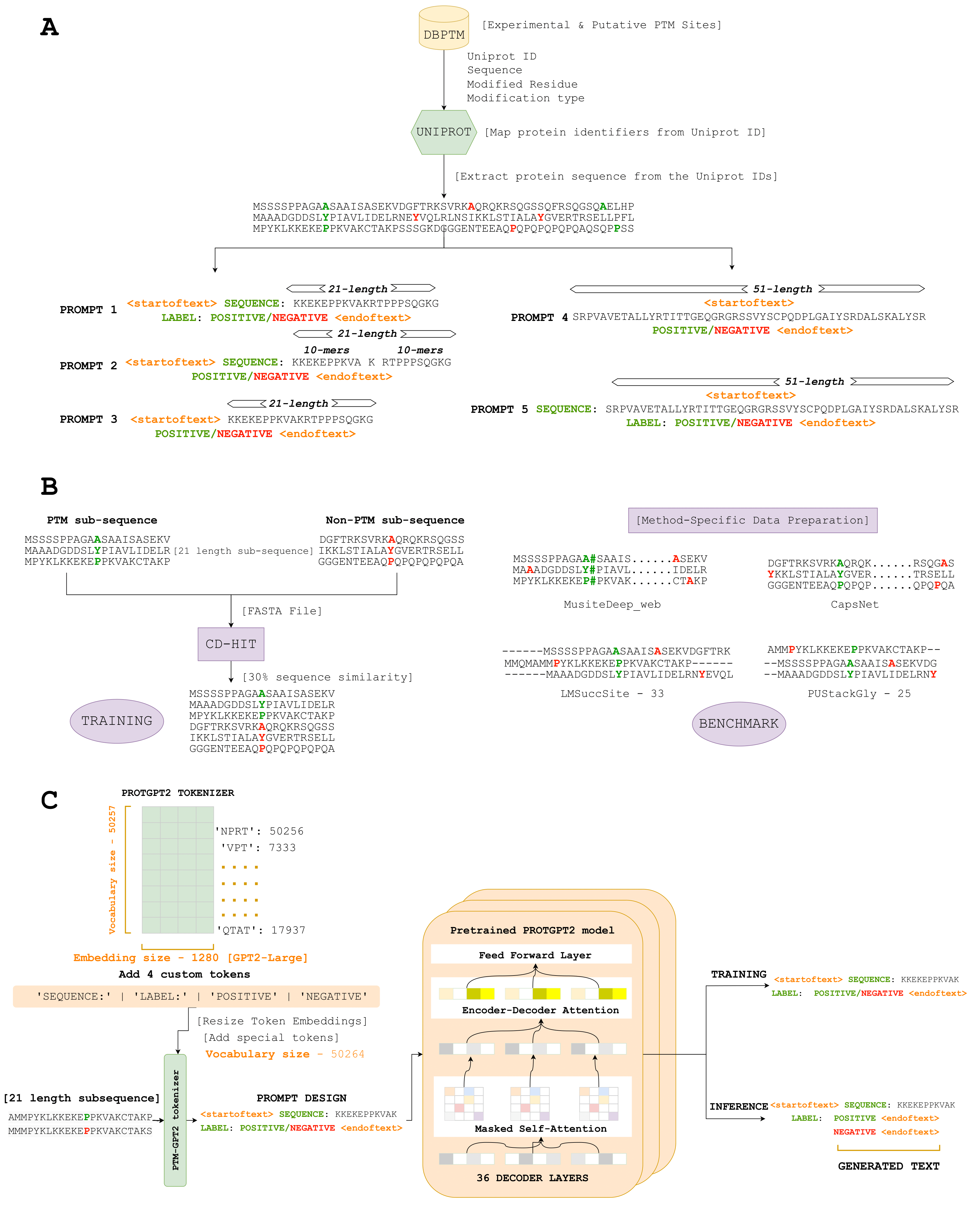
Aquí, presentamos PTMGPT2, un conjunto de modelos capaces de generar tokens que significan secuencias de proteínas modificadas, cruciales para identificar sitios PTM. En el núcleo de esta plataforma está Protgpt2, un modelo de transformador autorregresivo. Hemos adaptado ProTgpt2, utilizándolo como un modelo previamente capacitado, y lo ajustamos aún más para la tarea especial de generar etiquetas de clasificación para un tipo PTM dado. De manera única, PTMPPT2 utiliza una arquitectura de decodificador solo, que elimina la necesidad de un cabezal de clasificación específico de tarea durante el entrenamiento. En cambio, la capa final del decodificador funciona como una proyección al espacio de vocabulario, generando efectivamente el siguiente token posible en función de los patrones aprendidos entre los tokens en el indicador de entrada.
Enlace - (https://nsclbio.jbnu.ac.kr/gpt_model/)
Contáctenos directamente en [email protected] para predicciones masivas y modelos capacitados
Enlace - (https://nsclbio.jbnu.ac.kr/tools/ptmgpt2/)
Enlace - (https://doi.org/10.5281/zenodo.11371883)
Enlace - (https://zenodo.org/records/11362322)
Enlace - (https://doi.org/10.5281/zenodo.11377398)
Python 3.11.3
Transformers 4.29.2
Scikit-Learn 1.2.2
Pytorch 2.0.1
Pytorch-Cuda 11.7
• Modelo: esta carpeta aloja un modelo de muestra diseñado para predecir sitios PTM a partir de secuencias de proteínas dadas, ilustrando la aplicación de PTMPT2.
• Tokenizer: esta carpeta contiene un tokenizador de muestra responsable de la tokenización de secuencias de proteínas, incluidos los tokens hechos a mano para aminoácidos o motivos específicos.
• Inferencia.ipynb: este archivo proporciona código ejecutable para aplicar el modelo PTMPPT2 y el tokenizador para predecir los sitios PTM, que sirve como una guía práctica para que los usuarios apliquen el modelo a sus conjuntos de datos.