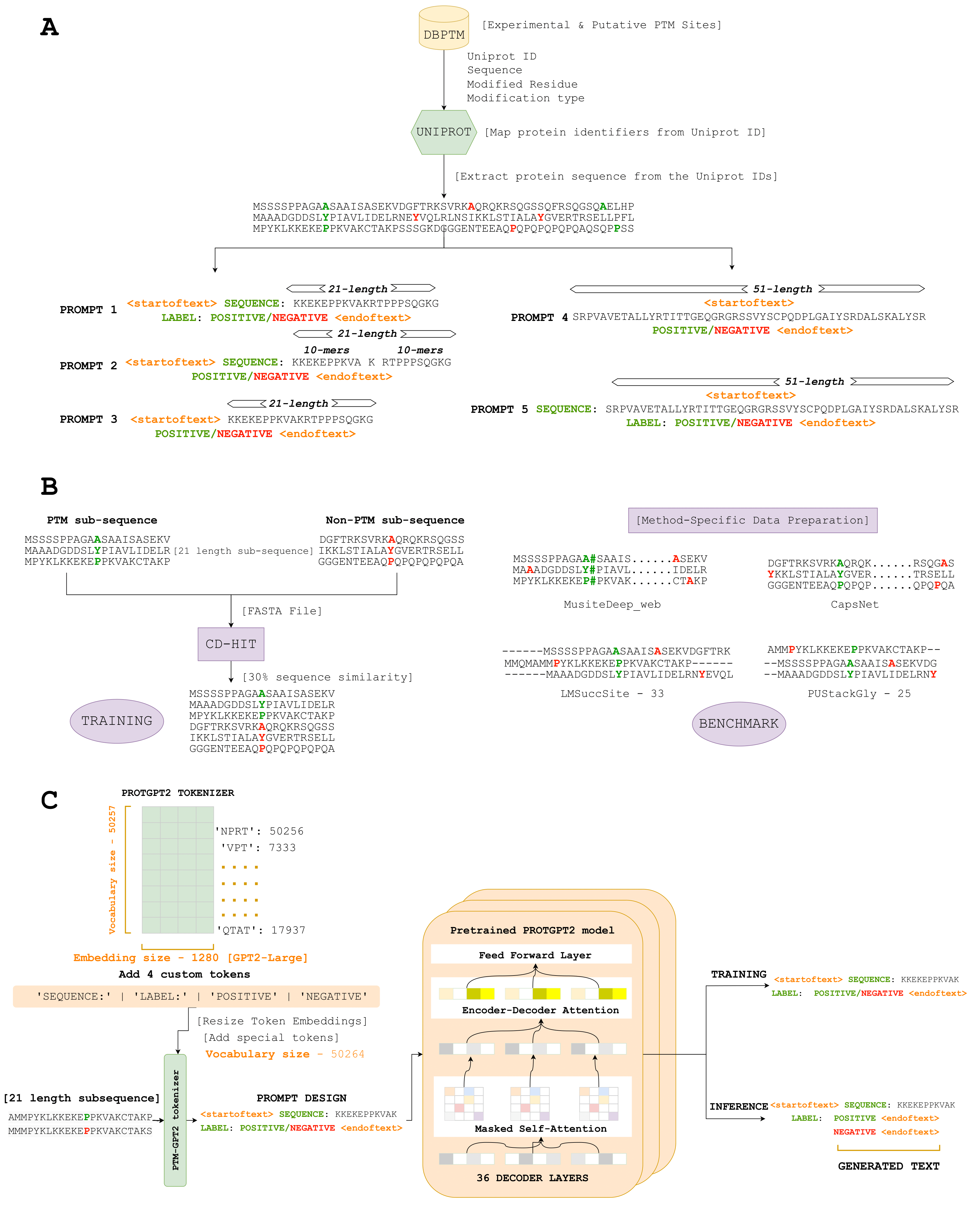
ここでは、PTMサイトを特定するために重要な修正タンパク質配列を示すトークンを生成できる一連のモデルであるPTMGPT2を導入します。このプラットフォームのコアには、自己回帰トランスモデルであるProtgpt2があります。 ProtGPT2を訓練を受けたモデルとして利用し、特定のPTMタイプの分類ラベルを生成するという特別なタスクのためにさらに微調整しました。ユニークなことに、PTMGPT2はデコーダーのみのアーキテクチャを使用しており、トレーニング中のタスク固有の継続ヘッドの必要性を排除します。代わりに、デコーダーの最終層は、語彙空間への投影として機能し、入力プロンプトのトークン間の学習パターンに基づいて次の可能なトークンを効果的に生成します。
リンク - (https://nsclbio.jbnu.ac.kr/gpt_model/)
バルク予測と訓練されたモデルについては、 palisthashrestha7@ jbnu.ac.krに直接お問い合わせください
リンク - (https://nsclbio.jbnu.ac.kr/tools/ptmgpt2/)
リンク - (https://doi.org/10.5281/zenodo.11371883)
リンク - (https://zenodo.org/records/11362322)
リンク - (https://doi.org/10.5281/zenodo.11377398)
Python 3.11.3
トランス4.29.2
Scikit-Learn1.2.2
Pytorch 2.0.1
Pytorch-Cuda 11.7
•モデル:このフォルダーは、特定のタンパク質配列からPTMサイトを予測するように設計されたサンプルモデルをホストし、PTMGPT2のアプリケーションを示しています。
•トークン剤:このフォルダーには、特定のアミノ酸またはモチーフ用の手作りのトークンを含む、タンパク質配列のトークン化に関与するサンプルトークネザーが含まれています。
•Inference.ipynb:このファイルは、PTMGPT2モデルとトークンザーを適用するための実行可能なコードを提供し、PTMサイトを予測し、ユーザーがモデルをデータセットに適用するための実用的なガイドとして機能します。