Мартен Лиенен, Дэвид Людке, Ян Хансен-Палмус, Стефан Гюннеманн

Этот репозиторий содержит код, используемый для получения результатов в нашей статье: OpenReview, Arxiv.
Помимо модели, загрузки данных и кода обучения, этот репозиторий также содержит код для настройки и запуска OpenFOAM и постороннего обработки его выходов. Эти инструменты могут быть чрезвычайно полезной отправной точкой для других исследователей в этой области. В частности, есть
# Clone the repository
git clone https://github.com/martenlienen/generative-turbulence.git
# Change into the repository
cd generative-turbulence
# Install package editably with dependencies
pip install -e .
# If you need a specific pytorch version, e.g. CPU-only or an older CUDA version, check
#
# https://pytorch.org/get-started/locally/
#
# and run, for example,
#
# pip install torch --extra-index-url https://download.pytorch.org/whl/cu117
#
# before installing this package. Набор данных форм размещен в Университетской библиотеке ТУМ. Чтобы загрузить его, либо следуйте инструкциям на их странице, либо выполните следующие шаги. Во -первых, загрузите файлы.
# Download all archives to data/shapes/download
scripts/download-dataset.sh Обратите внимание, что это может занять много времени, так как обработанные данные составляют примерно 2 ТБ. Сценарий использует rsync , поэтому вы можете возобновить частичные загрузки. Если вы также хотите загрузить данные Crave OpenFoam Case, вместо этого запустите
scripts/download-dataset.sh --with-rawПосле загрузки архивов Invididual вам необходимо извлечь файлы. Следующий сценарий делает это для вас
scripts/extract-dataset.shПосле этого вы можете начать обучение модели, как описано ниже.
Следующий фрагмент загружает данные из любого из файлов data.h5 в наборе данных, чтобы вы могли исследовать и экспериментировать.
import numpy as np
import h5py as h5
def load_data ( path , idx , features = [ "u" , "p" ]):
"""Load data from a data.h5 file into an easily digestible matrix format.
Arguments
---------
path
Path to a data.h5 file in the `shapes` dataset
idx
Index or indices of sample to load. Can be a number, list, boolean mask or a slice.
features
Features to load. By default loads only velocity and pressure but you can also
access the LES specific k and nut variables.
Returns
-------
t: np.ndarray of shape T
Time steps of the loaded data frames
data_3d: np.ndarray of shape T x W x H x D x F
3D data with all features concatenated in the order that they are requested, i.e.
in the default case the first 3 features will be the velocity vector and the fourth
will be the pressure
inside_mask: np.ndarray of shape W x H x D
Boolean mask that marks the inside cells of the domain, i.e. cells that are not part
of walls, inlets or outlets
boundary_masks: dict of str to nd.ndarray of shape W x H x D
Masks that mark cells belonging to each type of boundary
boundary_values: dict[str, dict[str, np.ndarray]]
Prescribed values for variables and boundaries with Dirichlet boundary conditions
"""
with h5 . File ( path , mode = "r" ) as f :
t = np . array ( f [ "data/times" ])
cell_data = np . concatenate ([ np . atleast_3d ( f [ "data" ][ name ][ idx ]) for name in features ], axis = - 1 )
padded_cell_counts = np . array ( f [ "grid/cell_counts" ])
cell_idx = np . array ( f [ "grid/cell_idx" ])
n_steps , n_features = cell_data . shape [ 0 ], cell_data . shape [ - 1 ]
data_3d = np . zeros (( n_steps , * padded_cell_counts , n_features ))
data_3d . reshape (( n_steps , - 1 , n_features ))[:, cell_idx ] = cell_data
inside_mask = np . zeros ( padded_cell_counts , dtype = bool )
inside_mask . reshape ( - 1 )[ cell_idx ] = 1
boundary_masks = { name : np . zeros ( padded_cell_counts , dtype = bool ) for name in f [ "grid/boundaries" ]. keys ()}
for name , mask in boundary_masks . items ():
mask . reshape ( - 1 )[ np . array ( f [ "grid/boundaries" ][ name ])] = 1
boundary_values = {
ft : {
name : np . atleast_1d ( desc [ "value" ])
for name , desc in f [ "boundary-conditions" ][ ft ]. items ()
if desc . attrs [ "type" ] == "fixed-value"
}
for ft in features
}
return t , data_3d , inside_mask , boundary_masks , boundary_valuesВы можете использовать его как
from matplotlib . pyplot import matshow
path = "data/shapes/data/2x2-large/data.h5"
t , data_3d , inside_mask , boundary_masks , boundary_values = load_data ( path , [ 50 , 300 ])
matshow ( np . linalg . norm ( data_3d [ - 1 , :, :, 20 , : 3 ], axis = - 1 ). T )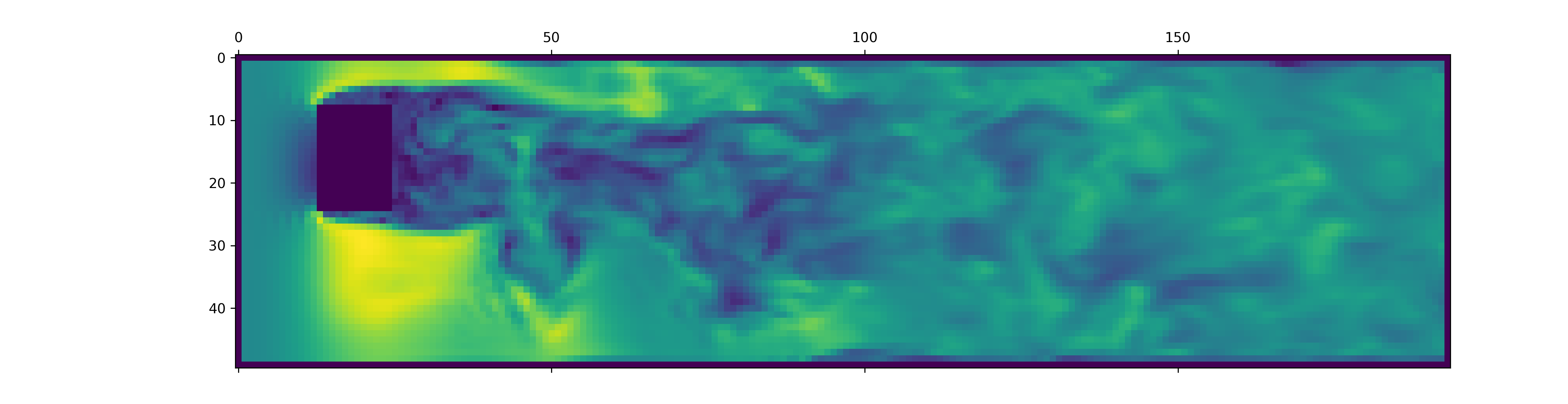
Файлы data.h5 содержат больше информации, чем этот фрагмент загружается. Чтобы изучить, что еще доступно, застрятся в нашем загрузке данных.
Чтобы сгенерировать данные для новых симуляций OpenFOAM, сначала убедитесь, что вы установили дополнительные зависимости и just имели доступность:
pip install -e " .[data] " Если вы не хотите использовать just , вы также можете прочитать justfile и самостоятельно запустить команды.
Начните с создания контейнера Docker с установленным OpenFoam:
just of-dockerТеперь генерируйте кучу новых случаев. Например, следующее устанавливает все случаи OpenFOAM (симуляции) из нашего набора данных:
./scripts/generate-shapes.py data/shapesКонечно, вы можете адаптировать скрипт для создания других форм или совершенно новых наборов данных.
Теперь вы можете решить чехол (запустить симуляцию) с помощью OpenFoAm
just of-solve path/to/caseили отправьте целую кучу из собственного кластера Slurm:
./scripts/solve-slurm.py data/shapes/data/ * /caseПосле этого примените постобработку, например, преобразование HDF5, к каждому моделированию, например,
just postprocess data/shapes/data/2x2Наконец, вычислите статистику обучающего набора для нормализации функций:
./scripts/dataset-stats.py data/shapes Чтобы начать обучение, позвоните train.py с помощью ваших настроек, например,
./train.py data.batch_size=128 Сценарий обучения использует HYDRA для конфигурации, поэтому ознакомьтесь с файлами в каталоге config , чтобы узнать обо всех доступных настройках.
Чтобы запустить эксперименты из бумаги, выполнить
./train.py -cn shapes_experiment -m который начинает обучение с настройками в config/shapes_experiment.yaml . Если у вас нет доступного кластера Slurm, удалите настройки, связанные с launcher .
Если вы опираетесь на эту работу, пожалуйста, укажите нашу статью следующим образом.
@inproceedings{lienen2024zero,
title = {From {{Zero}} to {{Turbulence}}: {{Generative Modeling}} for {{3D Flow Simulation}}},
author = {Lienen, Marten and L{"u}dke, David and {Hansen-Palmus}, Jan and G{"u}nnemann, Stephan},
booktitle = {International {{Conference}} on {{Learning Representations}}},
year = {2024},
}