Marten Lienen, David Lüdke, Jan Hansen-Palmus, Stephan Günnemann

이 저장소에는 논문에서 결과를 생성하는 데 사용되는 코드 인 OpenReview, Arxiv가 포함되어 있습니다.
모델, 데이터로드 및 교육 코드 외에도이 저장소에는 OpenFOAM을 구성하고 실행하고 출력을 사후 처리하는 코드도 포함되어 있습니다. 이 도구는 현장의 다른 연구자들에게 매우 유용한 출발점이 될 수 있습니다. 특히, 거기에 있습니다
# Clone the repository
git clone https://github.com/martenlienen/generative-turbulence.git
# Change into the repository
cd generative-turbulence
# Install package editably with dependencies
pip install -e .
# If you need a specific pytorch version, e.g. CPU-only or an older CUDA version, check
#
# https://pytorch.org/get-started/locally/
#
# and run, for example,
#
# pip install torch --extra-index-url https://download.pytorch.org/whl/cu117
#
# before installing this package. Shapes 데이터 세트는 Tum University Library에서 호스팅됩니다. 다운로드하려면 페이지의 지침을 따르거나 다음 단계를 실행하십시오. 먼저 파일을 다운로드하십시오.
# Download all archives to data/shapes/download
scripts/download-dataset.sh 처리 된 데이터가 대략 2TB이므로 시간이 오래 걸릴 수 있습니다. 스크립트는 rsync 사용하므로 부분 다운로드를 재개 할 수 있습니다. RAW OpenFoam 케이스 데이터를 다운로드하려면 대신 실행하십시오.
scripts/download-dataset.sh --with-rawIndidual Archives를 다운로드 한 후 파일을 추출해야합니다. 다음 스크립트가 그렇게합니다
scripts/extract-dataset.sh그 후 아래에 설명 된대로 모델을 훈련하기 시작할 수 있습니다.
다음 스 니펫은 데이터 세트의 모든 data.h5 파일에서 데이터를로드하여 탐색하고 실험 할 수 있습니다.
import numpy as np
import h5py as h5
def load_data ( path , idx , features = [ "u" , "p" ]):
"""Load data from a data.h5 file into an easily digestible matrix format.
Arguments
---------
path
Path to a data.h5 file in the `shapes` dataset
idx
Index or indices of sample to load. Can be a number, list, boolean mask or a slice.
features
Features to load. By default loads only velocity and pressure but you can also
access the LES specific k and nut variables.
Returns
-------
t: np.ndarray of shape T
Time steps of the loaded data frames
data_3d: np.ndarray of shape T x W x H x D x F
3D data with all features concatenated in the order that they are requested, i.e.
in the default case the first 3 features will be the velocity vector and the fourth
will be the pressure
inside_mask: np.ndarray of shape W x H x D
Boolean mask that marks the inside cells of the domain, i.e. cells that are not part
of walls, inlets or outlets
boundary_masks: dict of str to nd.ndarray of shape W x H x D
Masks that mark cells belonging to each type of boundary
boundary_values: dict[str, dict[str, np.ndarray]]
Prescribed values for variables and boundaries with Dirichlet boundary conditions
"""
with h5 . File ( path , mode = "r" ) as f :
t = np . array ( f [ "data/times" ])
cell_data = np . concatenate ([ np . atleast_3d ( f [ "data" ][ name ][ idx ]) for name in features ], axis = - 1 )
padded_cell_counts = np . array ( f [ "grid/cell_counts" ])
cell_idx = np . array ( f [ "grid/cell_idx" ])
n_steps , n_features = cell_data . shape [ 0 ], cell_data . shape [ - 1 ]
data_3d = np . zeros (( n_steps , * padded_cell_counts , n_features ))
data_3d . reshape (( n_steps , - 1 , n_features ))[:, cell_idx ] = cell_data
inside_mask = np . zeros ( padded_cell_counts , dtype = bool )
inside_mask . reshape ( - 1 )[ cell_idx ] = 1
boundary_masks = { name : np . zeros ( padded_cell_counts , dtype = bool ) for name in f [ "grid/boundaries" ]. keys ()}
for name , mask in boundary_masks . items ():
mask . reshape ( - 1 )[ np . array ( f [ "grid/boundaries" ][ name ])] = 1
boundary_values = {
ft : {
name : np . atleast_1d ( desc [ "value" ])
for name , desc in f [ "boundary-conditions" ][ ft ]. items ()
if desc . attrs [ "type" ] == "fixed-value"
}
for ft in features
}
return t , data_3d , inside_mask , boundary_masks , boundary_values당신은 그것을 사용할 수 있습니다
from matplotlib . pyplot import matshow
path = "data/shapes/data/2x2-large/data.h5"
t , data_3d , inside_mask , boundary_masks , boundary_values = load_data ( path , [ 50 , 300 ])
matshow ( np . linalg . norm ( data_3d [ - 1 , :, :, 20 , : 3 ], axis = - 1 ). T )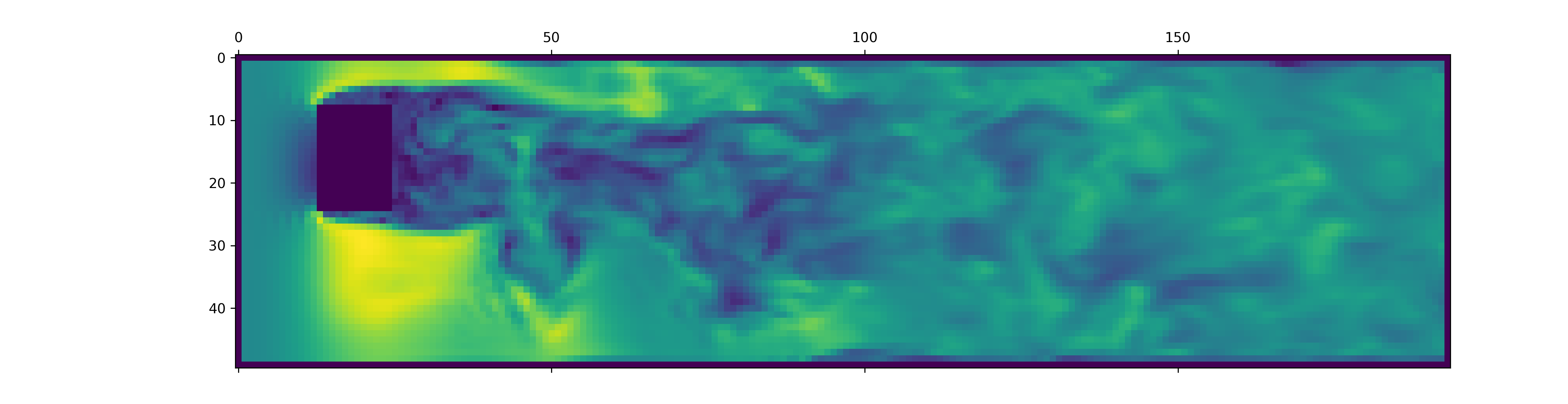
data.h5 파일에는이 스 니펫 하중보다 더 많은 정보가 포함되어 있습니다. 사용 가능한 다른 것을 탐색하려면 데이터 로더를 찌르십시오.
새로운 OpenFoam 시뮬레이션에 대한 데이터를 생성하려면 먼저 추가 종속성을 설치하고 just 수 있는지 확인하십시오.
pip install -e " .[data] " just 사용하고 싶지 않다면 justfile 을 읽고 명령을 직접 실행할 수도 있습니다.
OpenFOAM이 설치된 Docker 컨테이너를 작성하여 시작하십시오.
just of-docker이제 많은 새로운 사례를 생성하십시오. 예를 들어, 다음은 데이터 세트에서 모든 OpenFoam 케이스 (시뮬레이션)를 설정합니다.
./scripts/generate-shapes.py data/shapes물론, 스크립트를 조정하여 다른 모양 또는 완전히 새로운 데이터 세트를 만들 수 있습니다.
이제 OpenFoam으로 현지에서 케이스 (시뮬레이션 실행)를 해결할 수 있습니다.
just of-solve path/to/case또는 전체 무리를 자신의 Slurm 클러스터에 제출하십시오.
./scripts/solve-slurm.py data/shapes/data/ * /case그런 다음 후 처리 (예 : HDF5 변환)를 각 시뮬레이션에 적용합니다.
just postprocess data/shapes/data/2x2마지막으로 기능 정규화를위한 교육 세트 통계를 계산합니다.
./scripts/dataset-stats.py data/shapes 교육을 시작하려면 예를 들어 설정으로 train.py 전화하십시오.
./train.py data.batch_size=128 교육 스크립트는 구성을 위해 Hydra를 사용하므로 config 디렉토리의 파일을 확인하여 사용 가능한 모든 설정에 대해 알아보십시오.
논문에서 실험을 다시 실행하려면 실행하십시오
./train.py -cn shapes_experiment -m config/shapes_experiment.yaml 의 설정으로 교육을 시작합니다. 사용 가능한 Slurm 클러스터가없는 경우 launcher 와 관련된 설정을 제거하십시오.
이 작업을 기반으로한다면 다음과 같이 우리의 논문을 인용하십시오.
@inproceedings{lienen2024zero,
title = {From {{Zero}} to {{Turbulence}}: {{Generative Modeling}} for {{3D Flow Simulation}}},
author = {Lienen, Marten and L{"u}dke, David and {Hansen-Palmus}, Jan and G{"u}nnemann, Stephan},
booktitle = {International {{Conference}} on {{Learning Representations}}},
year = {2024},
}