Marten Lienen, David Lüdke, Jan Hansen-Palmus, Stephan Günnemann

Este repositorio contiene el código utilizado para producir los resultados en nuestro documento: OpenReview, ARXIV.
Además del modelo, el código de carga y capacitación de datos, este repositorio también contiene código para configurar y ejecutar OpenFoam y postprocesar sus salidas. Estas herramientas podrían ser un punto de partida inmensamente útil para otros investigadores en el campo. En particular, hay
# Clone the repository
git clone https://github.com/martenlienen/generative-turbulence.git
# Change into the repository
cd generative-turbulence
# Install package editably with dependencies
pip install -e .
# If you need a specific pytorch version, e.g. CPU-only or an older CUDA version, check
#
# https://pytorch.org/get-started/locally/
#
# and run, for example,
#
# pip install torch --extra-index-url https://download.pytorch.org/whl/cu117
#
# before installing this package. El conjunto de datos de formas está alojado en la Biblioteca de la Universidad Tum. Para descargarlo, siga las instrucciones en su página o ejecute los siguientes pasos. Primero, descargue los archivos.
# Download all archives to data/shapes/download
scripts/download-dataset.sh Tenga en cuenta que esto puede llevar mucho tiempo ya que los datos procesados son aproximadamente 2TB. El script usa rsync , por lo que puede reanudar descargas parciales. Si también desea descargar los datos de la caja OpenFoam sin procesar, ejecute en su lugar
scripts/download-dataset.sh --with-rawDespués de descargar los archivos Invididual, debe extraer los archivos. El siguiente script lo hace para ti
scripts/extract-dataset.shLuego, puede comenzar a entrenar el modelo como se describe a continuación.
El siguiente fragmento carga datos de cualquiera de los archivos data.h5 en el conjunto de datos para que pueda explorar y experimentar.
import numpy as np
import h5py as h5
def load_data ( path , idx , features = [ "u" , "p" ]):
"""Load data from a data.h5 file into an easily digestible matrix format.
Arguments
---------
path
Path to a data.h5 file in the `shapes` dataset
idx
Index or indices of sample to load. Can be a number, list, boolean mask or a slice.
features
Features to load. By default loads only velocity and pressure but you can also
access the LES specific k and nut variables.
Returns
-------
t: np.ndarray of shape T
Time steps of the loaded data frames
data_3d: np.ndarray of shape T x W x H x D x F
3D data with all features concatenated in the order that they are requested, i.e.
in the default case the first 3 features will be the velocity vector and the fourth
will be the pressure
inside_mask: np.ndarray of shape W x H x D
Boolean mask that marks the inside cells of the domain, i.e. cells that are not part
of walls, inlets or outlets
boundary_masks: dict of str to nd.ndarray of shape W x H x D
Masks that mark cells belonging to each type of boundary
boundary_values: dict[str, dict[str, np.ndarray]]
Prescribed values for variables and boundaries with Dirichlet boundary conditions
"""
with h5 . File ( path , mode = "r" ) as f :
t = np . array ( f [ "data/times" ])
cell_data = np . concatenate ([ np . atleast_3d ( f [ "data" ][ name ][ idx ]) for name in features ], axis = - 1 )
padded_cell_counts = np . array ( f [ "grid/cell_counts" ])
cell_idx = np . array ( f [ "grid/cell_idx" ])
n_steps , n_features = cell_data . shape [ 0 ], cell_data . shape [ - 1 ]
data_3d = np . zeros (( n_steps , * padded_cell_counts , n_features ))
data_3d . reshape (( n_steps , - 1 , n_features ))[:, cell_idx ] = cell_data
inside_mask = np . zeros ( padded_cell_counts , dtype = bool )
inside_mask . reshape ( - 1 )[ cell_idx ] = 1
boundary_masks = { name : np . zeros ( padded_cell_counts , dtype = bool ) for name in f [ "grid/boundaries" ]. keys ()}
for name , mask in boundary_masks . items ():
mask . reshape ( - 1 )[ np . array ( f [ "grid/boundaries" ][ name ])] = 1
boundary_values = {
ft : {
name : np . atleast_1d ( desc [ "value" ])
for name , desc in f [ "boundary-conditions" ][ ft ]. items ()
if desc . attrs [ "type" ] == "fixed-value"
}
for ft in features
}
return t , data_3d , inside_mask , boundary_masks , boundary_valuesPuedes usarlo como
from matplotlib . pyplot import matshow
path = "data/shapes/data/2x2-large/data.h5"
t , data_3d , inside_mask , boundary_masks , boundary_values = load_data ( path , [ 50 , 300 ])
matshow ( np . linalg . norm ( data_3d [ - 1 , :, :, 20 , : 3 ], axis = - 1 ). T )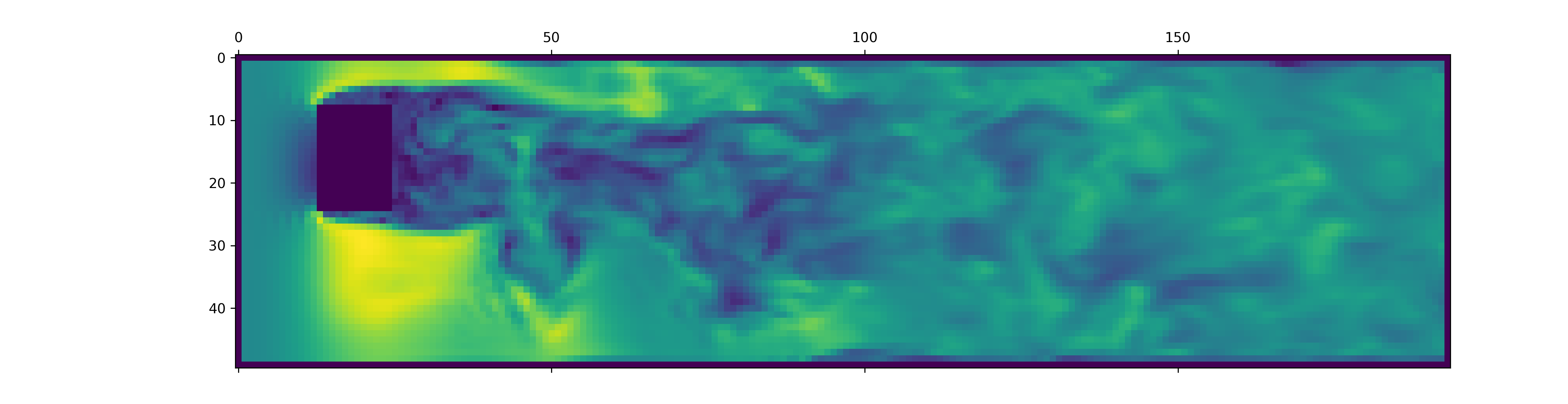
Los archivos data.h5 contienen más información que este fragmento. Para explorar qué más está disponible, hurgue en nuestro cargador de datos.
Para generar datos para nuevas simulaciones OpenFOAM, primero asegúrese de haber instalado las dependencias adicionales y que just disponibles:
pip install -e " .[data] " Si no desea usarlo just , también puede leer el justfile y ejecutar los comandos usted mismo.
Comience creando un contenedor Docker con OpenFoam instalado:
just of-dockerAhora genere un montón de casos nuevos. Por ejemplo, lo siguiente establece todos los casos de OpenFoam (simulaciones) de nuestro conjunto de datos:
./scripts/generate-shapes.py data/shapesPor supuesto, puede adaptar el script para crear otras formas o conjuntos de datos completamente nuevos.
Ahora puede resolver el caso (ejecutar la simulación) con OpenFoam localmente
just of-solve path/to/caseO envíe un montón de ellos a su propio clúster de shurm:
./scripts/solve-slurm.py data/shapes/data/ * /casePosteriormente, aplique el posprocesamiento, por ejemplo, la conversión HDF5, a cada simulación, por ejemplo
just postprocess data/shapes/data/2x2Finalmente, calcule las estadísticas del conjunto de capacitación para la normalización de características:
./scripts/dataset-stats.py data/shapes Para comenzar un entrenamiento, llame train.py con su configuración, por ejemplo
./train.py data.batch_size=128 El script de capacitación utiliza HYDRA para la configuración, así que consulte los archivos en el directorio config , para conocer todas las configuraciones disponibles.
Para volver a ejecutar los experimentos del documento, ejecute
./train.py -cn shapes_experiment -m que comienza a entrenar con la configuración en config/shapes_experiment.yaml . Si no tiene un clúster SLURM disponible, elimine la configuración relacionada con launcher .
Si se basa en este trabajo, cite nuestro artículo de la siguiente manera.
@inproceedings{lienen2024zero,
title = {From {{Zero}} to {{Turbulence}}: {{Generative Modeling}} for {{3D Flow Simulation}}},
author = {Lienen, Marten and L{"u}dke, David and {Hansen-Palmus}, Jan and G{"u}nnemann, Stephan},
booktitle = {International {{Conference}} on {{Learning Representations}}},
year = {2024},
}