マーテン・リーネン、デビッド・リュドケ、ヤン・ハンセン・パルムス、ステファン・ギュンネマン

このリポジトリには、私たちの論文で結果を生成するために使用されるコードが含まれています:openreview、arxiv。
モデル、データの読み込み、およびトレーニングコードに加えて、このリポジトリには、OpenFoamを構成および実行し、その出力をポストプロセスするコードも含まれています。これらのツールは、この分野の他の研究者にとって非常に有用な出発点になる可能性があります。特に、あります
# Clone the repository
git clone https://github.com/martenlienen/generative-turbulence.git
# Change into the repository
cd generative-turbulence
# Install package editably with dependencies
pip install -e .
# If you need a specific pytorch version, e.g. CPU-only or an older CUDA version, check
#
# https://pytorch.org/get-started/locally/
#
# and run, for example,
#
# pip install torch --extra-index-url https://download.pytorch.org/whl/cu117
#
# before installing this package. Shapes Datasetは、Tum University Libraryでホストされています。ダウンロードするには、ページの指示に従って、次の手順を実行します。まず、ファイルをダウンロードします。
# Download all archives to data/shapes/download
scripts/download-dataset.sh処理されたデータは約2TBであるため、これには長い時間がかかる場合があります。スクリプトはrsyncを使用するため、部分的なダウンロードを再開できます。生のオープンフォームケースデータもダウンロードしたい場合は、代わりに実行します
scripts/download-dataset.sh --with-rawInvididual Archivesをダウンロードした後、ファイルを抽出する必要があります。次のスクリプトがあなたのためにそうしています
scripts/extract-dataset.shその後、以下に説明するようにモデルのトレーニングを開始できます。
次のスニペットは、データセット内のdata.h5ファイルからデータをロードして、探索して実験します。
import numpy as np
import h5py as h5
def load_data ( path , idx , features = [ "u" , "p" ]):
"""Load data from a data.h5 file into an easily digestible matrix format.
Arguments
---------
path
Path to a data.h5 file in the `shapes` dataset
idx
Index or indices of sample to load. Can be a number, list, boolean mask or a slice.
features
Features to load. By default loads only velocity and pressure but you can also
access the LES specific k and nut variables.
Returns
-------
t: np.ndarray of shape T
Time steps of the loaded data frames
data_3d: np.ndarray of shape T x W x H x D x F
3D data with all features concatenated in the order that they are requested, i.e.
in the default case the first 3 features will be the velocity vector and the fourth
will be the pressure
inside_mask: np.ndarray of shape W x H x D
Boolean mask that marks the inside cells of the domain, i.e. cells that are not part
of walls, inlets or outlets
boundary_masks: dict of str to nd.ndarray of shape W x H x D
Masks that mark cells belonging to each type of boundary
boundary_values: dict[str, dict[str, np.ndarray]]
Prescribed values for variables and boundaries with Dirichlet boundary conditions
"""
with h5 . File ( path , mode = "r" ) as f :
t = np . array ( f [ "data/times" ])
cell_data = np . concatenate ([ np . atleast_3d ( f [ "data" ][ name ][ idx ]) for name in features ], axis = - 1 )
padded_cell_counts = np . array ( f [ "grid/cell_counts" ])
cell_idx = np . array ( f [ "grid/cell_idx" ])
n_steps , n_features = cell_data . shape [ 0 ], cell_data . shape [ - 1 ]
data_3d = np . zeros (( n_steps , * padded_cell_counts , n_features ))
data_3d . reshape (( n_steps , - 1 , n_features ))[:, cell_idx ] = cell_data
inside_mask = np . zeros ( padded_cell_counts , dtype = bool )
inside_mask . reshape ( - 1 )[ cell_idx ] = 1
boundary_masks = { name : np . zeros ( padded_cell_counts , dtype = bool ) for name in f [ "grid/boundaries" ]. keys ()}
for name , mask in boundary_masks . items ():
mask . reshape ( - 1 )[ np . array ( f [ "grid/boundaries" ][ name ])] = 1
boundary_values = {
ft : {
name : np . atleast_1d ( desc [ "value" ])
for name , desc in f [ "boundary-conditions" ][ ft ]. items ()
if desc . attrs [ "type" ] == "fixed-value"
}
for ft in features
}
return t , data_3d , inside_mask , boundary_masks , boundary_valuesそのように使用できます
from matplotlib . pyplot import matshow
path = "data/shapes/data/2x2-large/data.h5"
t , data_3d , inside_mask , boundary_masks , boundary_values = load_data ( path , [ 50 , 300 ])
matshow ( np . linalg . norm ( data_3d [ - 1 , :, :, 20 , : 3 ], axis = - 1 ). T )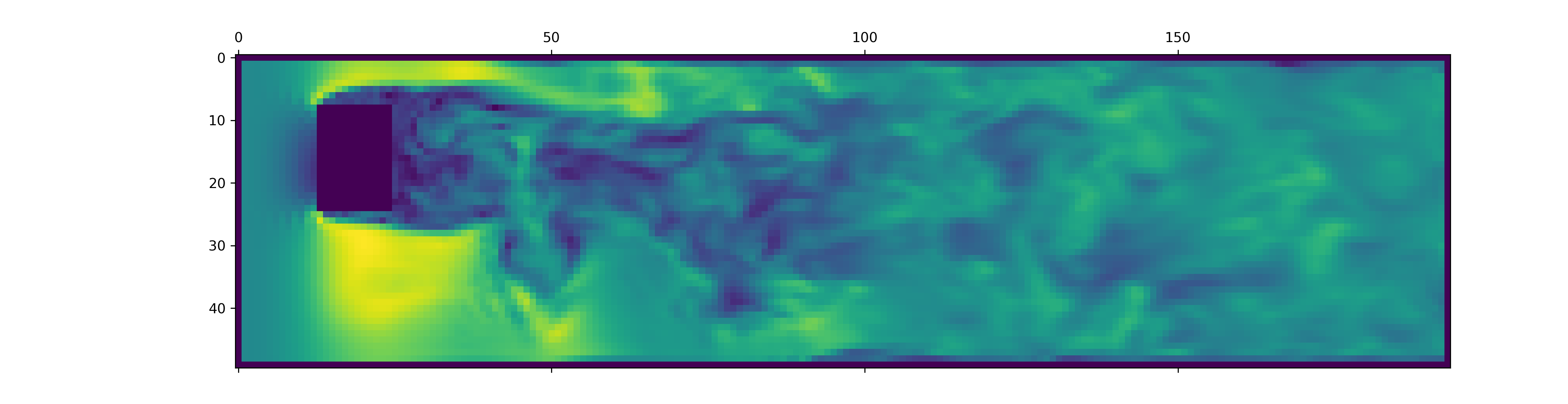
data.h5ファイルには、このスニペットの負荷よりも多くの情報が含まれています。他に何が利用できるかを調べるには、データローダーで突き刺してください。
新しいOpenFOAMシミュレーションのデータを生成するには、最初に追加の依存関係をインストールし、利用可能なjustあることを確認してください。
pip install -e " .[data] " just使用したくない場合は、 justfileを読んでコマンドを実行することもできます。
OpenFoamがインストールされたDockerコンテナを作成することから始めます。
just of-dockerここで、たくさんの新しいケースを生成します。たとえば、以下は、データセットからすべてのOpenFoamケース(シミュレーション)をセットアップします。
./scripts/generate-shapes.py data/shapesもちろん、スクリプトを適応させて、他の形状または完全に新しいデータセットを作成できます。
これで、オープンフォームをローカルに使用してケース(シミュレーションを実行)を解決できます
just of-solve path/to/caseまたは、それらの束を自分のスラークラスターに提出してください。
./scripts/solve-slurm.py data/shapes/data/ * /caseその後、たとえば、各シミュレーションにポストプロセスを各シミュレーションに適用します。
just postprocess data/shapes/data/2x2最後に、機能の正規化のためのトレーニングセット統計を計算します。
./scripts/dataset-stats.py data/shapesトレーニングを開始するには、たとえば、設定でtrain.pyに電話してください
./train.py data.batch_size=128トレーニングスクリプトは構成にHydraを使用するため、 configディレクトリのファイルをチェックして、利用可能なすべての設定について学びます。
論文からの実験を再実行するには、実行します
./train.py -cn shapes_experiment -m config/shapes_experiment.yamlの設定でトレーニングを開始します。 SluRMクラスターを使用できない場合は、 launcherに関連する設定を削除します。
この作業の上に構築されている場合は、次のように論文を引用してください。
@inproceedings{lienen2024zero,
title = {From {{Zero}} to {{Turbulence}}: {{Generative Modeling}} for {{3D Flow Simulation}}},
author = {Lienen, Marten and L{"u}dke, David and {Hansen-Palmus}, Jan and G{"u}nnemann, Stephan},
booktitle = {International {{Conference}} on {{Learning Representations}}},
year = {2024},
}