Этот репозиторий содержит код следующей статьи:
@misc{ziletti2024retrieval,
title={Retrieval augmented text-to-SQL generation for epidemiological question answering using electronic health records},
author={Angelo Ziletti and Leonardo D'Ambrosi},
year={2024},
eprint={2403.09226},
archivePrefix={arXiv},
primaryClass={cs.CL}
}
Пожалуйста, найдите предварительный результат здесь: Ziletti и D'Ambrosi, https://arxiv.org/abs/2403.09226
Эта статья принята на Clinical NLP Workshop NAACL 2024 (https://clinical-nlp.github.io/2024/)
Пожалуйста, цитируйте эту работу, если вы используете этот код в своей работе или в исследованиях.
Вот сводный рабочий процесс процедуры: 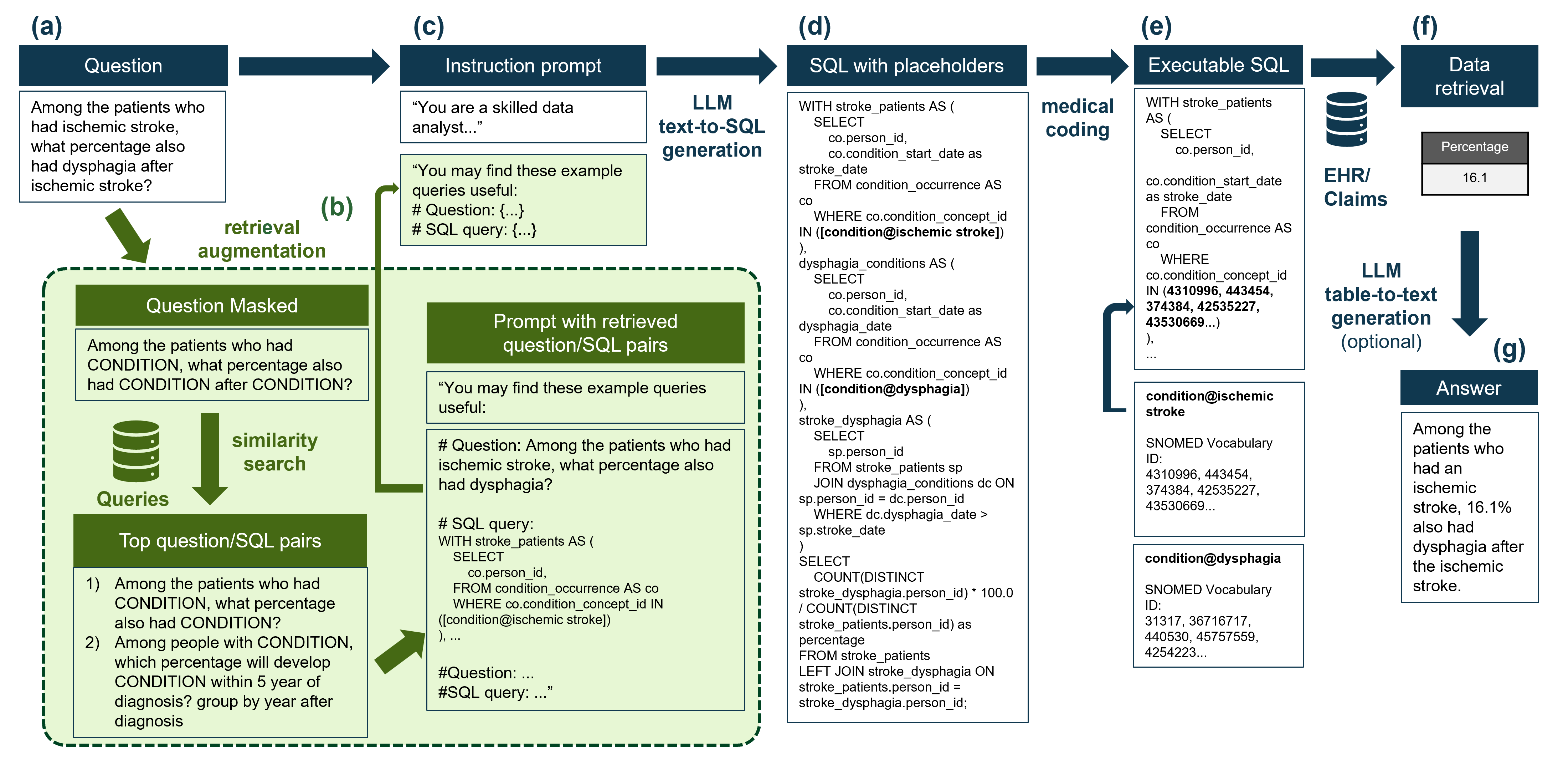 А вот основные результаты:
А вот основные результаты: 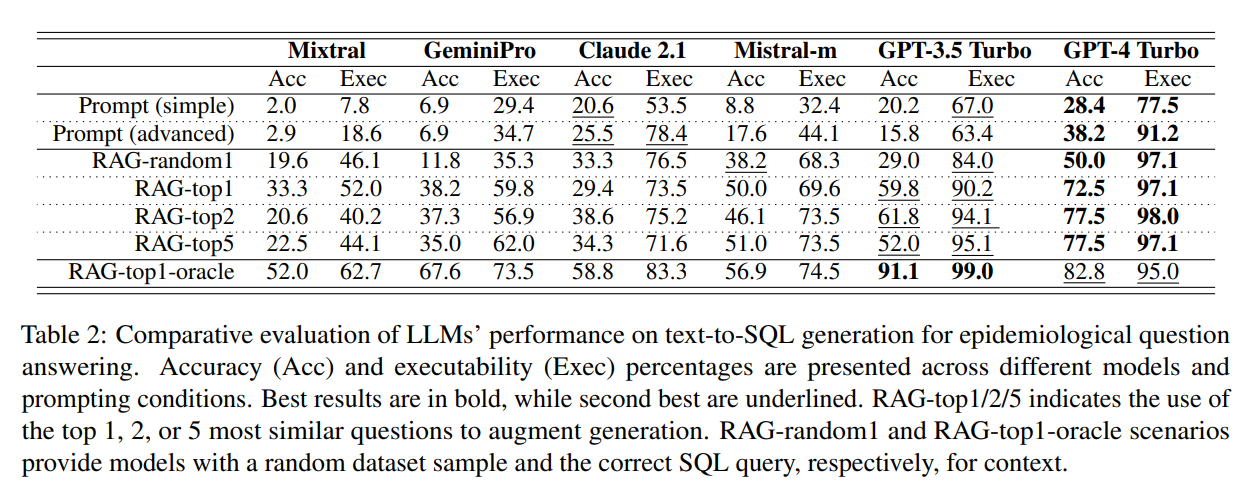
Пожалуйста, обратитесь к рукописи для получения более подробной информации: https://arxiv.org/abs/2403.09226
Парки с меченым вопросом SQL находятся в файле text2sql_epi_dataset_omop.xlsx в папке dataset .
Мы использовали Python 3.11 в наших экспериментах.
Установите последнюю версию из Master Branch On Github by:
git clone <GITHUB-URL>
cd text-to-sql-epi-ehr-naacl2024
pip install -r requirements.txt
Во -первых, нам нужно создать файл мариноза (библиотека запросов) из файла Excel, предоставленного в папке dataset
cd scripts
python run_querylib_calc.py
Этот маринованный файл будет использоваться для выполнения получения добычи из поиска (RAG) на стадии генерации запросов.
Чтобы выполнить прогноз, запустите Script prediction_pipeline.py с вашим вопросом в двойных кавычках. Например,
cd scripts
python prediction_pipeline.py --question "How many women with atopic dermatitis?"
Это должно вернуться:
Question: How many women with atopic dermatitis?
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Чтобы выполнить запрос, вы будете здесь ссылкой на набор данных в Google Cloud: https://console.cloud.google.com/marketplace/product/hhs/synpuf. Затем вы можете открыть ссылку (потребуется учетная запись Google) и запустить запрос в своей учетной записи Google.
Чтобы сделать исполняемый файл запроса в Google BigQuery, вам нужно будет изменить сгенерированные имена таблицы, чтобы соответствовать именам таблиц таблицы Google. Это просто переименование таблиц.
Например,
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM person AS p
JOIN condition_occurrence AS co ON p.person_id = co.person_id
JOIN concept AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
должен быть изменен на
SELECT
COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM `bigquery-public-data.cms_synthetic_patient_data_omop.person` AS p
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.condition_occurrence` AS co ON p.person_id = co.person_id
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.concept` AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
запустить на экземпляр BigQuery выше.
Если у вас есть собственное хранилище данных снежинки, вам, возможно, не нужно будет выполнять эти изменения.
В репозитории мы предоставляем версию макета для медицинского кодирования, чтобы показать, как медицинское кодирование может быть интегрировано в процесс, как показано на рисунке выше. Кодирование выполняется в рамках онтологии Snomed, поскольку это основная онтология для данных, хранящихся в модели общих данных OMOP (OMOP-CDM). Таблица обычно называется concept_table в OMOP-CDM.
Мы подготовили небольшую таблицу макетов в папке dataset с названием medcodes_mockup.xlsx . Здесь мы предоставляем синтетические данные после структуры данных OMOP-CDM. Вы можете скачать всю онтологию из Национальной медицинской библиотеки здесь. (Обратите внимание, что вам нужно принять их условия)
Более подробную информацию о Snomed-CT можно найти, например, здесь
Во -первых, нам нужно создать файл мариноза (библиотека MedCodeonto) из предоставленного файла Excel
cd scripts
python run_medcoding_calc.py
Этот маринованный файл будет использоваться для поиска медицинских кодов в файле Pickle. Запуск файла также даст пример того, как выполняется кодирование.
Обратите внимание, что это только реализация макета. Для готовой к производству версии мы предлагаем использовать векторную базу данных (например, Qdrant), индексируя всю таблицу концепций, например, из базы данных снежинки.
После выполнения шага Medical coding compilation вы готовы запустить полный трубопровод: генерация запросов SQL + медицинское кодирование.
Это пример:
cd scripts
python prediction_pipeline.py --med_coding True --question "How many females with atopic dermatitis"
Это вернется
Use medical coding: True
Use Snowflake database: False
No sentence-transformers model found with name BAAI/bge-large-en-v1.5. Creating a new one with MEAN pooling.
Question: How many females with atopic dermatitis
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Loading embedding from C:UsersGKENYOneDrive - BayerPersonal Dataascenttext-to-sql-epi-ehr-naacl2024data_outmedcodes_onto.pkl
Retrieved codes: {'atopic dermatitis': [{'Score': 0.8221014142036438, 'CONCEPT_NAME': 'Dermatitis in children', 'CONCEPT_ID': 4296192, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402196005}, {'Score': 0.8056577444076538, 'CONCEPT_NAME': 'Widespread dermatitis', 'CONCEPT_ID': 4298597, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402186001}, {'Score': 0.7999188899993896, 'CONCEPT_NAME': 'Early childhood dermatitis', 'CONCEPT_ID': 4298599, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402195009}, {'Score': 0.7992758750915527, 'CONCEPT_NAME': 'Facial dermatitis', 'CONCEPT_ID': 4298598, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402192007}]}
Please note that the medical coding is based on a mockup ontology. Results will not be reliable
SQL filled:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN (4296192,4298597,4298599,4298598)
AND p.gender_concept_id = 8532;
Обратите внимание, что этап медицинского кодирования здесь только образцом. Чтобы получить надежные результаты, вам необходимо получить доступ к полной онтологии Snomed (см. Раздел Medical coding compilation ).