이 저장소에는 다음 논문의 코드가 포함되어 있습니다.
@misc{ziletti2024retrieval,
title={Retrieval augmented text-to-SQL generation for epidemiological question answering using electronic health records},
author={Angelo Ziletti and Leonardo D'Ambrosi},
year={2024},
eprint={2403.09226},
archivePrefix={arXiv},
primaryClass={cs.CL}
}
Ziletti와 D 'Ambrosi, https://arxiv.org/abs/2403.09226을 참조하십시오
이 논문은 NAACL 2024 임상 NLP 워크숍 (https://clinical-nlp.github.io/2024/)에서 허용됩니다.
작업이나 연구 에서이 코드를 사용하는 경우이 작업을 인용하십시오.
절차의 요약 워크 플로우는 다음과 같습니다. 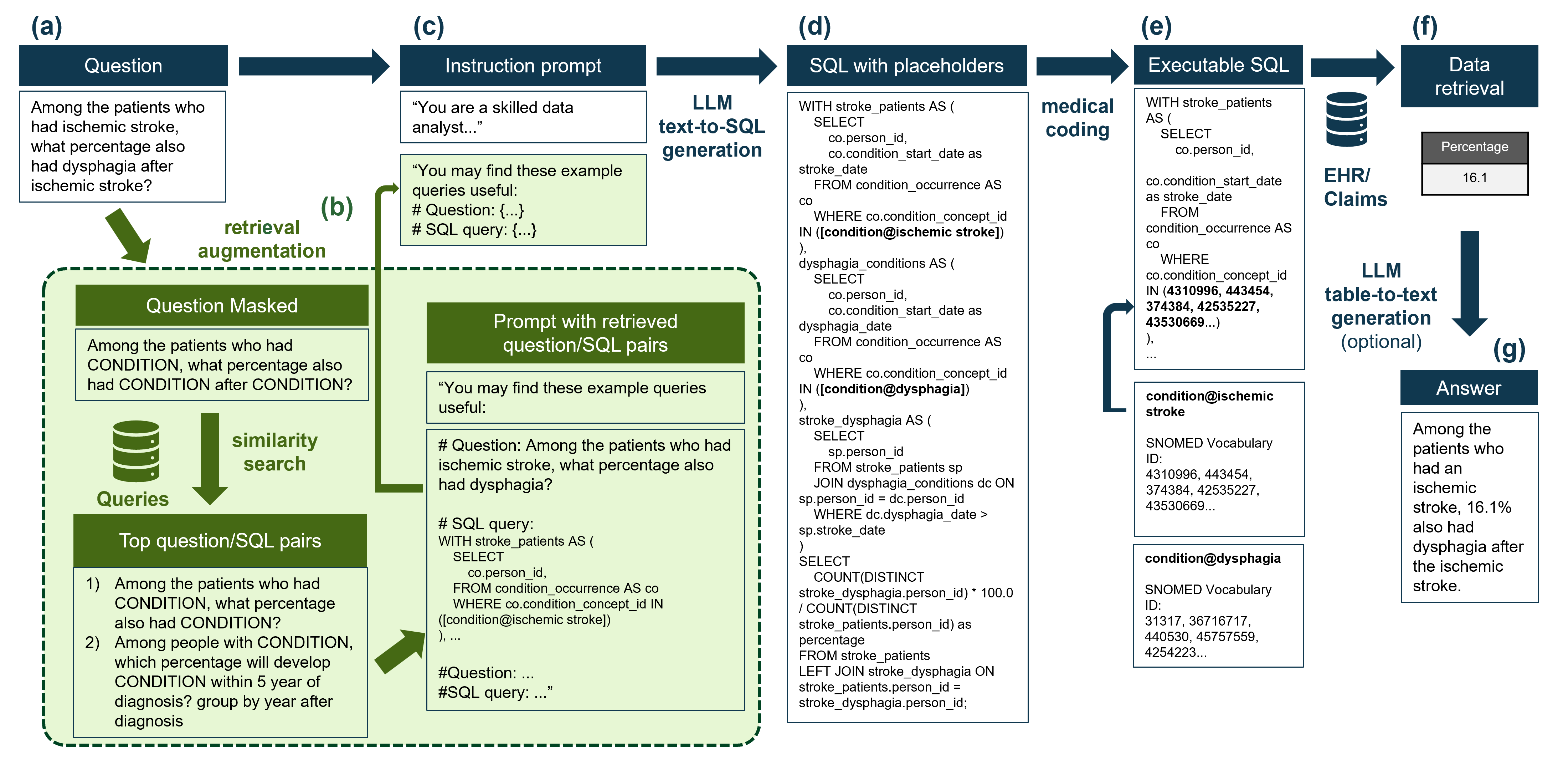 그리고 다음은 주요 결과입니다.
그리고 다음은 주요 결과입니다. 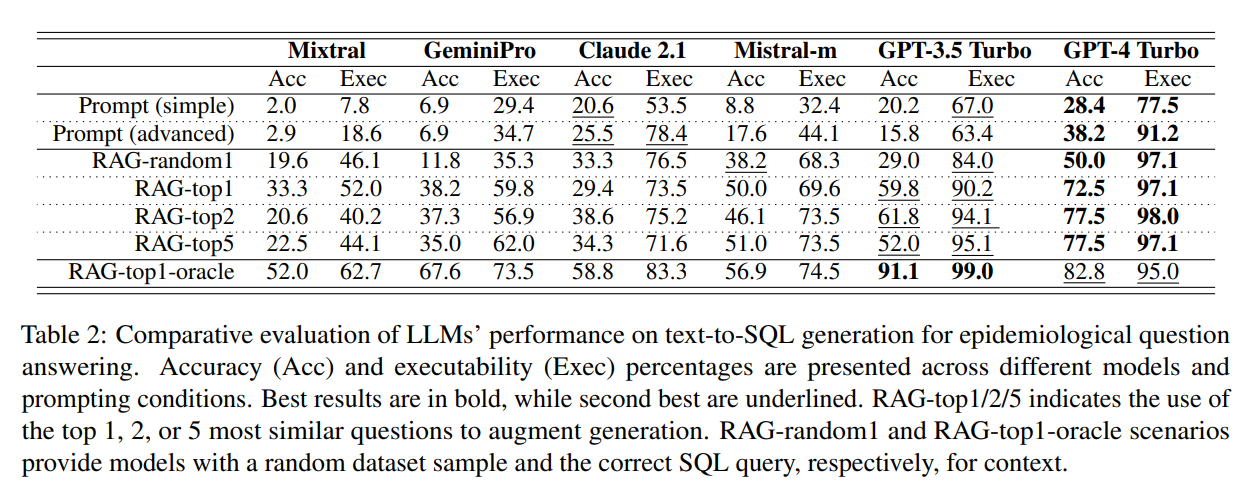
자세한 내용은 원고를 참조하십시오 : https://arxiv.org/abs/2403.09226
라벨이 붙은 Question-SQL 쌍은 dataset 폴더의 파일 text2sql_epi_dataset_omop.xlsx 파일에 있습니다.
우리는 실험에서 Python 3.11을 사용했습니다.
github의 마스터 브랜치에서 최신 버전을 설치하십시오.
git clone <GITHUB-URL>
cd text-to-sql-epi-ehr-naacl2024
pip install -r requirements.txt
먼저 dataset 폴더에 제공된 Excel 파일에서 피클 파일 (쿼리 라이브러리)을 작성해야합니다.
cd scripts
python run_querylib_calc.py
이 피클 파일은 쿼리 생성 단계에서 검색 증강 생성 (RAG)을 수행하는 데 사용됩니다.
예측을 수행하려면 질문으로 스크립트 prediction_pipeline.py 이중 인용문으로 실행하십시오. 예를 들어,
cd scripts
python prediction_pipeline.py --question "How many women with atopic dermatitis?"
이것은 돌아와야합니다 :
Question: How many women with atopic dermatitis?
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
쿼리를 실행하려면 여기에 Google Cloud의 데이터 세트 링크가 있습니다 : https://console.cloud.google.com/marketplace/product/hhs/synpuf 링크를 열고 Google 계정에서 쿼리를 실행할 수 있습니다.
Google BigQuery에서 쿼리 실행 파일을 만들려면 Google 쿼리 테이블 이름을 준수하려면 생성 된 테이블 이름을 수정해야합니다. 이것은 단순히 테이블의 이름을 바꾸는 것입니다.
예를 들어,
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM person AS p
JOIN condition_occurrence AS co ON p.person_id = co.person_id
JOIN concept AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
변경해야합니다
SELECT
COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM `bigquery-public-data.cms_synthetic_patient_data_omop.person` AS p
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.condition_occurrence` AS co ON p.person_id = co.person_id
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.concept` AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
위의 BigQuery 인스턴스에서 실행됩니다.
자신의 눈송이 데이터웨어 하우스가있는 경우 이러한 변경 사항을 수행 할 필요가 없습니다.
저장소에서는 위의 그림과 같이 의료 코딩이 프로세스에 어떻게 통합 될 수 있는지 보여주는 의료 코딩을위한 모형 버전을 제공합니다. 코딩은 Snomed Ontology 내에서 수행됩니다. OKOP Common Data Model (OMOP-CDM)에 저장된 데이터의 기본 온톨로지이기 때문입니다. 테이블은 일반적으로 Omop-CDM에서 Concept_table이라고합니다.
dataset 폴더에서 medcodes_mockup.xlsx 라는 작은 모형 테이블을 준비했습니다. 여기서 우리는 OMOP-CDM의 데이터 구조에 따라 합성 데이터를 제공합니다. 국립 의학 도서관에서 온톨로지 전체를 다운로드 할 수 있습니다. (이용 약관을 수락해야합니다)
Snomed-CT에 대한 자세한 내용은 예를 들어 여기를 참조하십시오.
먼저 제공된 Excel 파일에서 피클 파일 (Medcodeonto 라이브러리)을 작성해야합니다.
cd scripts
python run_medcoding_calc.py
이 피클 파일은 피클 파일 내에서 의료 코드를 검색하는 데 사용됩니다. 파일을 실행하면 코딩이 수행되는 방법의 예도 제공됩니다.
이것은 모형 구현 일뿐입니다. 프로덕션 준비 버전의 경우 벡터 데이터베이스 (예 : QDRANT)를 사용하여 예를 들어 눈송이 데이터베이스에서 전체 개념 테이블을 색인화하는 것이 좋습니다.
Medical coding compilation 단계를 실행 한 후에는 전체 파이프 라인을 실행할 준비가되어 있습니다 : SQL 쿼리 생성 + 의료 코딩.
이것은 예입니다.
cd scripts
python prediction_pipeline.py --med_coding True --question "How many females with atopic dermatitis"
이것은 돌아올 것입니다
Use medical coding: True
Use Snowflake database: False
No sentence-transformers model found with name BAAI/bge-large-en-v1.5. Creating a new one with MEAN pooling.
Question: How many females with atopic dermatitis
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Loading embedding from C:UsersGKENYOneDrive - BayerPersonal Dataascenttext-to-sql-epi-ehr-naacl2024data_outmedcodes_onto.pkl
Retrieved codes: {'atopic dermatitis': [{'Score': 0.8221014142036438, 'CONCEPT_NAME': 'Dermatitis in children', 'CONCEPT_ID': 4296192, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402196005}, {'Score': 0.8056577444076538, 'CONCEPT_NAME': 'Widespread dermatitis', 'CONCEPT_ID': 4298597, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402186001}, {'Score': 0.7999188899993896, 'CONCEPT_NAME': 'Early childhood dermatitis', 'CONCEPT_ID': 4298599, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402195009}, {'Score': 0.7992758750915527, 'CONCEPT_NAME': 'Facial dermatitis', 'CONCEPT_ID': 4298598, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402192007}]}
Please note that the medical coding is based on a mockup ontology. Results will not be reliable
SQL filled:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN (4296192,4298597,4298599,4298598)
AND p.gender_concept_id = 8532;
의료 코딩 단계는 여기에서만 예시 적입니다. 신뢰할 수있는 결과를 얻으려면 전체 노골적인 온톨로지에 액세스해야합니다 ( Medical coding compilation 섹션 참조).