Este repositorio contiene el código del siguiente documento:
@misc{ziletti2024retrieval,
title={Retrieval augmented text-to-SQL generation for epidemiological question answering using electronic health records},
author={Angelo Ziletti and Leonardo D'Ambrosi},
year={2024},
eprint={2403.09226},
archivePrefix={arXiv},
primaryClass={cs.CL}
}
Encuentre la preimpresión aquí: Ziletti y D'Ambrosi, https://arxiv.org/abs/2403.09226
Este documento se acepta en el Taller NLP Clinical NAACL 2024 (https://clinical-nlp.github.io/2024/)
Cite este trabajo si usa este código en su trabajo o investigación.
Aquí hay un flujo de trabajo resumido del procedimiento: 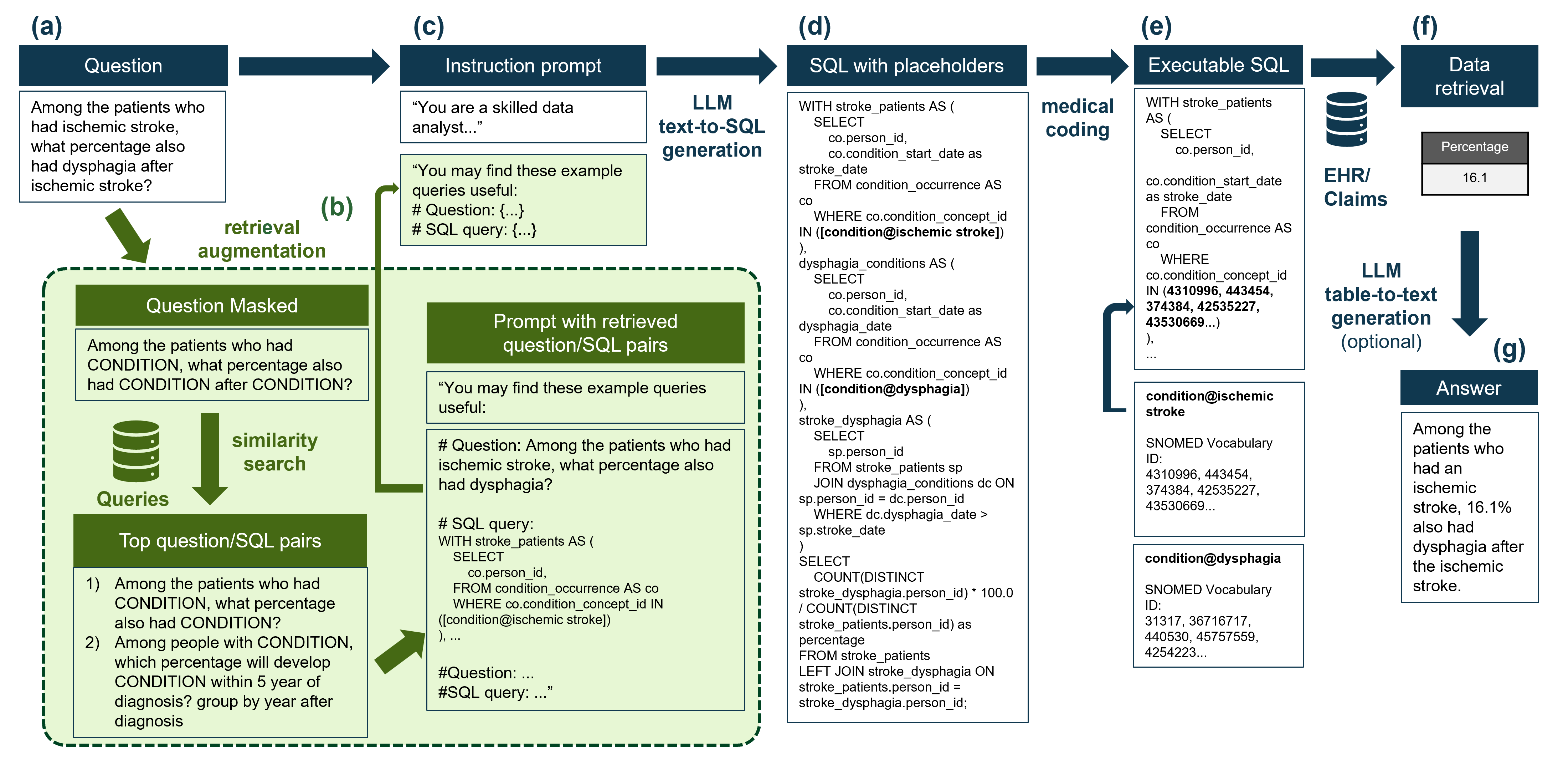 Y aquí están los principales resultados:
Y aquí están los principales resultados: 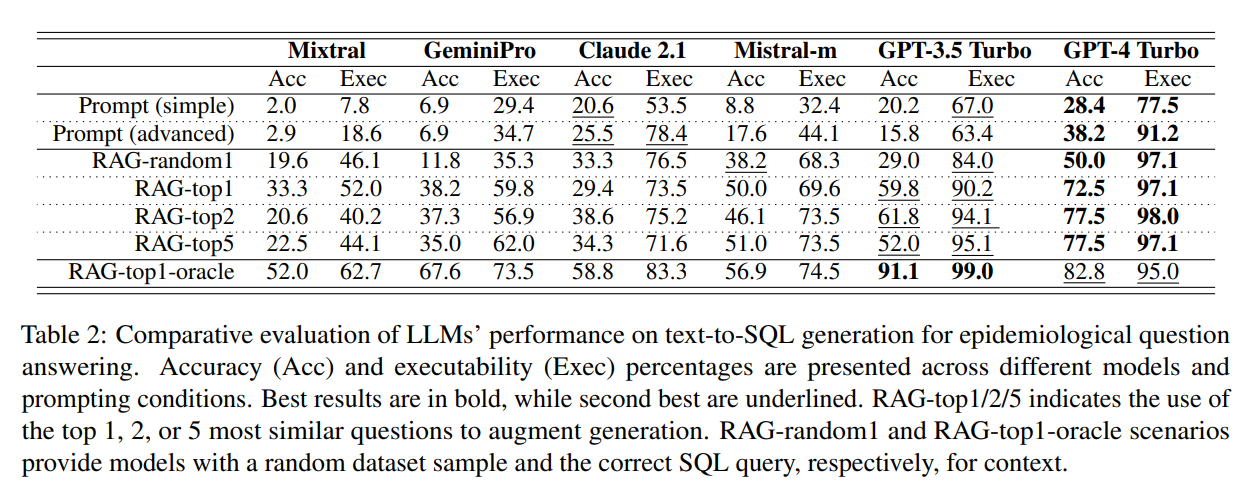
Consulte el manuscrito para obtener más detalles: https://arxiv.org/abs/2403.09226
Los pares de cuestionamiento de SQL etiquetados están en el archivo text2sql_epi_dataset_omop.xlsx en la carpeta dataset .
Usamos Python 3.11 en nuestros experimentos.
Instale la última versión de la rama maestra en GitHub por:
git clone <GITHUB-URL>
cd text-to-sql-epi-ehr-naacl2024
pip install -r requirements.txt
Primero, necesitamos crear un archivo de pickle (la biblioteca de consultas) desde el archivo de Excel proporcionado en la carpeta dataset
cd scripts
python run_querylib_calc.py
Este archivo de encurtido se utilizará para realizar la generación aumentada de recuperación (RAG) en la etapa de generación de consultas.
Para realizar una predicción, ejecute el script prediction_pipeline.py con su pregunta en citas dobles. Por ejemplo,
cd scripts
python prediction_pipeline.py --question "How many women with atopic dermatitis?"
Esto debería volver:
Question: How many women with atopic dermatitis?
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Para ejecutar la consulta, aquí será un enlace al conjunto de datos en Google Cloud: https://console.cloud.google.com/marketplace/product/hhs/synpuf puede abrir el enlace (necesitará una cuenta de Google), y ejecutar la consulta en su cuenta de Google.
Para hacer el ejecutable de consulta en Google BigQuery, deberá modificar los nombres generados de la tabla para cumplir con los nombres de la tabla de consulta de Google. Esto es simplemente un cambio de nombre de las tablas.
Por ejemplo,
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM person AS p
JOIN condition_occurrence AS co ON p.person_id = co.person_id
JOIN concept AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
debe cambiarse a
SELECT
COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM `bigquery-public-data.cms_synthetic_patient_data_omop.person` AS p
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.condition_occurrence` AS co ON p.person_id = co.person_id
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.concept` AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
para ejecutar en la instancia de BigQuery arriba.
Si tiene su propio almacén de datos de copo de nieve, es posible que no necesite realizar esos cambios.
En el repositorio, proporcionamos una versión de maqueta para la codificación médica para mostrar cómo la codificación médica se puede integrar en el proceso, como se muestra en la figura anterior. La codificación se realiza dentro de la ontología Snomed, ya que esa es la ontología subyacente para los datos almacenados en el modelo OMOP Common Data (OMOP-CDM). La tabla generalmente se llama concept_table en el OMOP-CDM.
Preparamos una pequeña tabla de maquetas en la carpeta dataset llamada medcodes_mockup.xlsx . Aquí proporcionamos datos sintéticos siguiendo la estructura de datos del OMOP-CDM. Puede descargar toda la ontología de la Biblioteca Nacional de Medicina aquí. (Tenga en cuenta que debe aceptar sus términos y condiciones)
Puede encontrar más información sobre Snomed-CT, por ejemplo, aquí
Primero, necesitamos crear un archivo de encurtido (la biblioteca MedCodeonto) desde el archivo de Excel proporcionado
cd scripts
python run_medcoding_calc.py
Este archivo de encurtido se utilizará para buscar códigos médicos dentro del archivo de encurtido. Ejecutar el archivo también proporcionará un ejemplo de cómo se realiza la codificación.
Tenga en cuenta que esto es solo una implementación de maquetas. Para una versión lista para la producción, sugerimos usar una base de datos vectorial (por ejemplo Qdrant), indexando toda la tabla conceptual, por ejemplo, de una base de datos de copo de nieve.
Después de haber ejecutado el paso Medical coding compilation , está listo para ejecutar la tubería completa: SQL Generación de consultas + codificación médica.
Este es un ejemplo:
cd scripts
python prediction_pipeline.py --med_coding True --question "How many females with atopic dermatitis"
Esto volverá
Use medical coding: True
Use Snowflake database: False
No sentence-transformers model found with name BAAI/bge-large-en-v1.5. Creating a new one with MEAN pooling.
Question: How many females with atopic dermatitis
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Loading embedding from C:UsersGKENYOneDrive - BayerPersonal Dataascenttext-to-sql-epi-ehr-naacl2024data_outmedcodes_onto.pkl
Retrieved codes: {'atopic dermatitis': [{'Score': 0.8221014142036438, 'CONCEPT_NAME': 'Dermatitis in children', 'CONCEPT_ID': 4296192, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402196005}, {'Score': 0.8056577444076538, 'CONCEPT_NAME': 'Widespread dermatitis', 'CONCEPT_ID': 4298597, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402186001}, {'Score': 0.7999188899993896, 'CONCEPT_NAME': 'Early childhood dermatitis', 'CONCEPT_ID': 4298599, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402195009}, {'Score': 0.7992758750915527, 'CONCEPT_NAME': 'Facial dermatitis', 'CONCEPT_ID': 4298598, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402192007}]}
Please note that the medical coding is based on a mockup ontology. Results will not be reliable
SQL filled:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN (4296192,4298597,4298599,4298598)
AND p.gender_concept_id = 8532;
Tenga en cuenta que el paso de codificación médica solo es ejemplificador aquí. Para obtener resultados confiables, deberá tener acceso a la ontología snomed completa (ver sección Medical coding compilation ).