このリポジトリには、次の論文のコードが含まれています。
@misc{ziletti2024retrieval,
title={Retrieval augmented text-to-SQL generation for epidemiological question answering using electronic health records},
author={Angelo Ziletti and Leonardo D'Ambrosi},
year={2024},
eprint={2403.09226},
archivePrefix={arXiv},
primaryClass={cs.CL}
}
Ziletti and D'Ambrosi、https://arxiv.org/abs/2403.09226をご覧ください
このペーパーは、NAACL 2024 Clinical NLPワークショップ(https://clinical-nlp.github.io/2024/)で受け入れられています。
このコードを作業や調査で使用する場合は、この作業を引用してください。
手順の要約ワークフローは次のとおりです。 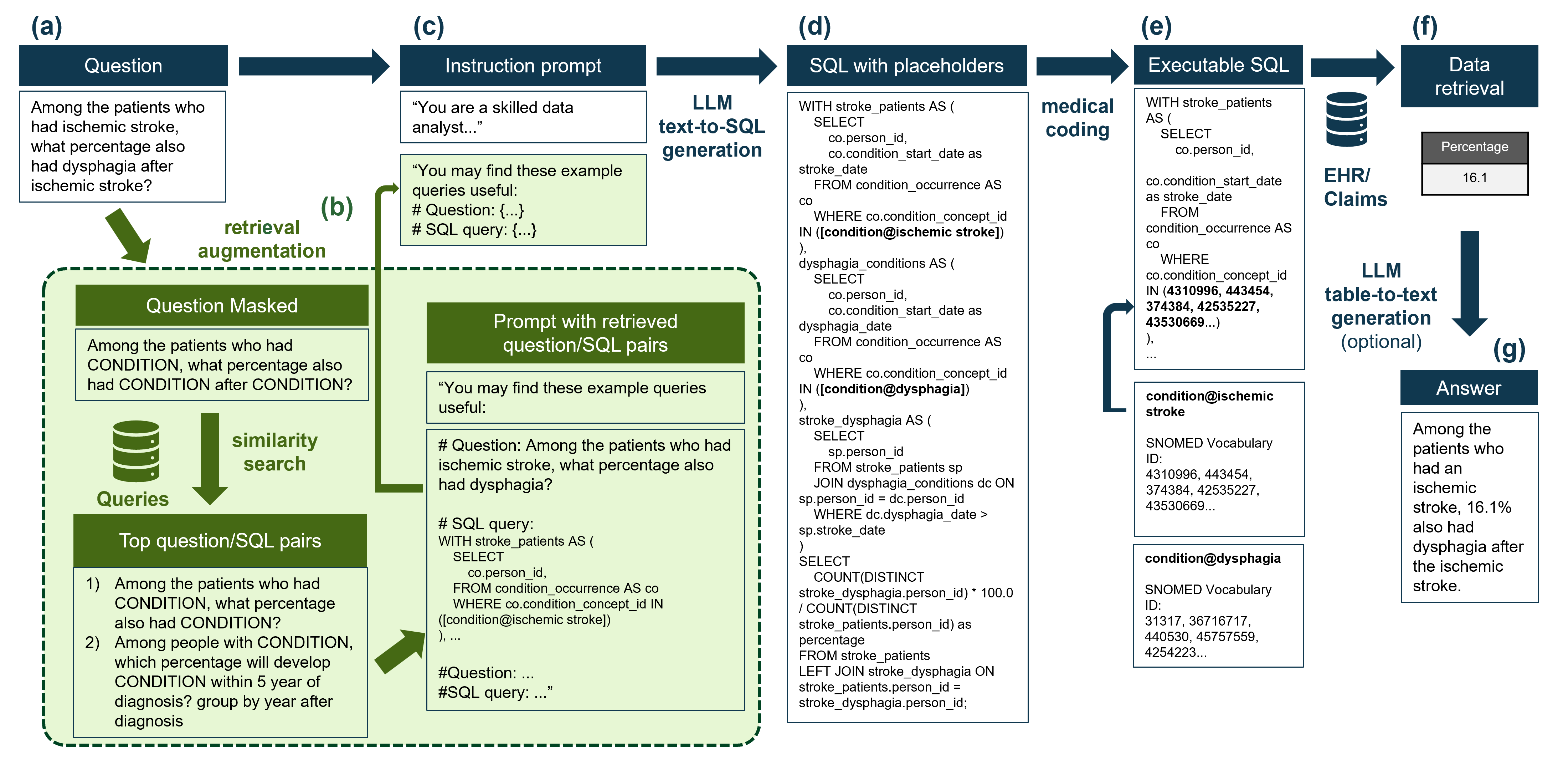 そして、これが主な結果です:
そして、これが主な結果です: 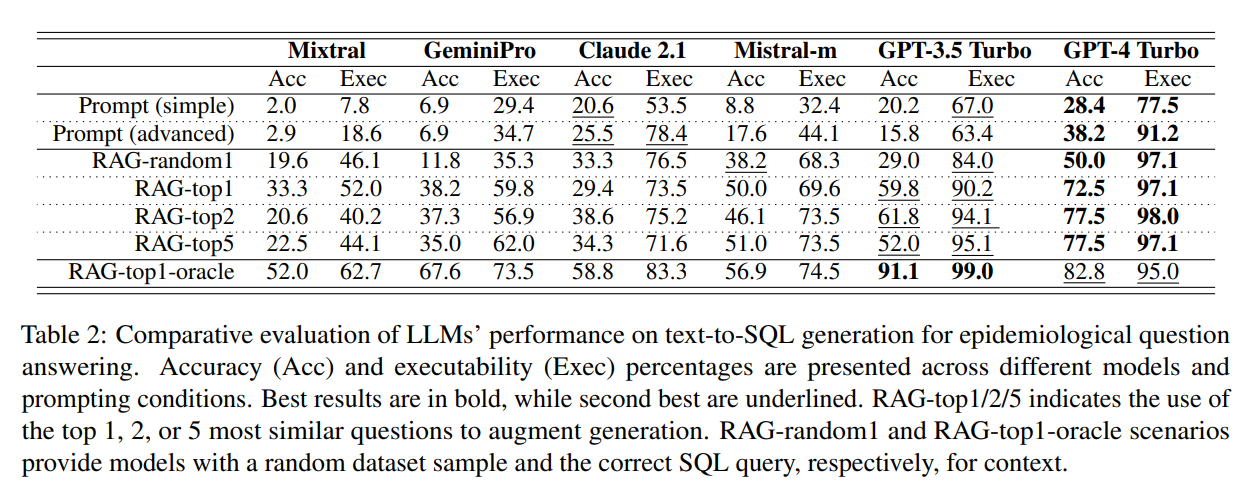
詳細については、原稿を参照してください:https://arxiv.org/abs/2403.09226
ラベル付きの質問SQLペアは、 datasetフォルダーのファイルtext2sql_epi_dataset_omop.xlsxにあります。
実験でPython 3.11を使用しました。
GitHubのMaster Branchから最新バージョンをインストールしてください。
git clone <GITHUB-URL>
cd text-to-sql-epi-ehr-naacl2024
pip install -r requirements.txt
まず、 datasetフォルダーに記載されているExcelファイルからピクルスファイル(クエリライブラリ)を作成する必要があります
cd scripts
python run_querylib_calc.py
このピクルスファイルは、クエリ生成段階で検索拡張生成(RAG)を実行するために使用されます。
予測を実行するには、質問を二重引用符で掲載したスクリプトprediction_pipeline.pyを実行します。例えば、
cd scripts
python prediction_pipeline.py --question "How many women with atopic dermatitis?"
これは戻ります:
Question: How many women with atopic dermatitis?
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
クエリを実行するには、Googleクラウドのデータセットへのリンクがあります:https://console.cloud.google.com/marketplace/product/hhs/synpuf次に、リンクを開くことができます(Googleアカウントが必要です)。
Google BigQueryでクエリ実行可能ファイルを作成するには、Googleクエリテーブル名に準拠するために、テーブルの生成された名前を変更する必要があります。これは、単にテーブルの名前の変更です。
例えば、
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM person AS p
JOIN condition_occurrence AS co ON p.person_id = co.person_id
JOIN concept AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
に変更する必要があります
SELECT
COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM `bigquery-public-data.cms_synthetic_patient_data_omop.person` AS p
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.condition_occurrence` AS co ON p.person_id = co.person_id
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.concept` AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
上記のBigQueryインスタンスで実行します。
独自のスノーフレークデータウェアハウスをお持ちの場合は、これらの変更を実行する必要がない場合があります。
リポジトリでは、上の図に示すように、医療コーディングをプロセスに統合する方法を示すために、医療コーディング用のモックアップバージョンを提供します。コーディングは、Snomed Ontology内で実行されます。これは、OMOP Common Data Model(OMOP-CDM)に保存されているデータの基礎となるオントロジーです。テーブルは通常、OMOP-CDMのConcept_Tableと呼ばれます。
medcodes_mockup.xlsxと呼ばれるdatasetセットフォルダーに小さなモックアップテーブルを準備しました。ここでは、OMOP-CDMのデータ構造に従って合成データを提供します。ここでは、医学国立図書館からオントロジー全体をダウンロードできます。 (あなたは彼らの利用規約を受け入れる必要があることに注意してください)
Snomed-CTの詳細については、たとえばこちらをご覧ください。
まず、提供されたExcelファイルからピクルスファイル(Medcodeontoライブラリ)を作成する必要があります
cd scripts
python run_medcoding_calc.py
このピクルスファイルは、ピクルファイル内の医療コードを検索するために使用されます。ファイルを実行すると、コーディングの実行方法も表示されます。
これはモックアップの実装にすぎないことに注意してください。制作対応バージョンの場合、ベクトルデータベース(QDrantなど)を使用して、Snowflakeデータベースからコンセプトテーブル全体をインデックス作成することをお勧めします。
Medical coding compilationステップを実行した後、完全なパイプライン:SQLクエリ生成 +医療コーディングを実行する準備ができています。
これは例です:
cd scripts
python prediction_pipeline.py --med_coding True --question "How many females with atopic dermatitis"
これは戻ります
Use medical coding: True
Use Snowflake database: False
No sentence-transformers model found with name BAAI/bge-large-en-v1.5. Creating a new one with MEAN pooling.
Question: How many females with atopic dermatitis
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Loading embedding from C:UsersGKENYOneDrive - BayerPersonal Dataascenttext-to-sql-epi-ehr-naacl2024data_outmedcodes_onto.pkl
Retrieved codes: {'atopic dermatitis': [{'Score': 0.8221014142036438, 'CONCEPT_NAME': 'Dermatitis in children', 'CONCEPT_ID': 4296192, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402196005}, {'Score': 0.8056577444076538, 'CONCEPT_NAME': 'Widespread dermatitis', 'CONCEPT_ID': 4298597, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402186001}, {'Score': 0.7999188899993896, 'CONCEPT_NAME': 'Early childhood dermatitis', 'CONCEPT_ID': 4298599, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402195009}, {'Score': 0.7992758750915527, 'CONCEPT_NAME': 'Facial dermatitis', 'CONCEPT_ID': 4298598, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402192007}]}
Please note that the medical coding is based on a mockup ontology. Results will not be reliable
SQL filled:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN (4296192,4298597,4298599,4298598)
AND p.gender_concept_id = 8532;
医療コーディングステップはここでのみ例示されることに注意してください。信頼できる結果を得るには、完全なスノムオントロジーにアクセスできる必要があります( Medical coding compilationセクションを参照)。