Ce référentiel contient le code du document suivant:
@misc{ziletti2024retrieval,
title={Retrieval augmented text-to-SQL generation for epidemiological question answering using electronic health records},
author={Angelo Ziletti and Leonardo D'Ambrosi},
year={2024},
eprint={2403.09226},
archivePrefix={arXiv},
primaryClass={cs.CL}
}
Veuillez trouver la préimpression ici: Ziletti et D'Ambrosi, https://arxiv.org/abs/2403.09226
Cet article est accepté lors de l'atelier NLP clinique NAACL 2024 (https://clinical-nlp.github.io/2024/)
Veuillez citer ce travail si vous utilisez ce code dans votre travail ou votre recherche.
Voici un flux de travail sommaire de la procédure: 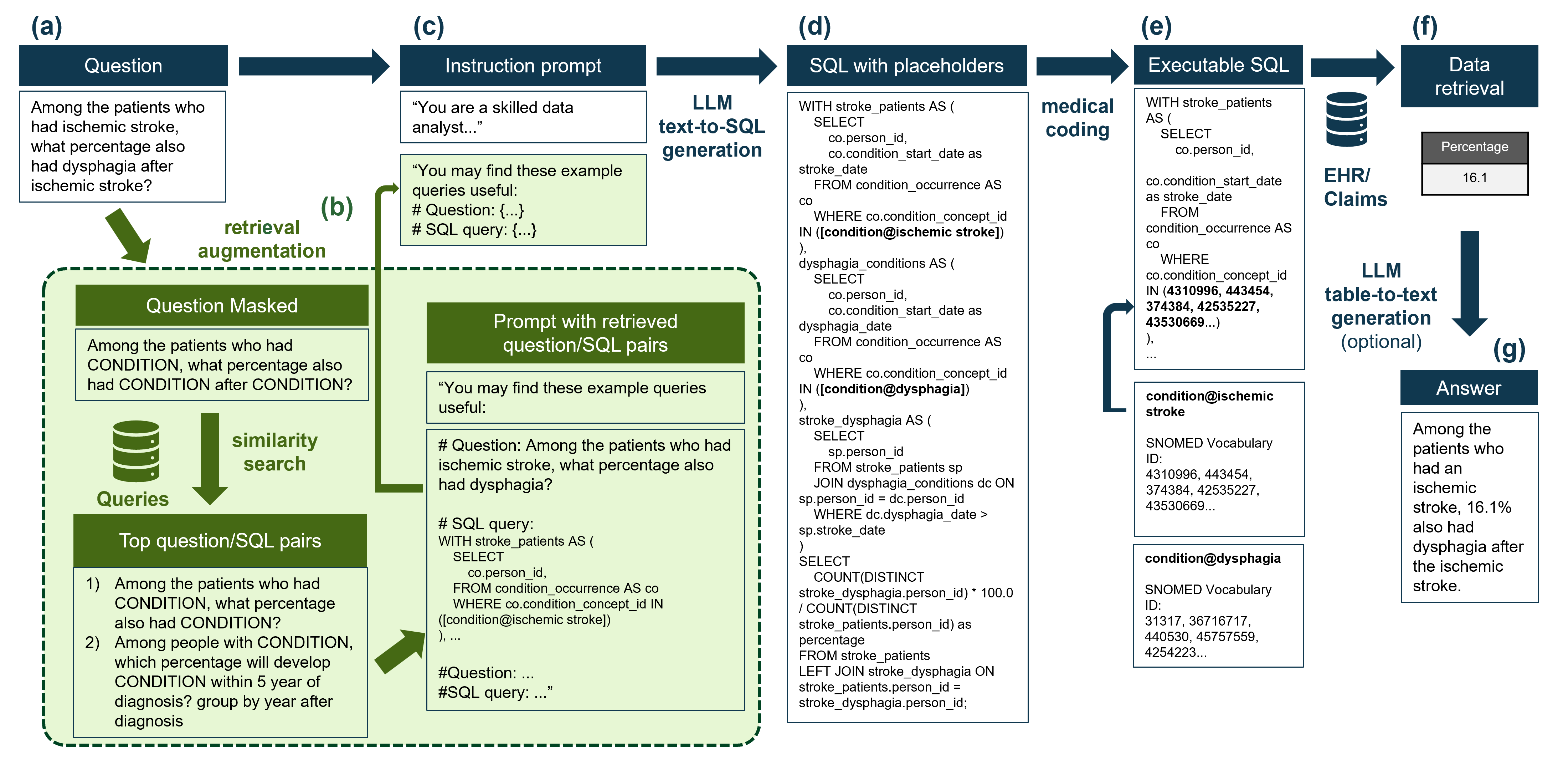 Et voici les principaux résultats:
Et voici les principaux résultats: 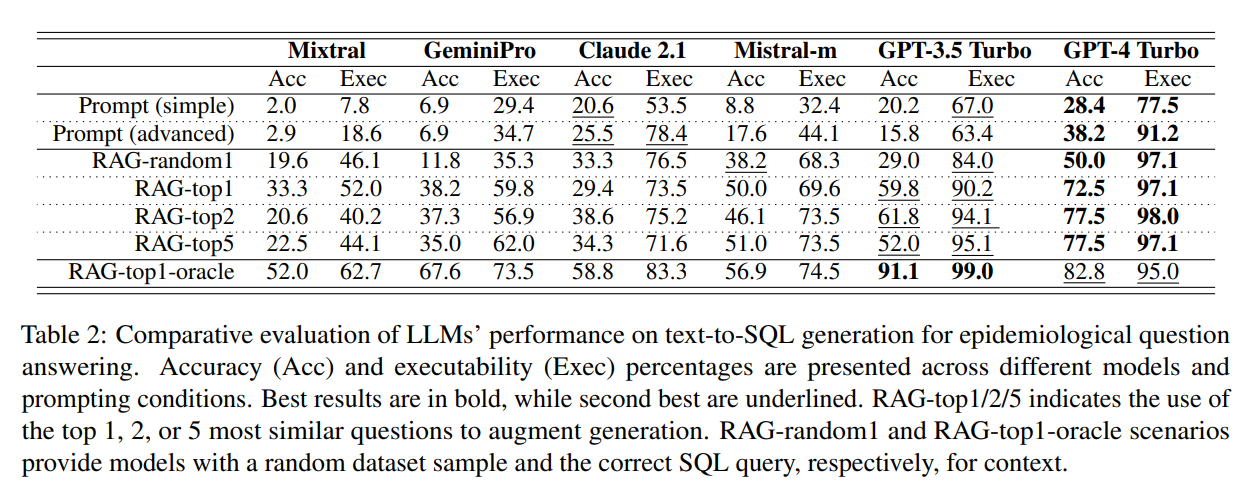
Veuillez vous référer au manuscrit pour plus de détails: https://arxiv.org/abs/2403.09226
Les paires de questions-sql étiquetées sont dans le fichier text2sql_epi_dataset_omop.xlsx dans le dossier dataset .
Nous avons utilisé Python 3.11 dans nos expériences.
Installez la dernière version de la branche maître sur github par:
git clone <GITHUB-URL>
cd text-to-sql-epi-ehr-naacl2024
pip install -r requirements.txt
Tout d'abord, nous devons créer un fichier Pickle (la bibliothèque de requêtes) à partir du fichier Excel fourni dans le dossier de dataset
cd scripts
python run_querylib_calc.py
Ce fichier de cornichon sera ensuite utilisé pour effectuer la génération augmentée (RAG) de récupération au stade de la génération de requêtes.
Pour effectuer une prédiction, exécutez le script prediction_pipeline.py avec votre question en doubles guillemets. Par exemple,
cd scripts
python prediction_pipeline.py --question "How many women with atopic dermatitis?"
Cela devrait revenir:
Question: How many women with atopic dermatitis?
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Pour exécuter la requête, vous serez ici un lien vers l'ensemble de données sur Google Cloud: https://console.cloud.google.com/marketplace/product/hhs/synpuf, vous pouvez ensuite ouvrir le lien (vous aurez besoin d'un compte Google) et exécuter la requête dans votre compte Google.
Pour rendre l'exécutable de requête sur Google BigQuery, vous devrez modifier les noms générés de la table pour vous conformer aux noms de table de requête Google. Il s'agit simplement d'un changement de nom des tables.
Par exemple,
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM person AS p
JOIN condition_occurrence AS co ON p.person_id = co.person_id
JOIN concept AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
doit être changé en
SELECT
COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM `bigquery-public-data.cms_synthetic_patient_data_omop.person` AS p
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.condition_occurrence` AS co ON p.person_id = co.person_id
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.concept` AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
Pour fonctionner sur l'instance BigQuery ci-dessus.
Si vous avez votre propre entrepôt de données Snowflake, vous n'aurez peut-être pas besoin d'effectuer ces changements.
Dans le référentiel, nous fournissons une version de maquette pour le codage médical pour montrer comment le codage médical peut être intégré dans le processus, comme le montre la figure ci-dessus. Le codage est effectué dans l'ontologie SNOMED, car il s'agit de l'ontologie sous-jacente pour les données stockées dans le modèle de données Common OPOP (OMOP-CDM). Le tableau est généralement appelé concept_table dans l'OMOP-CDM.
Nous avons préparé une petite table de maquette dans le dossier de dataset appelé medcodes_mockup.xlsx . Ici, nous fournissons des données synthétiques suivant la structure des données de l'OMOP-CDM. Vous pouvez télécharger toute l'ontologie de la National Library of Medicine ici. (Veuillez noter que vous devez accepter leurs termes et conditions)
Plus d'informations sur Snomed-CT peuvent être trouvées par exemple ici
Tout d'abord, nous devons créer un fichier Pickle (la bibliothèque MedCodeonto) à partir du fichier Excel fourni
cd scripts
python run_medcoding_calc.py
Ce fichier de cornichon sera ensuite utilisé pour rechercher des codes médicaux dans le fichier Pickle. L'exécution du fichier fournira également un exemple de la façon dont le codage est effectué.
Notez qu'il s'agit seulement d'une implémentation de maquette. Pour une version prête pour la production, nous suggérons d'utiliser une base de données vectorielle (par exemple QDRANT), indexant l'ensemble du tableau concept par exemple à partir d'une base de données Snowflake.
Après avoir exécuté l'étape Medical coding compilation , vous êtes prêt à exécuter le pipeline complet: SQL Query Generation + Medical Coding.
Ceci est un exemple:
cd scripts
python prediction_pipeline.py --med_coding True --question "How many females with atopic dermatitis"
Cela reviendra
Use medical coding: True
Use Snowflake database: False
No sentence-transformers model found with name BAAI/bge-large-en-v1.5. Creating a new one with MEAN pooling.
Question: How many females with atopic dermatitis
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Loading embedding from C:UsersGKENYOneDrive - BayerPersonal Dataascenttext-to-sql-epi-ehr-naacl2024data_outmedcodes_onto.pkl
Retrieved codes: {'atopic dermatitis': [{'Score': 0.8221014142036438, 'CONCEPT_NAME': 'Dermatitis in children', 'CONCEPT_ID': 4296192, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402196005}, {'Score': 0.8056577444076538, 'CONCEPT_NAME': 'Widespread dermatitis', 'CONCEPT_ID': 4298597, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402186001}, {'Score': 0.7999188899993896, 'CONCEPT_NAME': 'Early childhood dermatitis', 'CONCEPT_ID': 4298599, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402195009}, {'Score': 0.7992758750915527, 'CONCEPT_NAME': 'Facial dermatitis', 'CONCEPT_ID': 4298598, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402192007}]}
Please note that the medical coding is based on a mockup ontology. Results will not be reliable
SQL filled:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN (4296192,4298597,4298599,4298598)
AND p.gender_concept_id = 8532;
Veuillez noter que l'étape de codage médical n'est que exemplaire ici. Pour obtenir des résultats fiables, vous devrez avoir accès à l'ontologie SNOMED complète (voir la section Medical coding compilation ).