Este repositório contém o código do seguinte artigo:
@misc{ziletti2024retrieval,
title={Retrieval augmented text-to-SQL generation for epidemiological question answering using electronic health records},
author={Angelo Ziletti and Leonardo D'Ambrosi},
year={2024},
eprint={2403.09226},
archivePrefix={arXiv},
primaryClass={cs.CL}
}
Encontre a pré -impressão aqui: Ziletti e D'Ambrosi, https://arxiv.org/abs/2403.09226
Este artigo é aceito no NAACL 2024 Clinical PNL Workshop (https://clinical-nlp.github.io/2024/)
Cite este trabalho se você usar este código em seu trabalho ou pesquisa.
Aqui está um fluxo de trabalho resumido do procedimento: 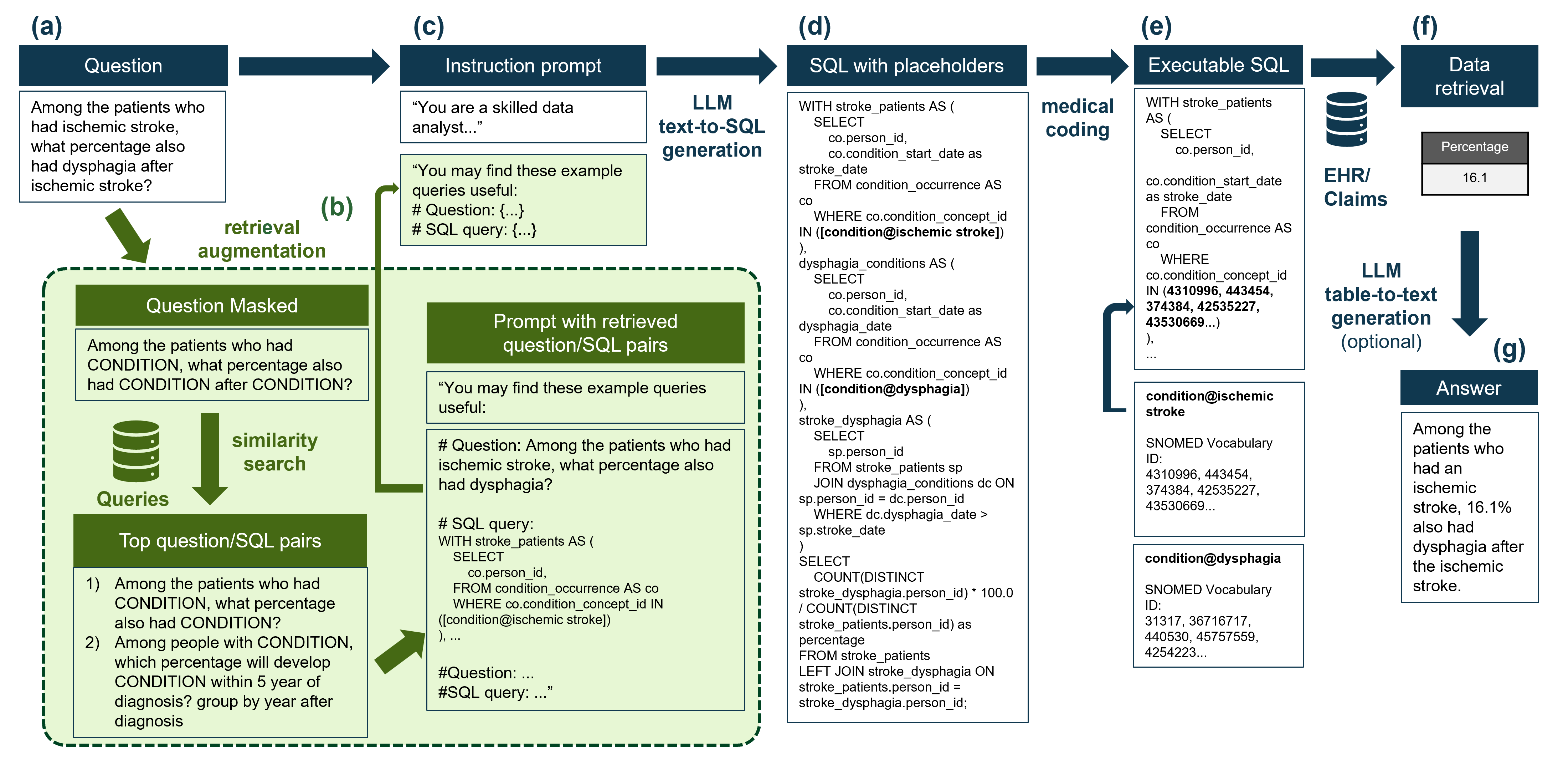 E aqui estão os principais resultados:
E aqui estão os principais resultados: 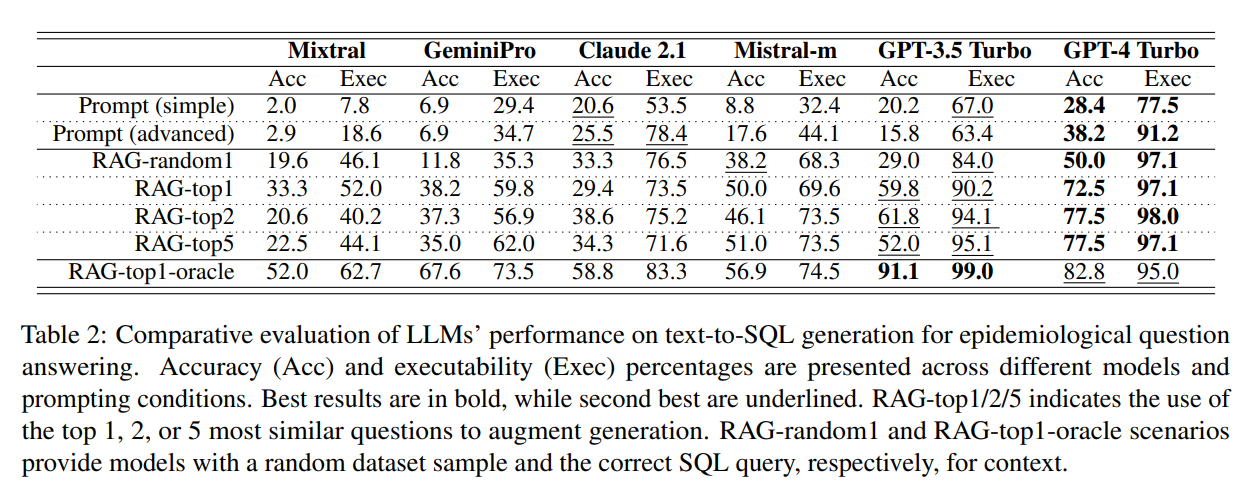
Consulte o manuscrito para obter mais detalhes: https://arxiv.org/abs/2403.09226
Os pares de perguntas-SQL rotuladas estão no arquivo text2sql_epi_dataset_omop.xlsx na pasta dataset .
Usamos o Python 3.11 em nossos experimentos.
Instale a versão mais recente do The Master Branch no GitHub por:
git clone <GITHUB-URL>
cd text-to-sql-epi-ehr-naacl2024
pip install -r requirements.txt
Primeiro, precisamos criar um arquivo de picles (a biblioteca de consultas) a partir do arquivo Excel fornecido na pasta dataset
cd scripts
python run_querylib_calc.py
Este arquivo de picles será usado para executar a geração aumentada de recuperação (RAG) no estágio de geração de consultas.
Para realizar uma previsão, execute o script prediction_pipeline.py com sua pergunta em cotações duplas. Por exemplo,
cd scripts
python prediction_pipeline.py --question "How many women with atopic dermatitis?"
Isso deve retornar:
Question: How many women with atopic dermatitis?
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Para executar a consulta, você estará aqui um link para o conjunto de dados no Google Cloud: https://console.cloud.google.com/marketplace/product/hhs/synpuf Você pode abrir o link (precisará de uma conta do Google) e executar a consulta em sua conta do Google.
Para fazer a consulta executável no Google BigQuery, você precisará modificar os nomes gerados da tabela para cumprir os nomes da tabela de consultas do Google. Isso é simplesmente uma renomeação das tabelas.
Por exemplo,
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM person AS p
JOIN condition_occurrence AS co ON p.person_id = co.person_id
JOIN concept AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
deve ser alterado para
SELECT
COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM `bigquery-public-data.cms_synthetic_patient_data_omop.person` AS p
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.condition_occurrence` AS co ON p.person_id = co.person_id
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.concept` AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
Para executar na instância BigQuery acima.
Se você tiver seu próprio data warehouse de floco de neve, talvez não precise executar essas alterações.
No repositório, fornecemos uma versão de maquete para a codificação médica para mostrar como a codificação médica pode ser integrada no processo, conforme mostrado na figura acima. A codificação é realizada dentro da ontologia snomed, pois essa é a ontologia subjacente para dados armazenados no Modelo de Dados Comuns da OMOP (OMOP-CDM). A tabela é geralmente chamada de conceito_table no OMOP-CDM.
Preparamos uma pequena tabela de maquete na pasta dataset chamada medcodes_mockup.xlsx . Aqui fornecemos dados sintéticos seguindo a estrutura de dados do OMOP-CDM. Você pode baixar toda a ontologia da Biblioteca Nacional de Medicina aqui. (Observe que você precisa aceitar seus termos e condições)
Mais informações sobre Snomed-CT podem ser encontradas, por exemplo, aqui
Primeiro, precisamos criar um arquivo de picles (a biblioteca MedCodeOnto) a partir do arquivo do Excel fornecido
cd scripts
python run_medcoding_calc.py
Este arquivo de picles será usado para pesquisar códigos médicos dentro do arquivo de picles. A execução do arquivo também fornecerá um exemplo de como a codificação é executada.
Observe que esta é apenas uma implementação de maquete. Para uma versão pronta para produção, sugerimos usar um banco de dados vetorial (por exemplo, QDRANT), indexando toda a tabela conceitual, por exemplo, de um banco de dados de floco de neve.
Depois de executar a etapa Medical coding compilation , você está pronto para executar o pipeline completo: geração de consulta SQL + codificação médica.
Este é um exemplo:
cd scripts
python prediction_pipeline.py --med_coding True --question "How many females with atopic dermatitis"
Isso retornará
Use medical coding: True
Use Snowflake database: False
No sentence-transformers model found with name BAAI/bge-large-en-v1.5. Creating a new one with MEAN pooling.
Question: How many females with atopic dermatitis
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Loading embedding from C:UsersGKENYOneDrive - BayerPersonal Dataascenttext-to-sql-epi-ehr-naacl2024data_outmedcodes_onto.pkl
Retrieved codes: {'atopic dermatitis': [{'Score': 0.8221014142036438, 'CONCEPT_NAME': 'Dermatitis in children', 'CONCEPT_ID': 4296192, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402196005}, {'Score': 0.8056577444076538, 'CONCEPT_NAME': 'Widespread dermatitis', 'CONCEPT_ID': 4298597, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402186001}, {'Score': 0.7999188899993896, 'CONCEPT_NAME': 'Early childhood dermatitis', 'CONCEPT_ID': 4298599, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402195009}, {'Score': 0.7992758750915527, 'CONCEPT_NAME': 'Facial dermatitis', 'CONCEPT_ID': 4298598, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402192007}]}
Please note that the medical coding is based on a mockup ontology. Results will not be reliable
SQL filled:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN (4296192,4298597,4298599,4298598)
AND p.gender_concept_id = 8532;
Observe que a etapa de codificação médica é apenas exemplificatória aqui. Para obter resultados confiáveis, você precisará ter acesso à ontologia snomed completa (consulte a seção Medical coding compilation ).