Dieses Repository enthält den Code des folgenden Papiers:
@misc{ziletti2024retrieval,
title={Retrieval augmented text-to-SQL generation for epidemiological question answering using electronic health records},
author={Angelo Ziletti and Leonardo D'Ambrosi},
year={2024},
eprint={2403.09226},
archivePrefix={arXiv},
primaryClass={cs.CL}
}
Weitere Informationen finden Sie hier: Ziletti und D'Ambrosi, https://arxiv.org/abs/2403.09226
Dieses Papier wird bei NAACL 2024 Clinical NLP Workshop (https://clinical-nlp.github.io/2024/) akzeptiert
Bitte zitieren Sie diese Arbeit, wenn Sie diesen Code in Ihrer Arbeit oder Forschung verwenden.
Hier ist ein zusammenfassender Workflow des Verfahrens: 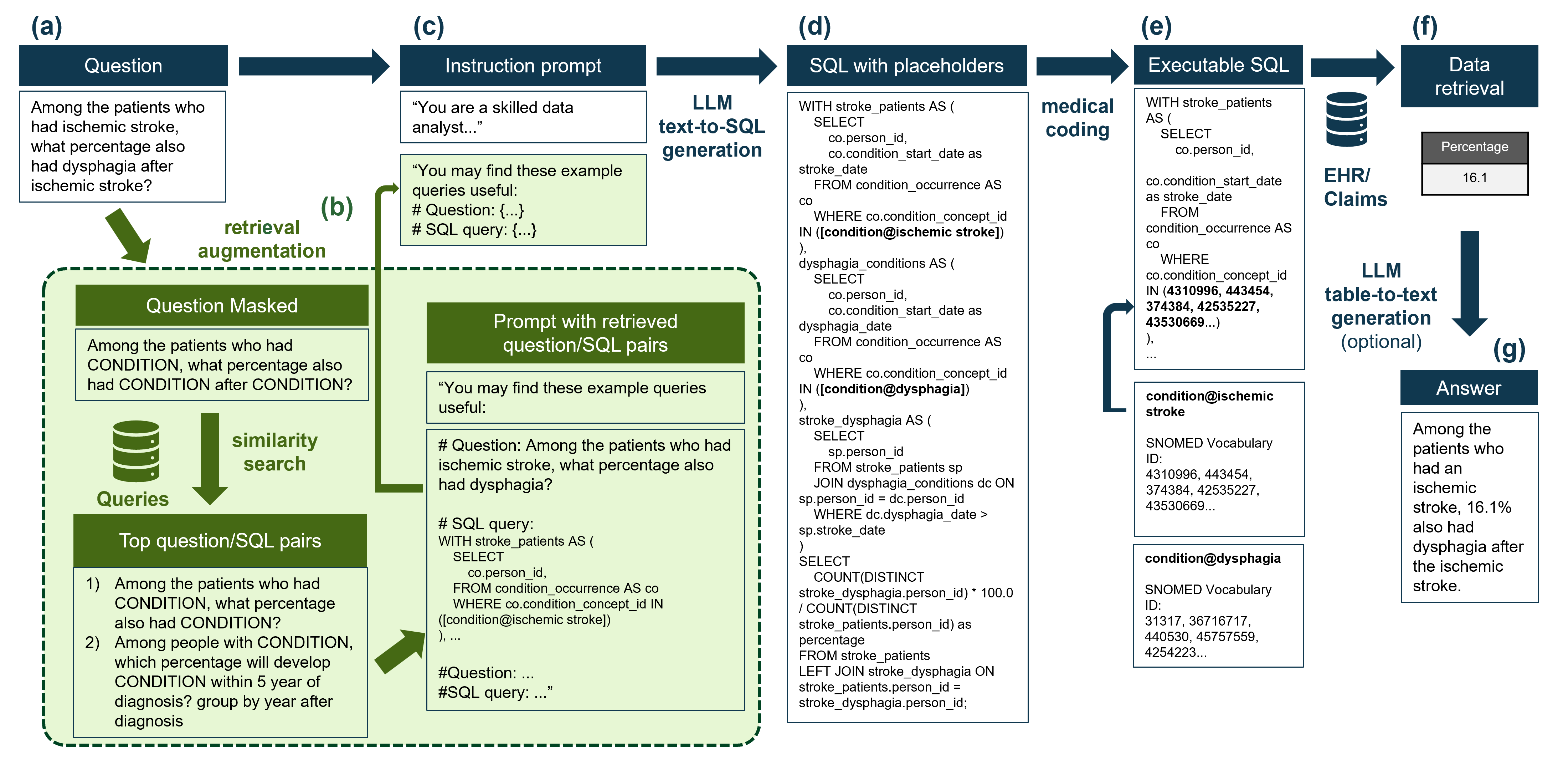 Und hier sind die Hauptergebnisse:
Und hier sind die Hauptergebnisse: 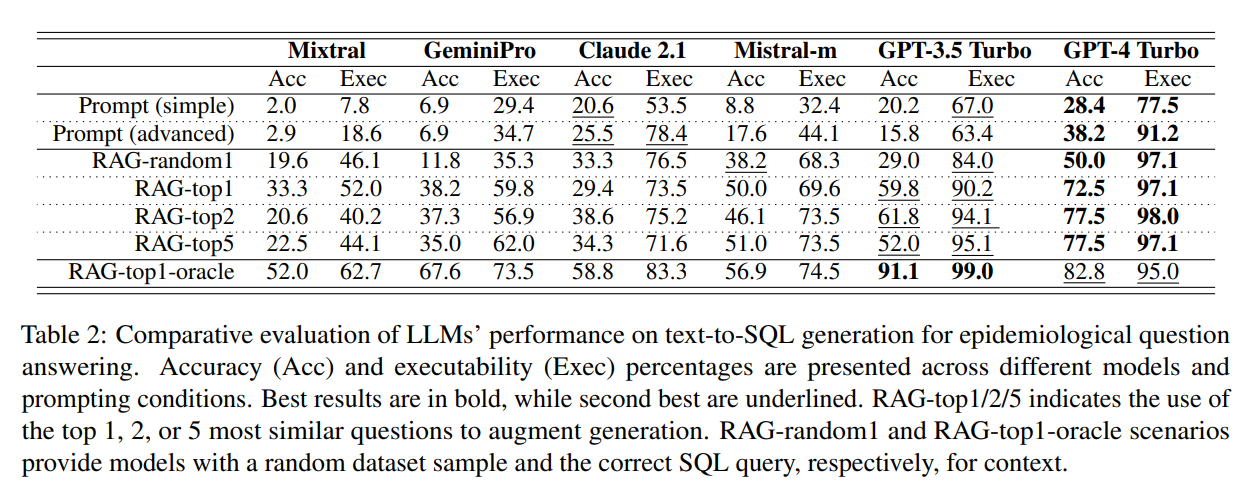
Weitere Informationen finden Sie im Manuskript: https://arxiv.org/abs/2403.09226
Die gekennzeichneten Fragen-SQL-Paare befinden sich im Datei text2sql_epi_dataset_omop.xlsx im dataset Ordner.
Wir haben Python 3.11 in unseren Experimenten verwendet.
Installieren Sie die neueste Version aus der Master -Filiale in GitHub von:
git clone <GITHUB-URL>
cd text-to-sql-epi-ehr-naacl2024
pip install -r requirements.txt
Zunächst müssen wir eine Gurkendatei (die Abfragebibliothek) aus der Excel -Datei im dataset erstellen
cd scripts
python run_querylib_calc.py
Diese Gurkendatei wird dann verwendet, um die Abruf Augmented Generation (LAB) in der Abfrageerzeugungsphase durchzuführen.
Um eine Vorhersage durchzuführen, führen Sie die Skript prediction_pipeline.py mit Ihrer Frage in doppelten Zitaten aus. Zum Beispiel,
cd scripts
python prediction_pipeline.py --question "How many women with atopic dermatitis?"
Dies sollte zurückkehren:
Question: How many women with atopic dermatitis?
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Um die Abfrage auszuführen, finden Sie hier einen Link zum Datensatz in Google Cloud: https://console.cloud.google.com/marketplace/product/hhs/synpuf Sie können dann den Link öffnen (benötigen ein Google -Konto) und die Abfrage in Ihrem Google -Konto ausführen.
Um die Abfrage in Google BigQuery ausführbar zu machen, müssen Sie die generierten Namen der Tabelle ändern, um den Namen der Google Query -Tabelle einzuhalten. Dies ist einfach eine Umbenennung der Tische.
Zum Beispiel,
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM person AS p
JOIN condition_occurrence AS co ON p.person_id = co.person_id
JOIN concept AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
muss in geändert werden in
SELECT
COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM `bigquery-public-data.cms_synthetic_patient_data_omop.person` AS p
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.condition_occurrence` AS co ON p.person_id = co.person_id
JOIN `bigquery-public-data.cms_synthetic_patient_data_omop.concept` AS c ON co.condition_concept_id = c.concept_id
WHERE p.gender_concept_id = 8532
AND c.concept_id IN (133834,4298597,4066382,4298599,4296193,4080929,4296192,4290738,4290734,4206125,4290736,4080928,4033671,4031630,4297478,4296190,4031631,4080927,4298598,4298601,4031013,4297362,4290740,4297495,40482226,4298600,4236759);
auf der obigen BigQuery -Instanz laufen.
Wenn Sie Ihr eigenes Snowflake Data Warehouse haben, müssen Sie diese Änderungen möglicherweise nicht vornehmen.
Im Repository bieten wir eine Modellversion für die medizinische Codierung, um zu zeigen, wie medizinische Codierung in den Prozess integriert werden kann, wie in der obigen Abbildung gezeigt. Die Codierung wird innerhalb der Snomed-Ontologie durchgeführt, da dies die zugrunde liegende Ontologie für Daten ist, die im OMOP Common Data Model (OMOP-CDM) gespeichert sind. Die Tabelle wird normalerweise im OMOP-CDM CONCECT_TABLE bezeichnet.
Wir haben eine kleine Modellentelle im dataset mit dem Namen medcodes_mockup.xlsx erstellt. Hier liefern wir synthetische Daten nach der Datenstruktur des OMOP-CDM. Sie können die gesamte Ontologie hier aus der National Library of Medicine herunterladen. (Bitte beachten Sie, dass Sie ihre Allgemeinen Geschäftsbedingungen akzeptieren müssen)
Weitere Informationen zu SNOMED-CT finden Sie beispielsweise hier
Zunächst müssen wir eine Gurkendatei (die MedCodeonto -Bibliothek) aus der angegebenen Excel -Datei erstellen
cd scripts
python run_medcoding_calc.py
Diese Wickle -Datei wird dann verwendet, um in der Gurkendatei nach medizinischen Codes zu suchen. Das Ausführen der Datei liefert auch ein Beispiel dafür, wie die Codierung durchgeführt wird.
Beachten Sie, dass dies nur eine Modellimplementierung ist. Für eine produktionsbereite Version empfehlen wir, eine Vektor-Datenbank (z. B. QDRant) zu verwenden, wobei die gesamte Konzepttabelle beispielsweise aus einer Snowflake-Datenbank indiziert wird.
Nachdem Sie den Schritt der Medical coding compilation ausgeführt haben, können Sie die vollständige Pipeline: SQL -Abfrage -Generierung + medizinische Codierung ausführen.
Dies ist ein Beispiel:
cd scripts
python prediction_pipeline.py --med_coding True --question "How many females with atopic dermatitis"
Dies wird zurückkehren
Use medical coding: True
Use Snowflake database: False
No sentence-transformers model found with name BAAI/bge-large-en-v1.5. Creating a new one with MEAN pooling.
Question: How many females with atopic dermatitis
SQL template:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN ([condition@atopic dermatitis])
AND p.gender_concept_id = 8532;
Loading embedding from C:UsersGKENYOneDrive - BayerPersonal Dataascenttext-to-sql-epi-ehr-naacl2024data_outmedcodes_onto.pkl
Retrieved codes: {'atopic dermatitis': [{'Score': 0.8221014142036438, 'CONCEPT_NAME': 'Dermatitis in children', 'CONCEPT_ID': 4296192, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402196005}, {'Score': 0.8056577444076538, 'CONCEPT_NAME': 'Widespread dermatitis', 'CONCEPT_ID': 4298597, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402186001}, {'Score': 0.7999188899993896, 'CONCEPT_NAME': 'Early childhood dermatitis', 'CONCEPT_ID': 4298599, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402195009}, {'Score': 0.7992758750915527, 'CONCEPT_NAME': 'Facial dermatitis', 'CONCEPT_ID': 4298598, 'DOMAIN_ID': 'Condition', 'VOCABULARY_ID': 'SNOMED', 'STANDARD_CONCEPT': 'S', 'CONCEPT_CODE': 402192007}]}
Please note that the medical coding is based on a mockup ontology. Results will not be reliable
SQL filled:
SELECT COUNT(DISTINCT p.person_id) AS female_patients_with_atopic_dermatitis
FROM condition_occurrence AS co
JOIN person AS p ON co.person_id = p.person_id
WHERE co.condition_concept_id IN (4296192,4298597,4298599,4298598)
AND p.gender_concept_id = 8532;
Bitte beachten Sie, dass der medizinische Codierungsschritt hier nur ein Beispiel ist. Um zuverlässige Ergebnisse zu erzielen, müssen Sie Zugang zur vollständigen Snomed -Ontologie haben (siehe Abschnitt Medical coding compilation ).