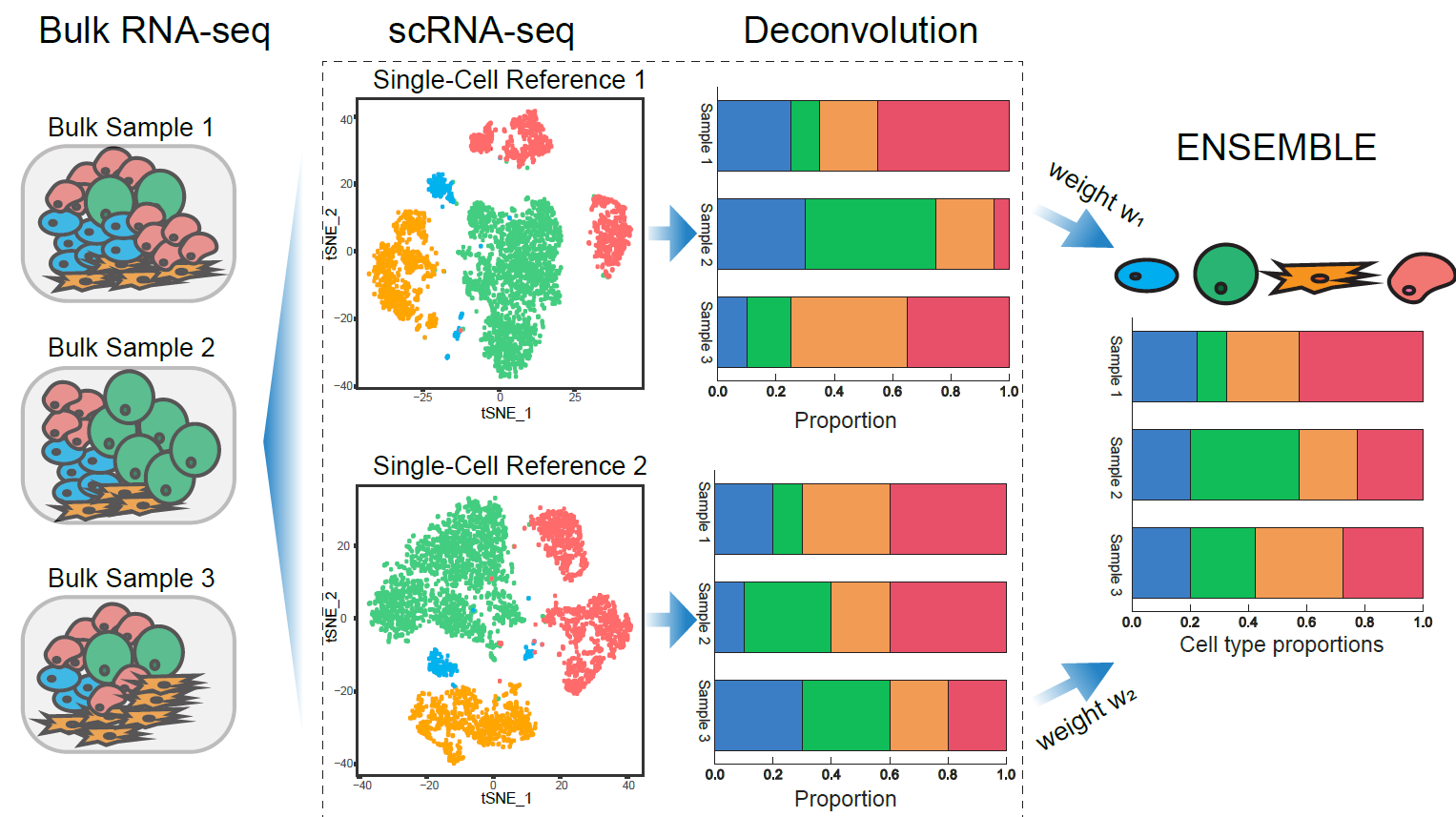
SCDC-это метод деконволюции для объемной РНК-seq, который использует специфические генные экспрессии клеточного типа из множества эталонных наборов данных SCRNA-seq . SCDC принимает метод ансамбля для интеграции результатов деконволюции из различных наборов данных SCRNA-Seq, которые производятся в разных лабораториях и в разное время, неявное обращение к пакетному эффекту.
Мейхен Донг, Аатиш Теннаван, Юджин Уррутия, Юн Ли, Чарльз М. Пеу, Фей Зу, Ючао Цзян, SCDC: Деконволюция экспрессии гена с помощью множественных ссылок на РНК с одной ячейкой, // doi. org/10.1093/bib/bbz166
Лицензия: MIT
Вы можете установить выпущенную версию SCDC из GitHub с:
if (! require ("devtools")) {
install.packages ("devtools")
} devtools :: install_github ("meichendong/scdc")Проблема пакета зависимостей относительно «xbioc» может быть решена:
install.packages ("remotes") remotes :: install_github ("renozao/xbioc")Пожалуйста, смотрите страницу Vignettes.
Бумага SCDC опубликована на брифингах в биоинформатике.
Вопросы, касающиеся пакета, можно отправить по электронной почте: [email protected]
Когда в одном ячейке есть только «один субъект/человек», используйте функции SCDC_qc_ONE() , SCDC_prop_ONE() .
Аспекты, которые могут повлиять на результаты деконволюции:
Формат данных: Образцы объемного и одноклеточных образцов как необработанные подсчеты / тот же формат? Мы ожидаем, что формат данных будет последовательным и сопоставимым.
Фильтрация генов: вы отфильтровали с низко экспрессированными генами / рибосомными генами / митохондриальными генами? Эти гены могут повлиять на анализ нижнего течения.
Размер ячейки и коэффициенты размера библиотеки: для одной ячейки вы думаете, что сумма всех генов (размер библиотеки) может отражать ее реальный размер ячейки? Это одно из наших предположений: отношение размеров библиотеки между типами клеток может отражать отношение реальных размеров клеток между типами клеток. Если нет, вы можете вручную ввести коэффициент размера ячейки при построении «базисной матрицы».
Подобные типы клеток: существуют ли типы клеток, которые потенциально могут запутать анализ? Например, типы клеток, которые имеют очень похожие профили /маркерные гены.
Отсутствуют основные типы ячеек / технические проблемы: ожидаете ли вы, что процедура секвенирования изменит большое значение в объеме и SC. Даже техника такая же? Иногда ссылки на одно ячейки могут потерять информацию для некоторых типов ячеек. Например, в ваших объемных образцах есть жировые клетки, но почему -то у вас их нет для данных с одной ячейкой.
Деконволюция с использованием одного эталонного набора данных: вы пытались использовать один справочный набор данных, чтобы проверить, имеют ли результаты в целом? Я вижу, ты пробовал бисквит. Вы пробовали другие методы, такие как Cibersortx? Если результаты других методов деконволюции «одноразовый» деконволюции имеют больше смысла, то вы можете ввести их непосредственно, используя наш ансамбль.