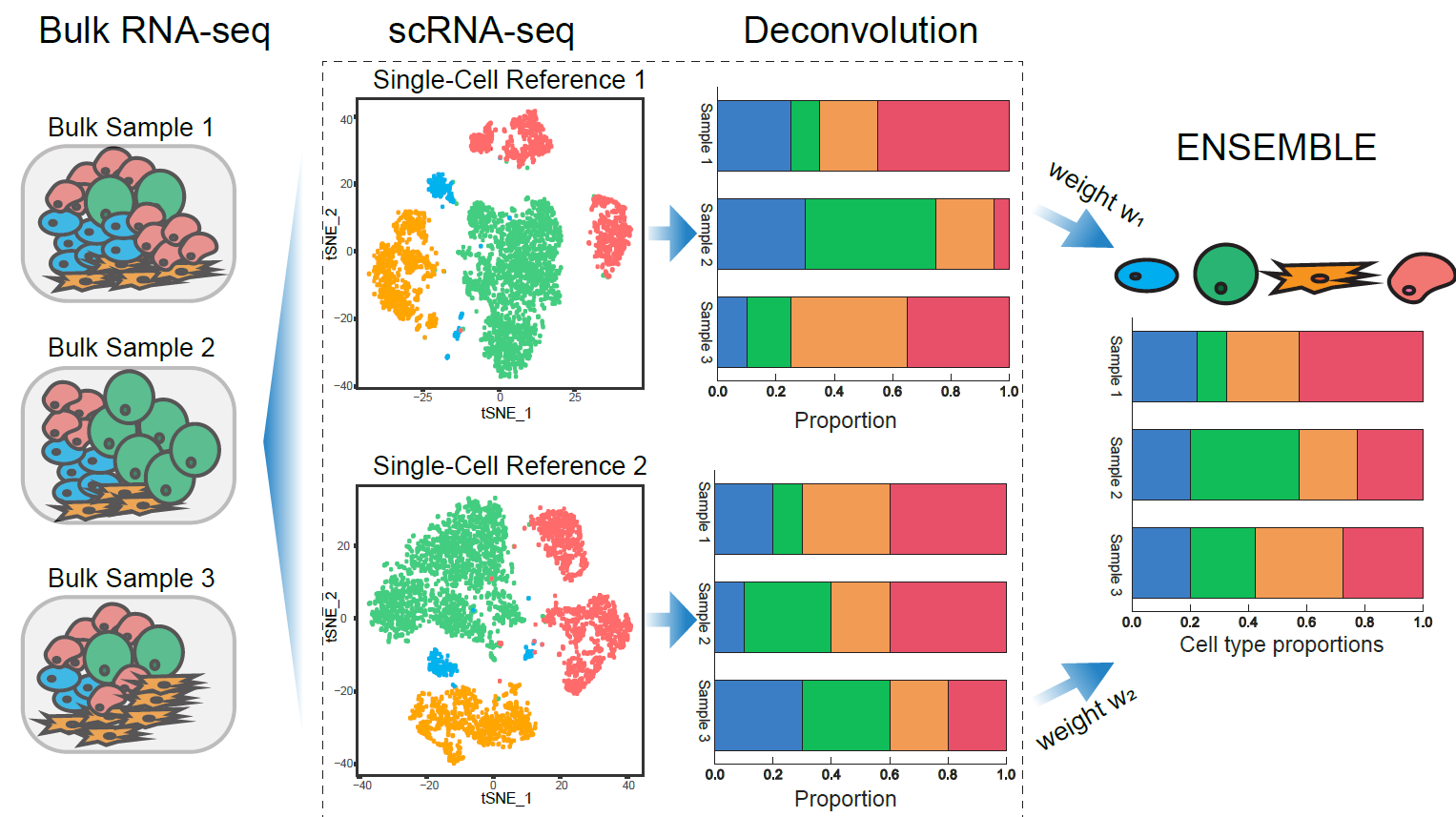
SCDC adalah metode dekonvolusi untuk RNA-seq curah yang memanfaatkan ekspresi gen spesifik tipe sel dari beberapa dataset referensi SCRNA-seq . SCDC mengadopsi metode ensemble untuk mengintegrasikan hasil dekonvolusi dari dataset SCRNA-seq yang berbeda yang diproduksi di laboratorium yang berbeda dan pada waktu yang berbeda, secara implisit menangani perancu efek batch.
Meichen Dong, Aatish Thennavan, Eugene Urrutia, Yun Li, Charles M Perou, Fei Zou, Yuchao Jiang, SCDC: Dekonvolusi Ekspresi Gen Bulk oleh beberapa referensi sekuensing RNA sel tunggal, briefing dalam bioinformatika ,, bbz166, https: https: https: https: https: https: https. //. org/10.1093/bib/bbz166
Lisensi: MIT
Anda dapat menginstal versi SCDC yang dirilis dari GitHub dengan:
if (! membutuhkan ("devtools")) {
install.packages ("devtools")
} devtools :: install_github ("meichendong/scdc")Masalah Paket Ketergantungan tentang 'XBIOC' dapat diselesaikan dengan:
install.packages ("remotes") remote :: install_github ("renozao/xbioc")Silakan lihat halaman Vignettes.
Makalah SCDC diterbitkan di Briefings in Bioinformatics.
Pertanyaan tentang paket dapat diemail ke: [email protected]
Ketika hanya ada 'satu subjek/individu' dalam dataset sel tunggal, silakan gunakan fungsi SCDC_qc_ONE() , SCDC_prop_ONE() .
Aspek yang dapat mempengaruhi hasil dekonvolusi:
Format Data: Apakah sampel sel massal dan tunggal baik jumlah mentah / format yang sama? Kami berharap format data konsisten dan sebanding.
Penyaringan Gen: Apakah Anda menyaring gen / gen ribosom yang diekspresikan rendah / gen mitokondria? Gen -gen ini dapat mempengaruhi analisis hilir.
Ukuran sel dan faktor ukuran perpustakaan: Untuk satu sel, apakah menurut Anda jumlah semua jumlah gen (ukuran perpustakaan) dapat mencerminkan ukuran sel sebenarnya? Ini adalah salah satu asumsi kami: rasio ukuran perpustakaan antara jenis sel dapat mencerminkan rasio ukuran sel nyata antara jenis sel. Jika tidak, Anda dapat secara manual memasukkan faktor ukuran sel saat membangun "matriks basis".
Jenis sel yang serupa: Apakah ada jenis sel yang berpotensi mengacaukan analisis? Misalnya, tipe sel yang memiliki profil /gen penanda yang sangat mirip.
Jenis sel utama yang hilang / masalah teknis: Apakah Anda mengharapkan prosedur sekuensing untuk membuat perbedaan besar dalam curah dan SC bahkan tekniknya sama? Terkadang data referensi sel tunggal dapat kehilangan informasi untuk beberapa jenis sel. Misalnya, ada sel lemak dalam sampel curah Anda, tetapi entah bagaimana Anda tidak memilikinya untuk data sel tunggal.
Dekonvolusi Menggunakan Dataset Referensi Tunggal: Apakah Anda mencoba menggunakan satu dataset referensi untuk menguji jika hasilnya masuk akal secara umum? Saya melihat Anda mencoba Bisque. Sudahkah Anda mencoba metode lain seperti Cibersortx? Jika hasil dari metode dekonvolusi "satu-referensi" lainnya lebih masuk akal, maka Anda dapat memasukkan ini secara langsung menggunakan langkah ensemble kami.