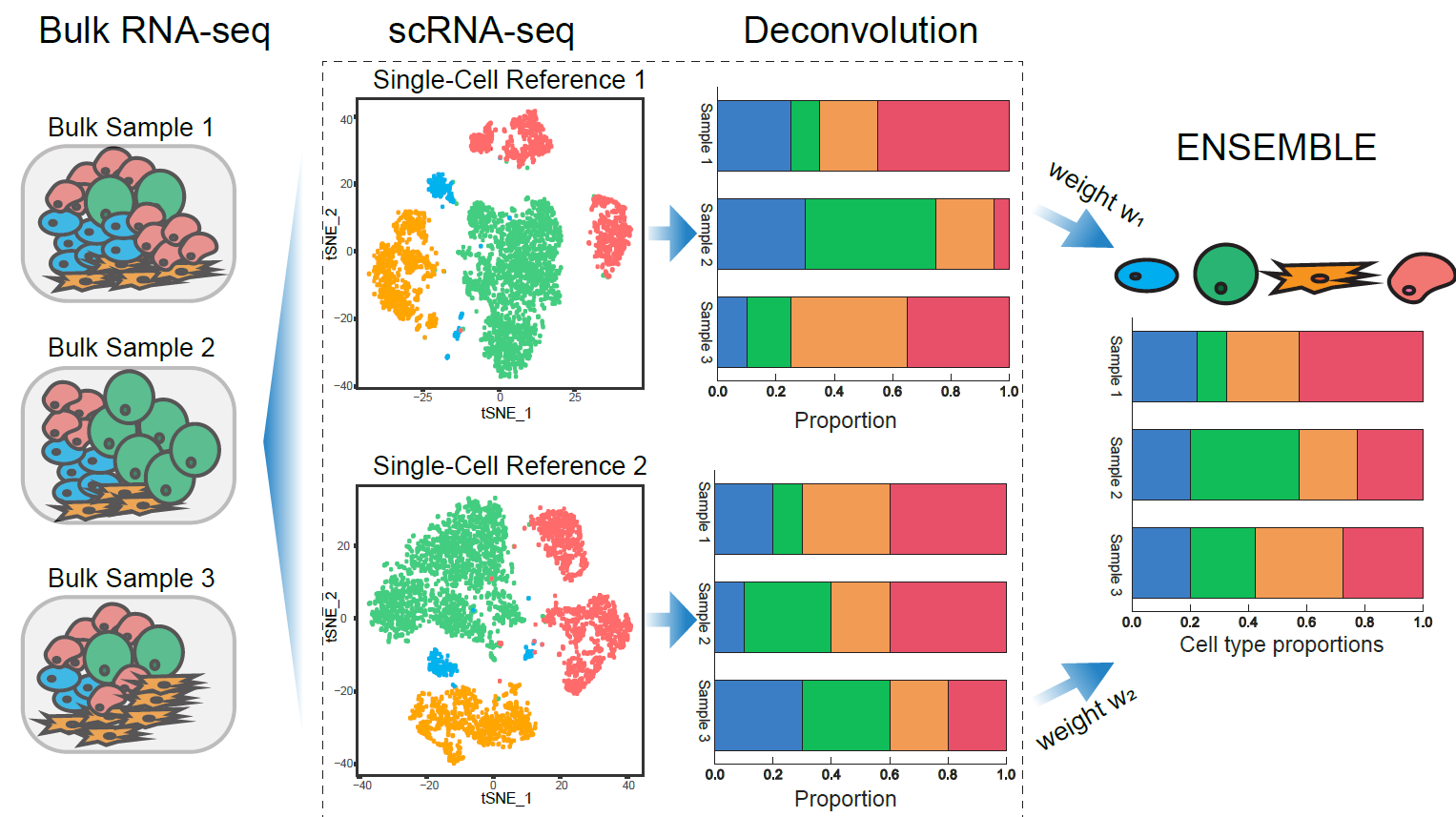
SCDCは、複数のSCRNA-SEQ参照データセットから細胞型特異的遺伝子発現を活用するバルクRNA-seqのデコンボリューション法です。 SCDCはアンサンブル法を採用して、異なる研究所やさまざまな時期に生成されるさまざまなSCRNA-seqデータセットのデコンボリューション結果を統合し、暗黙的にバッチ効果の交絡に対処します。
Meichen Dong、Aatish Thennavan、Eugene Urrutia、Yun Li、Charles M Perou、Fei Zou、Yuchao Jiang、Scdc:バルク遺伝子発現デコンボリューション、複数の単一細胞RNAシーケンス参照、バイオインフォマティクスのブリーフィング、Bbz166、 https://doi.org/10.1093/bib/bbz166
ライセンス:MIT
GithubからSCDCのリリースバージョンをインストールできます。
if(!require( "devtools"))){
install.packages( "devtools")
} devtools :: install_github( "meichendong/scdc")「XBIOC」に関する依存関係パッケージの問題は、次のように解決できます。
install.packages( "Remotes")Remotes :: install_github( "nemozao/xbioc")
ビネットページをご覧ください。
SCDCペーパーは、Bioinformaticsのブリーフィングで公開されています。
パッケージに関する質問は、[email protected]にメールで送信できます。
単一のセルデータセットに「1つのサブジェクト/個体」のみがある場合は、 SCDC_qc_ONE() 、 SCDC_prop_ONE()関数を使用してください。
デコンボリューションの結果に影響を与える可能性のある側面:
データ形式:バルクと単一のセルのサンプルは両方とも生のカウント /同じ形式ですか?データ形式は一貫性があり、比較可能であると予想しています。
遺伝子フィルタリング:低発現遺伝子 /リボソーム遺伝子 /ミトコンドリア遺伝子を除外しましたか?これらの遺伝子は、下流の分析に影響を与える可能性があります。
セルサイズとライブラリーサイズの要因:単一のセルの場合、すべての遺伝子数(ライブラリサイズ)の合計がその実際のセルサイズを反映できると思いますか?これは私たちの仮定の1つです。細胞型間のライブラリサイズの比率は、細胞型間の実際の細胞サイズの比を反映しています。そうでない場合は、「基底マトリックス」を構築するときにセルサイズ係数を手動で入力できます。
同様の細胞タイプ:分析を潜在的に混乱させる可能性のある細胞タイプはありますか?たとえば、非常に類似したプロファイル /マーカー遺伝子を持つ細胞タイプ。
主要な細胞タイプ /技術的な問題が欠落している:シーケンス手順がバルクとSCに大きな違いを生むことを期待していますか?単一のセル参照データが一部の細胞タイプの情報を失うことがあります。たとえば、バルクサンプルには脂肪細胞がありますが、どういうわけか単一の細胞データのためにそれを持っていません。
単一の参照データセットを使用したデコンボリューション:1つのリファレンスデータセットを使用して、結果が一般的に理にかなっているかどうかをテストしようとしましたか?ビスクを試してみました。 CiberSortxのような他の方法を試しましたか?他の「1つの参照」デコンボリューション方法の結果がより理にかなっている場合、アンサンブルステップを使用してこれらを直接入力できます。