SCDC ist eine Entfaltungsmethode für Bulk-RNA-seq, die spezifische Genexpressionen vom Typ Zellen aus mehreren SCRNA-Seq-Referenzdatensätzen nutzt. SCDC übernimmt eine Ensemble-Methode zur Integration von Entfaltungsergebnissen aus verschiedenen SCRNA-Seq-Datensätzen, die in verschiedenen Laboratorien und zu unterschiedlichen Zeiten hergestellt werden, wobei implizit die Stapel-Effekt-Verwirrung angesprochen wird.
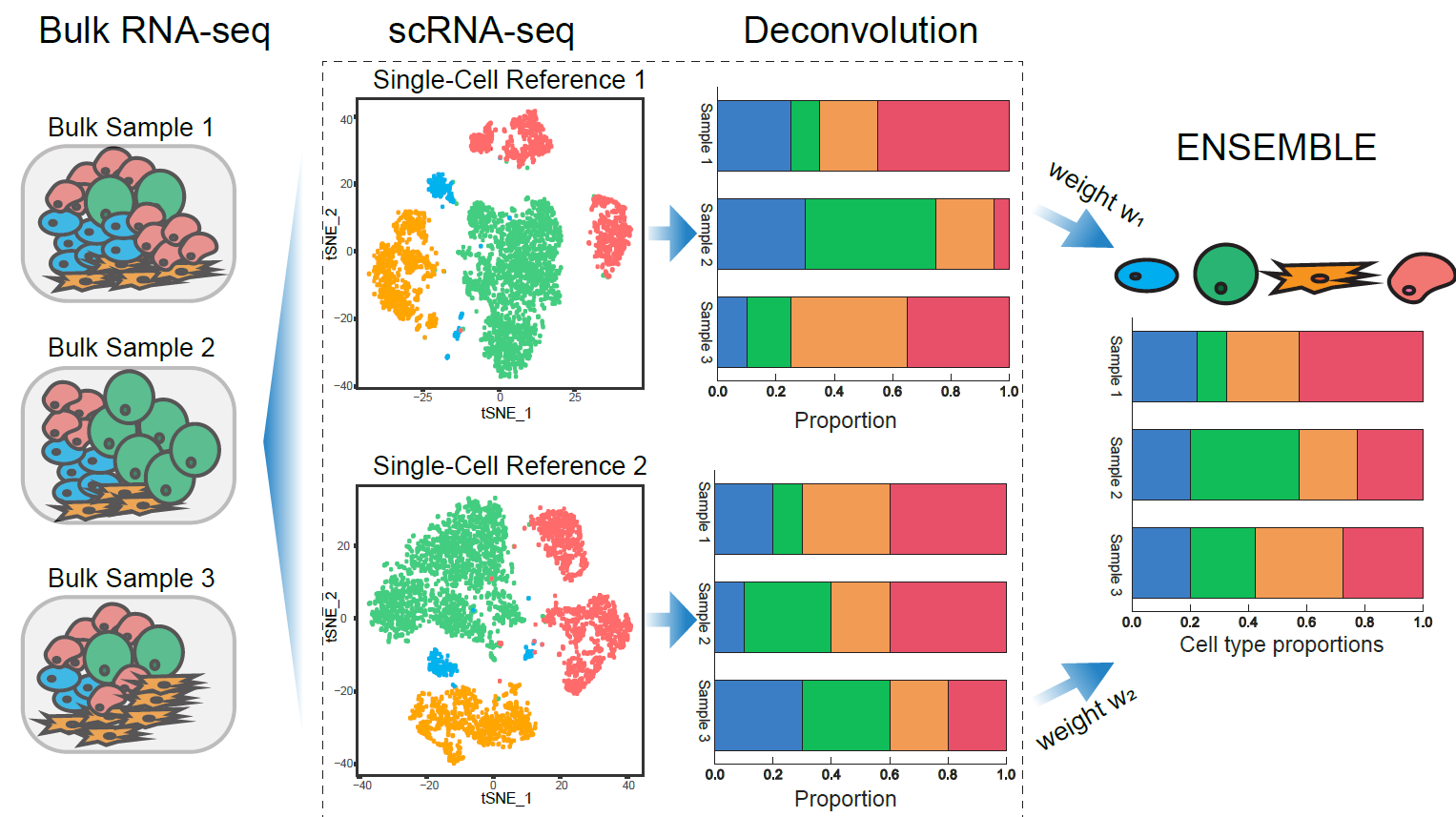
Michen Dong, aatish thennavan, Eugene Urrutia, Yun Li, Charles M Perou, Fei Zou, Yuchao Jiang, SCDC: Bulk-Genexpression Dekonvolution durch mehrere Einzelzell-RNA-Sequenzierungsreferenzen, Briefings in Bioinformatik, BBZ166, HTTPS://///DOI. org/10.1093/bib/bbz166
Lizenz: MIT
Sie können die freigegebene Version von SCDC von GitHub installieren mit:
if (! Erfordert ("devtools")) {
install.packages ("devtools")
} devtools :: install_github ("meichendong/scdc")Das Problem des Abhängigkeitspakets in Bezug auf 'XBIOC' könnte gelöst werden, wenn:
install.packages ("remotes") remotes :: install_github ("renozao/xbioc")Bitte beachten Sie die Seite der Vignetten.
Das SCDC -Papier wird bei Briefings in Bioinformatik veröffentlicht.
Fragen zum Paket können per E -Mail an: [email protected] gesendet werden
Wenn es im Einzelzell -Datensatz nur 'ein Thema/eine Person' gibt, verwenden Sie bitte SCDC_qc_ONE() , SCDC_prop_ONE() Funktionen.
Aspekte, die die Entfaltungsergebnisse beeinflussen könnten:
Datenformat: Sind Bulk- und Einzelzellproben beide Rohzahlen / gleiches Format? Wir erwarten, dass das Datenformat konsistent und vergleichbar ist.
Genfilterung: Haben Sie niedrig exprimierte Gene / ribosomale Gene / mitochondriale Gene herausgefiltert? Diese Gene können die nachgelagerte Analyse beeinflussen.
Zellgröße und Bibliotheksgröße Faktoren: Für eine einzelne Zelle könnte die Summe aller Genzahlen (die Bibliotheksgröße) ihre reale Zellgröße widerspiegeln? Dies ist eine unserer Annahmen: das Verhältnis der Bibliotheksgrößen zwischen Zelltypen kann das Verhältnis der realen Zellgrößen zwischen Zelltypen widerspiegeln. Wenn nicht, können Sie den Zellgrößenfaktor manuell eingeben, wenn Sie die "Basismatrix" erstellen.
Ähnliche Zelltypen: Gibt es Zelltypen, die die Analyse möglicherweise verwechseln könnten? Zum Beispiel Zelltypen mit sehr ähnlichen Profilen /Markergenen.
Fehlende wichtige Zelltypen / technische Probleme: Erwarten Sie, dass das Sequenzierungsverfahren einen großen Unterschied in der Masse und SC bewirkt, und auch die Technik ist gleich? Manchmal können Einzelzellreferenzdaten für einige Zelltypen Informationen verlieren. Zum Beispiel gibt es Fettzellen in Ihren Massenproben, aber irgendwie haben Sie sie nicht für einzelne Zelldaten.
Dekorvolution mit einem einzelnen Referenzdatensatz: Haben Sie versucht, einen Referenzdatensatz zu verwenden, um zu testen, ob die Ergebnisse im Allgemeinen sinnvoll sind? Ich sehe, du hast Bisque versucht. Haben Sie andere Methoden wie Cibersortx ausprobiert? Wenn Ergebnisse aus anderen "Ein-Referenz" -Delferenzmethoden sinnvoller sind, können Sie diese direkt mit unserem Ensemble-Schritt eingeben.