O SCDC é um método de deconvolução para RNA-seq a granel que aproveita expressões genéticas específicas do tipo célula de vários conjuntos de dados de referência de scRNA-seq . O SCDC adota um método de conjunto para integrar a deconvolução resulta de diferentes conjuntos de dados SCRNA-seq que são produzidos em diferentes laboratórios e em momentos diferentes, abordando implicitamente a confusão do efeito em lote.
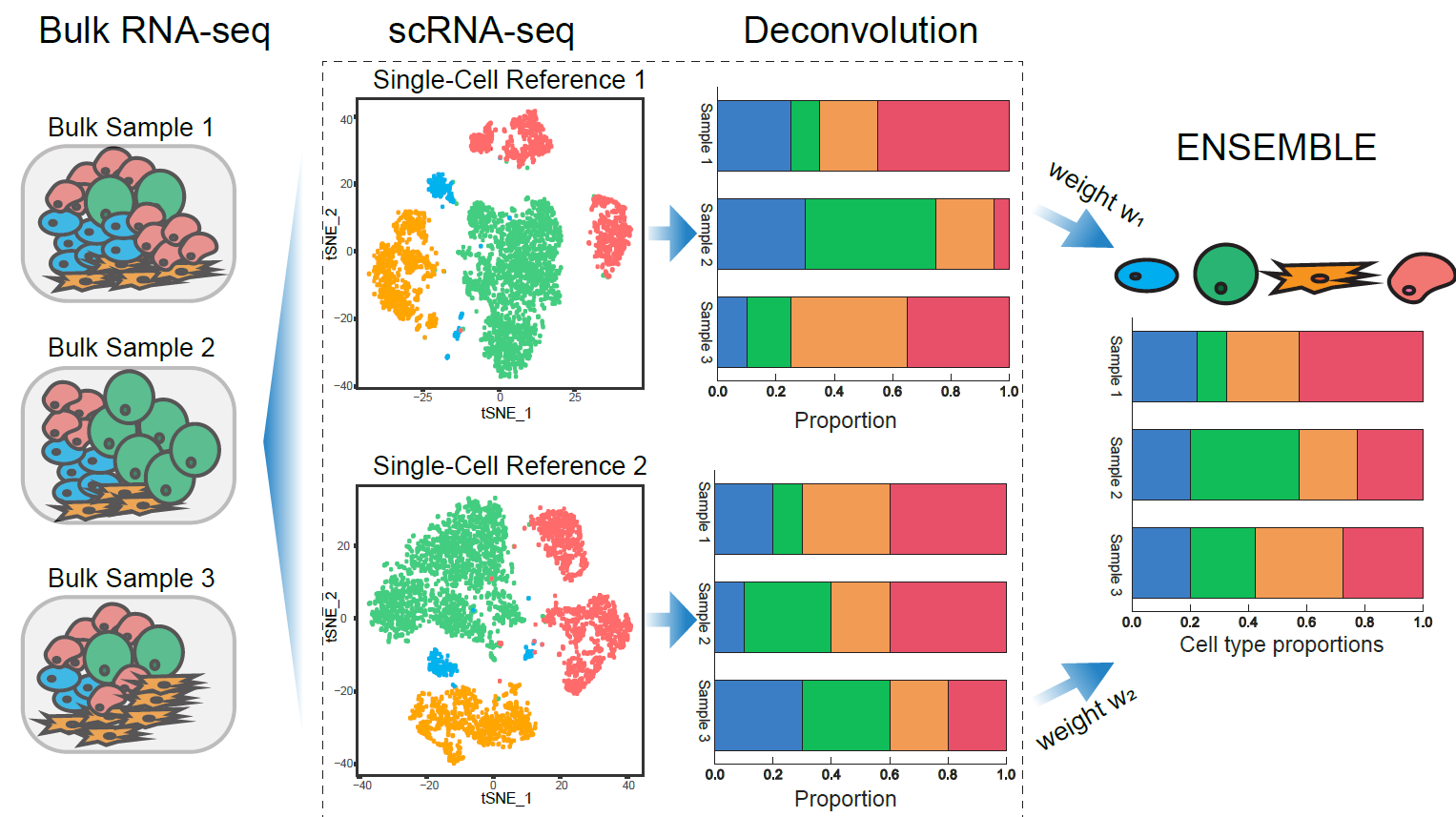
Meichen Dong, Aatish Thennavan, Eugene Urrutia, Yun Li, Charles M Perou, Fei Zou, Yuchao Jiang, SCDC: Deconvolução de expressão do gene a granel por várias referências de sequenciamento de RNA de célula única: // não: // não. org/10.1093/bib/bbz166
Licença: MIT
Você pode instalar a versão liberada do SCDC no GitHub com:
if (! requer ("devTools")) {
Install.packages ("Devtools")
} Devtools :: Install_github ("Meichendong/SCDC")O problema do pacote de dependência em relação ao 'xbioc' pode ser resolvido por:
Install.packages ("Remotes") Remotes :: Install_github ("Renozao/Xbioc")Por favor, veja a página Vignettes.
O artigo do SCDC é publicado no Briefings em Bioinformática.
Perguntas sobre o pacote podem ser enviadas por e -mail para: [email protected]
Quando houver apenas 'um sujeito/indivíduo' no conjunto de dados de células únicas, use funções SCDC_qc_ONE() , SCDC_prop_ONE() .
Aspectos que podem afetar os resultados da deconvolução:
Formato de dados: amostras de células a granel e únicas são contagens brutas / o mesmo formato? Esperamos que o formato de dados seja consistente e comparável.
Filtragem de genes: você filtrou genes de forma baixa / genes ribossômicos / genes mitocondriais? Esses genes podem afetar a análise a jusante.
Fatores do tamanho da célula e da biblioteca: para uma única célula, você acha que a soma de todas as contagens de genes (o tamanho da biblioteca) pode refletir seu tamanho real da célula? Essa é uma das nossas suposições: a proporção de tamanhos de biblioteca entre os tipos de células pode refletir a proporção de tamanhos de células reais entre os tipos de células. Caso contrário, você pode inserir manualmente o fator de tamanho da célula ao construir a "matriz base".
Tipos de células semelhantes: existem tipos de células que podem confundir a análise? Por exemplo, tipos de células que possuem perfis /genes marcadores muito semelhantes.
Faltando os principais tipos de células / problemas técnicos: você espera que o procedimento de seqüenciamento faça uma grande diferença no volume e o SC até a técnica é a mesma? Às vezes, os dados de referência de células únicas podem perder informações para alguns tipos de células. Por exemplo, existem células adiposas em suas amostras a granel, mas de alguma forma você não as possui para dados de células únicas.
Deconvolução usando um único conjunto de dados de referência: você tentou usar um conjunto de dados de referência para testar se os resultados fazem sentido em geral? Vejo que você tentou bisque. Você já tentou outros métodos como o Cibersortx? Se os resultados de outros métodos de desconvolução de "referência" fizer mais sentido, você poderá inseri-los diretamente usando nossa etapa de conjunto.