O repo contém:
Publicamos o DNABERT-S, um modelo de fundação baseado no DNABERT-2 projetado especificamente para gerar incorporação de DNA que se agrupa e segrega naturalmente o genoma de diferentes espécies no espaço de incorporação. Confira aqui se estiver interessado.
O DNABERT-2 é um modelo de fundação treinado em genoma de várias espécies em larga escala que atinge o desempenho de ponta no
Os modelos pré-treinados estão disponíveis no Huggingface como zhihan1996/DNABERT-2-117M . Link para HuggingFace ModelHub. Link para downloads diretos.
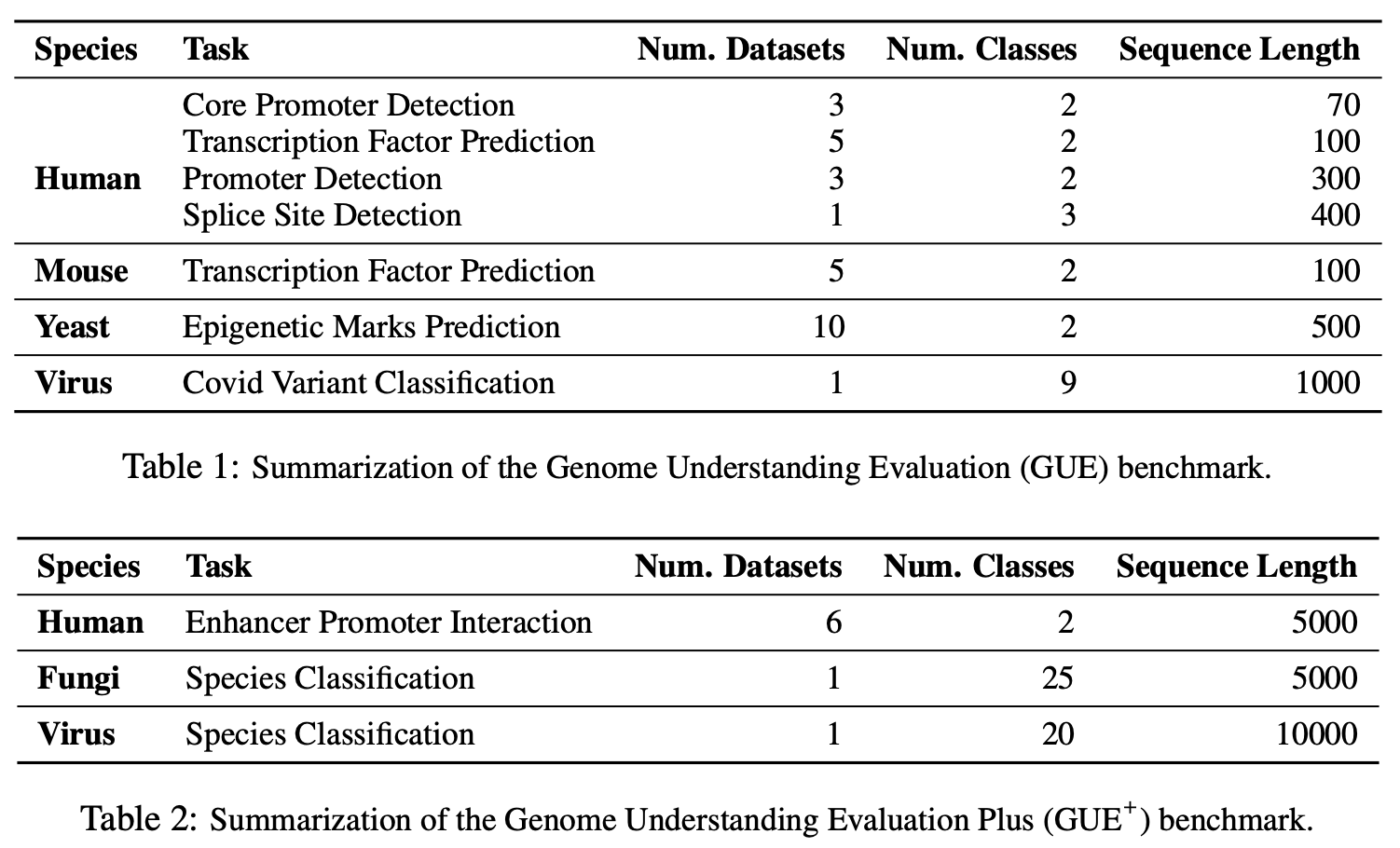
Gue é uma referência abrangente para a compreensão do genoma
# create and activate virtual python environment
conda create -n dna python=3.8
conda activate dna
# (optional if you would like to use flash attention)
# install triton from source
git clone https://github.com/openai/triton.git;
cd triton/python;
pip install cmake; # build-time dependency
pip install -e .
# install required packages
python3 -m pip install -r requirements.txt
Nosso modelo é fácil de usar com o pacote Transformers.
Para carregar o modelo de Huggingface (versão 4.28):
import torch
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True )
model = AutoModel . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True )Para carregar o modelo de Huggingface (versão> 4.28):
from transformers . models . bert . configuration_bert import BertConfig
config = BertConfig . from_pretrained ( "zhihan1996/DNABERT-2-117M" )
model = AutoModel . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True , config = config )Para calcular a incorporação de uma sequência de DNA
dna = "ACGTAGCATCGGATCTATCTATCGACACTTGGTTATCGATCTACGAGCATCTCGTTAGC"
inputs = tokenizer(dna, return_tensors = 'pt')["input_ids"]
hidden_states = model(inputs)[0] # [1, sequence_length, 768]
# embedding with mean pooling
embedding_mean = torch.mean(hidden_states[0], dim=0)
print(embedding_mean.shape) # expect to be 768
# embedding with max pooling
embedding_max = torch.max(hidden_states[0], dim=0)[0]
print(embedding_max.shape) # expect to be 768
Utilizamos e modificamos ligeiramente a implementação em Mosaicbert para dnabert-2 https://github.com/mosicml/examples/tree/main/examples/benchmarks/bert. Você deve poder replicar o treinamento do modelo seguindo as instruções.
Or you can use the run_mlm.py at https://github.com/huggingface/transformers/tree/main/examples/pytorch/language-modeling by importing the BertModelForMaskedLM from https://huggingface.co/zhihan1996/DNABERT-2-117M/blob/main/bert_layers.py. Deve produzir um modelo muito semelhante.
Os dados de treinamento estão disponíveis aqui.
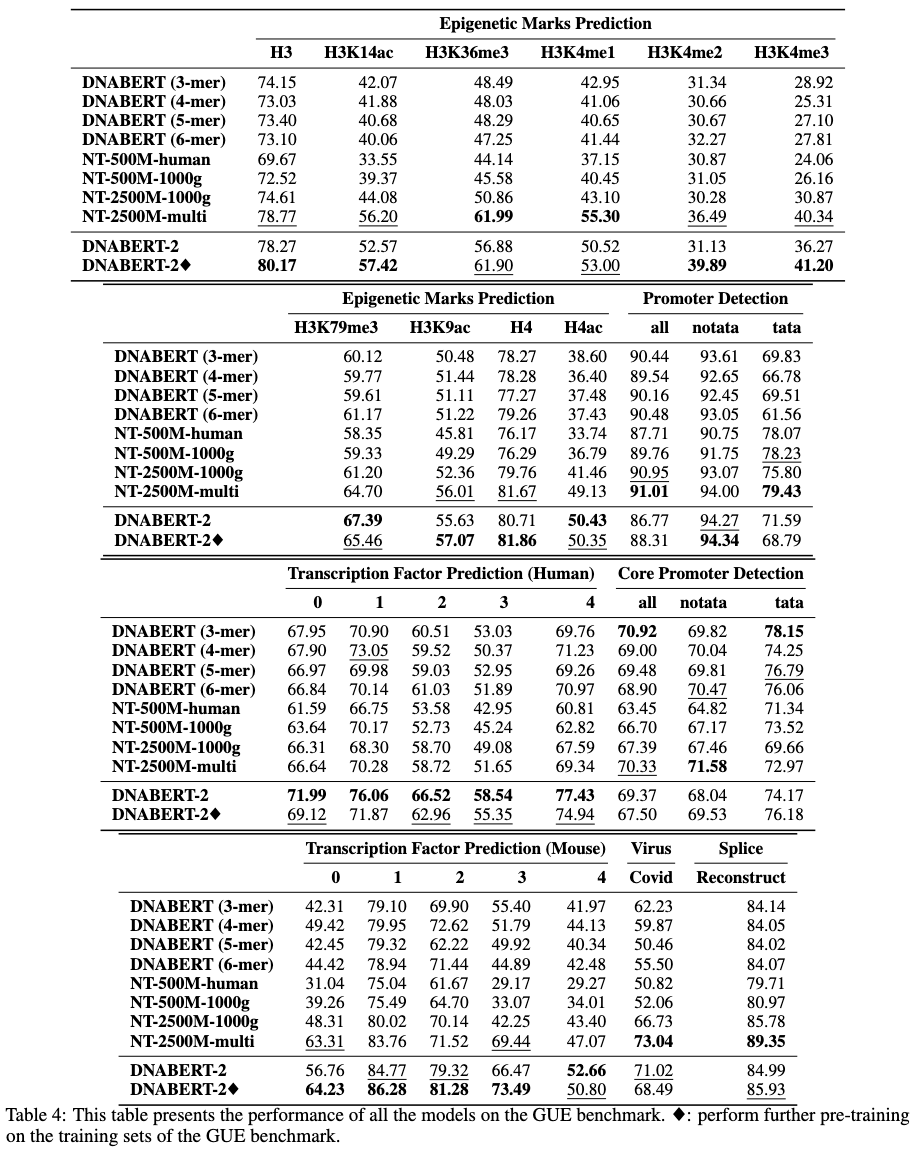
Por favor, faça o download do conjunto de dados Gue daqui. Em seguida, execute os scripts para avaliar todas as tarefas.
O script atual está definido para usar DataParallel para treinamento em 4 GPUs. Se você tiver um número diferente de GPUs, altere o per_device_train_batch_size e gradient_accumulation_steps de acordo para ajustar o tamanho do lote global para 32 para replicar os resultados no artigo. Se você deseja executar o treinamento multi-GPU distribuído (por exemplo, com DistributedDataParallel ), basta alterar python para torchrun --nproc_per_node ${n_gpu} .
export DATA_PATH=/path/to/GUE #(e.g., /home/user)
cd finetune
# Evaluate DNABERT-2 on GUE
sh scripts/run_dnabert2.sh DATA_PATH
# Evaluate DNABERT (e.g., DNABERT with 3-mer) on GUE
# 3 for 3-mer, 4 for 4-mer, 5 for 5-mer, 6 for 6-mer
sh scripts/run_dnabert1.sh DATA_PATH 3
# Evaluate Nucleotide Transformers on GUE
# 0 for 500m-1000g, 1 for 500m-human-ref, 2 for 2.5b-1000g, 3 for 2.5b-multi-species
sh scripts/run_nt.sh DATA_PATH 0
Aqui fornecemos um exemplo de DNABERT2 de ajuste fino em seus próprios conjuntos de dados.
Primeiro, gere 3 arquivos csv do seu conjunto de dados: train.csv , dev.csv e test.csv . No processo de treinamento, o modelo é treinado no train.csv e é avaliado no arquivo dev.csv . Após o treinamento, se terminar, o ponto de verificação com a menor perda no arquivo dev.csv é carregado e avaliado no test.csv . Se você não tiver um conjunto de validação, basta fazer o dev.csv e test.csv o mesmo.
Consulte a pasta sample_data para obter uma amostra de formato de dados. Cada arquivo deve estar no mesmo formato, com a primeira linha como o chefe de documentos chamado sequence, label . Cada linha a seguir deve conter uma sequência de DNA e um rótulo numérico concatenado por a , (por exemplo, ACGTCAGTCAGCGTACGT, 1 ).
Em seguida, você pode finalizar o DNABERT-2 no seu próprio conjunto de dados com o seguinte código:
cd finetune
export DATA_PATH=$path/to/data/folder # e.g., ./sample_data
export MAX_LENGTH=100 # Please set the number as 0.25 * your sequence length.
# e.g., set it as 250 if your DNA sequences have 1000 nucleotide bases
# This is because the tokenized will reduce the sequence length by about 5 times
export LR=3e-5
# Training use DataParallel
python train.py
--model_name_or_path zhihan1996/DNABERT-2-117M
--data_path ${DATA_PATH}
--kmer -1
--run_name DNABERT2_${DATA_PATH}
--model_max_length ${MAX_LENGTH}
--per_device_train_batch_size 8
--per_device_eval_batch_size 16
--gradient_accumulation_steps 1
--learning_rate ${LR}
--num_train_epochs 5
--fp16
--save_steps 200
--output_dir output/dnabert2
--evaluation_strategy steps
--eval_steps 200
--warmup_steps 50
--logging_steps 100
--overwrite_output_dir True
--log_level info
--find_unused_parameters False
# Training use DistributedDataParallel (more efficient)
export num_gpu=4 # please change the value based on your setup
torchrun --nproc_per_node=${num_gpu} train.py
--model_name_or_path zhihan1996/DNABERT-2-117M
--data_path ${DATA_PATH}
--kmer -1
--run_name DNABERT2_${DATA_PATH}
--model_max_length ${MAX_LENGTH}
--per_device_train_batch_size 8
--per_device_eval_batch_size 16
--gradient_accumulation_steps 1
--learning_rate ${LR}
--num_train_epochs 5
--fp16
--save_steps 200
--output_dir output/dnabert2
--evaluation_strategy steps
--eval_steps 200
--warmup_steps 50
--logging_steps 100
--overwrite_output_dir True
--log_level info
--find_unused_parameters False
Se você tiver alguma dúvida sobre nosso artigo ou códigos, sinta -se à vontade para iniciar um problema ou enviar um e -mail para Zhihan Zhou ([email protected]).
Se você usar o DNABERT-2 em seu trabalho, cite gentilmente nosso papel:
Dnabert-2
@misc{zhou2023dnabert2,
title={DNABERT-2: Efficient Foundation Model and Benchmark For Multi-Species Genome},
author={Zhihan Zhou and Yanrong Ji and Weijian Li and Pratik Dutta and Ramana Davuluri and Han Liu},
year={2023},
eprint={2306.15006},
archivePrefix={arXiv},
primaryClass={q-bio.GN}
}
Dnabert
@article{ji2021dnabert,
author = {Ji, Yanrong and Zhou, Zhihan and Liu, Han and Davuluri, Ramana V},
title = "{DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome}",
journal = {Bioinformatics},
volume = {37},
number = {15},
pages = {2112-2120},
year = {2021},
month = {02},
issn = {1367-4803},
doi = {10.1093/bioinformatics/btab083},
url = {https://doi.org/10.1093/bioinformatics/btab083},
eprint = {https://academic.oup.com/bioinformatics/article-pdf/37/15/2112/50578892/btab083.pdf},
}