Le repo contient:
Nous publions DNABERT-S, un modèle de fondation basé sur le DNABERT-2 spécialement conçu pour générer de l'intégration d'ADN qui grappe et sépare naturellement le génome de différentes espèces dans l'espace d'incorporation. Veuillez le vérifier ici si vous êtes intéressé.
DNABERT-2 est un modèle de fondation formé sur le génome multi-espèces à grande échelle qui atteint les performances de pointe sur
Les modèles pré-formés sont disponibles chez HuggingFace sous le nom de zhihan1996/DNABERT-2-117M . Lien vers HuggingFace ModelHub. Lien pour les téléchargements directs.
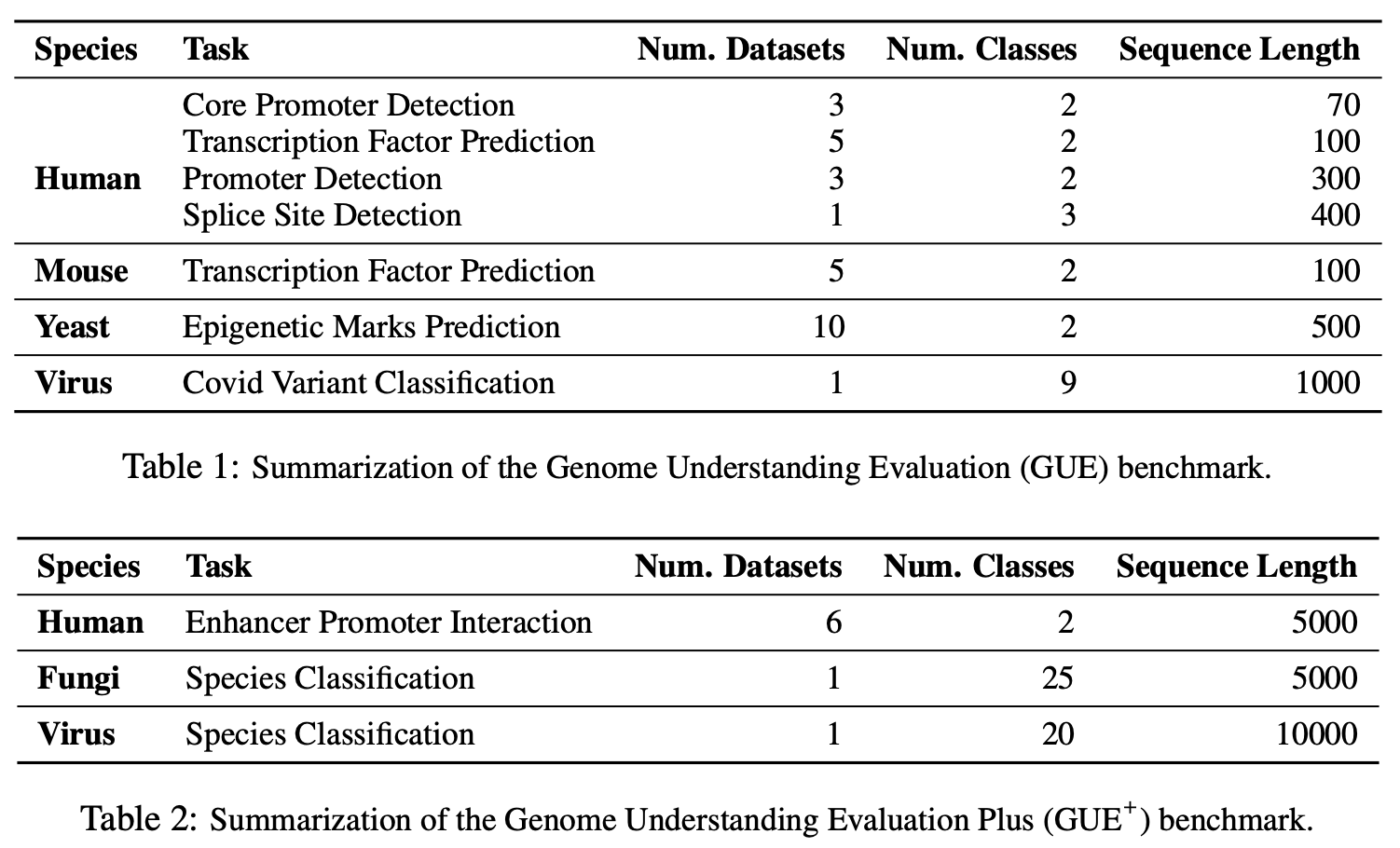
Gue est une référence complète pour la compréhension du génome
# create and activate virtual python environment
conda create -n dna python=3.8
conda activate dna
# (optional if you would like to use flash attention)
# install triton from source
git clone https://github.com/openai/triton.git;
cd triton/python;
pip install cmake; # build-time dependency
pip install -e .
# install required packages
python3 -m pip install -r requirements.txt
Notre modèle est facile à utiliser avec le package Transformers.
Pour charger le modèle de HuggingFace (version 4.28):
import torch
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True )
model = AutoModel . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True )Pour charger le modèle de HuggingFace (version> 4.28):
from transformers . models . bert . configuration_bert import BertConfig
config = BertConfig . from_pretrained ( "zhihan1996/DNABERT-2-117M" )
model = AutoModel . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True , config = config )Pour calculer l'incorporation d'une séquence d'ADN
dna = "ACGTAGCATCGGATCTATCTATCGACACTTGGTTATCGATCTACGAGCATCTCGTTAGC"
inputs = tokenizer(dna, return_tensors = 'pt')["input_ids"]
hidden_states = model(inputs)[0] # [1, sequence_length, 768]
# embedding with mean pooling
embedding_mean = torch.mean(hidden_states[0], dim=0)
print(embedding_mean.shape) # expect to be 768
# embedding with max pooling
embedding_max = torch.max(hidden_states[0], dim=0)[0]
print(embedding_max.shape) # expect to be 768
Nous avons utilisé et modifié légèrement l'implémentation de Mosaicbert pour dnabert-2 https://github.com/mosaicml/examples/tree/main/examples/benchmarks/bert. Vous devriez être en mesure de reproduire la formation du modèle suite aux instructions.
Ou vous pouvez utiliser le run_mlm.py sur https://github.com/huggingface/transformers/tree/main/examples/pytorch/language-modeling en important le bertmodelformasydlm à partir de https://huggingface. Il devrait produire un modèle très similaire.
Les données de formation sont disponibles ici.
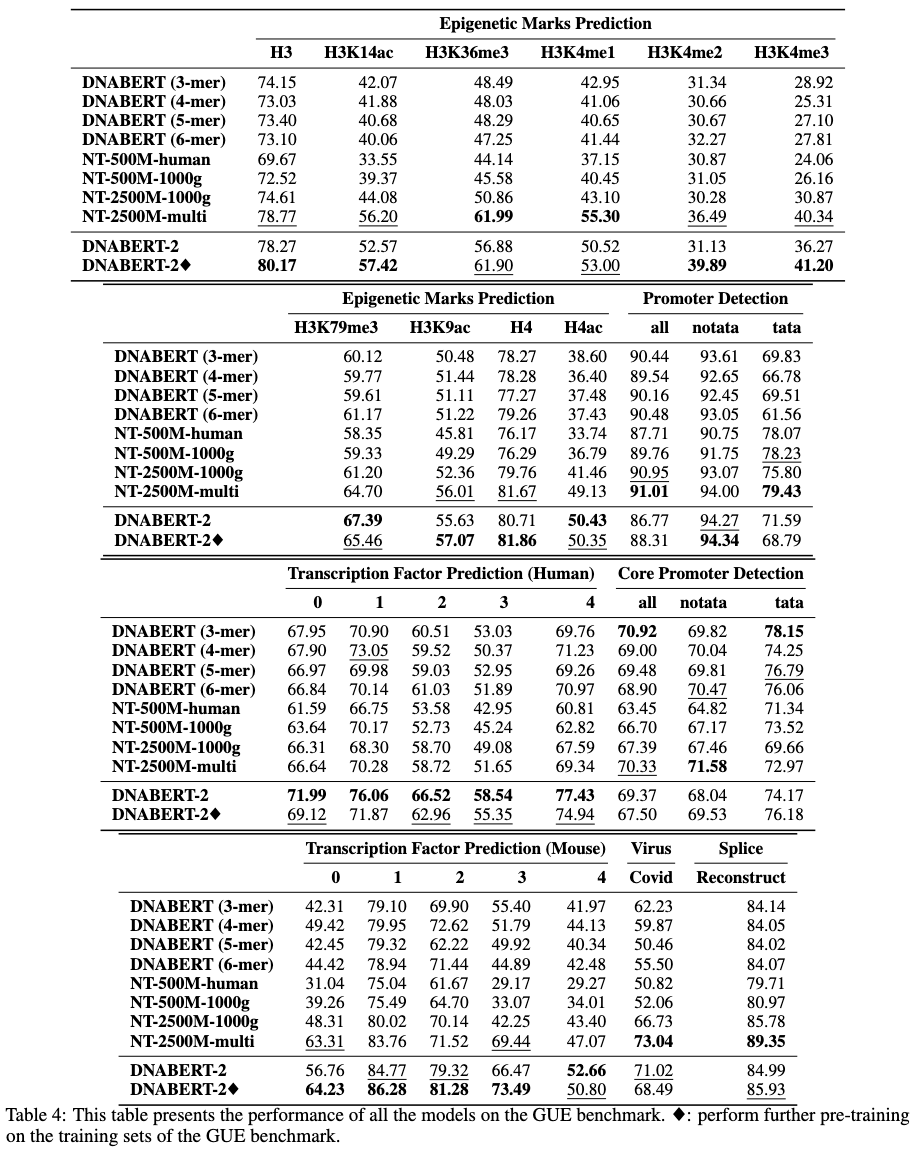
Veuillez d'abord télécharger l'ensemble de données Gue à partir d'ici. Exécutez ensuite les scripts pour évaluer toutes les tâches.
Le script actuel est défini pour utiliser DataParallel pour une formation sur 4 GPU. Si vous avez un nombre différent de GPU, veuillez modifier le per_device_train_batch_size et gradient_accumulation_steps en conséquence pour ajuster la taille globale du lot à 32 pour reproduire les résultats du papier. Si vous souhaitez effectuer une formation multi-GPU distribuée (par exemple, avec DistributedDataParallel ), changez simplement python en torchrun --nproc_per_node ${n_gpu} .
export DATA_PATH=/path/to/GUE #(e.g., /home/user)
cd finetune
# Evaluate DNABERT-2 on GUE
sh scripts/run_dnabert2.sh DATA_PATH
# Evaluate DNABERT (e.g., DNABERT with 3-mer) on GUE
# 3 for 3-mer, 4 for 4-mer, 5 for 5-mer, 6 for 6-mer
sh scripts/run_dnabert1.sh DATA_PATH 3
# Evaluate Nucleotide Transformers on GUE
# 0 for 500m-1000g, 1 for 500m-human-ref, 2 for 2.5b-1000g, 3 for 2.5b-multi-species
sh scripts/run_nt.sh DATA_PATH 0
Ici, nous fournissons un exemple de réglage fin DNABert2 sur vos propres ensembles de données.
Tout d'abord, veuillez générer 3 fichiers csv à partir de votre ensemble de données: train.csv , dev.csv et test.csv . Dans le processus de formation, le modèle est formé sur train.csv et est évalué sur le fichier dev.csv . Une fois la formation si elle est terminée, le point de contrôle avec la plus petite perte du fichier dev.csv est chargé et est évalué sur test.csv . Si vous n'avez pas de jeu de validation, veuillez simplement faire le dev.csv et test.csv .
Veuillez consulter le dossier sample_data pour un échantillon de format de données. Chaque fichier doit être dans le même format, avec la première ligne comme sequence, label . Chaque ligne suivante doit contenir une séquence d'ADN et une étiquette numérique concaténée par A , (par exemple, ACGTCAGTCAGCGTACGT, 1 ).
Ensuite, vous pouvez finertune dnabert-2 sur votre propre ensemble de données avec le code suivant:
cd finetune
export DATA_PATH=$path/to/data/folder # e.g., ./sample_data
export MAX_LENGTH=100 # Please set the number as 0.25 * your sequence length.
# e.g., set it as 250 if your DNA sequences have 1000 nucleotide bases
# This is because the tokenized will reduce the sequence length by about 5 times
export LR=3e-5
# Training use DataParallel
python train.py
--model_name_or_path zhihan1996/DNABERT-2-117M
--data_path ${DATA_PATH}
--kmer -1
--run_name DNABERT2_${DATA_PATH}
--model_max_length ${MAX_LENGTH}
--per_device_train_batch_size 8
--per_device_eval_batch_size 16
--gradient_accumulation_steps 1
--learning_rate ${LR}
--num_train_epochs 5
--fp16
--save_steps 200
--output_dir output/dnabert2
--evaluation_strategy steps
--eval_steps 200
--warmup_steps 50
--logging_steps 100
--overwrite_output_dir True
--log_level info
--find_unused_parameters False
# Training use DistributedDataParallel (more efficient)
export num_gpu=4 # please change the value based on your setup
torchrun --nproc_per_node=${num_gpu} train.py
--model_name_or_path zhihan1996/DNABERT-2-117M
--data_path ${DATA_PATH}
--kmer -1
--run_name DNABERT2_${DATA_PATH}
--model_max_length ${MAX_LENGTH}
--per_device_train_batch_size 8
--per_device_eval_batch_size 16
--gradient_accumulation_steps 1
--learning_rate ${LR}
--num_train_epochs 5
--fp16
--save_steps 200
--output_dir output/dnabert2
--evaluation_strategy steps
--eval_steps 200
--warmup_steps 50
--logging_steps 100
--overwrite_output_dir True
--log_level info
--find_unused_parameters False
Si vous avez une question concernant notre article ou nos codes, n'hésitez pas à démarrer un problème ou à envoyer un courriel à Zhihan Zhou ([email protected]).
Si vous utilisez DNABERT-2 dans votre travail, veuillez citer notre papier:
Dnabert-2
@misc{zhou2023dnabert2,
title={DNABERT-2: Efficient Foundation Model and Benchmark For Multi-Species Genome},
author={Zhihan Zhou and Yanrong Ji and Weijian Li and Pratik Dutta and Ramana Davuluri and Han Liu},
year={2023},
eprint={2306.15006},
archivePrefix={arXiv},
primaryClass={q-bio.GN}
}
Dnabert
@article{ji2021dnabert,
author = {Ji, Yanrong and Zhou, Zhihan and Liu, Han and Davuluri, Ramana V},
title = "{DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome}",
journal = {Bioinformatics},
volume = {37},
number = {15},
pages = {2112-2120},
year = {2021},
month = {02},
issn = {1367-4803},
doi = {10.1093/bioinformatics/btab083},
url = {https://doi.org/10.1093/bioinformatics/btab083},
eprint = {https://academic.oup.com/bioinformatics/article-pdf/37/15/2112/50578892/btab083.pdf},
}