Das Repo enthält:
Wir veröffentlichen DNABERT-S, ein Fundamentmodell, das auf DNABERT-2 basiert, das speziell für die Erzeugung von DNA-Einbettung entwickelt wurde und das das Genom verschiedener Arten im Einbettungsraum natürlich Cluster und Trennung von DNOMEMODEN ist. Bitte überprüfen Sie es hier, wenn Sie interessiert sind.
DNABERT-2 ist ein Foundation-Modell, das auf großem Maßstab ausgebildet ist, das die hochmoderne Leistung erreicht
Die vorgebauten Modelle sind bei Huggingface als zhihan1996/DNABERT-2-117M erhältlich. Link zum Umarmungsface -ModellHub. Link für direkte Downloads.
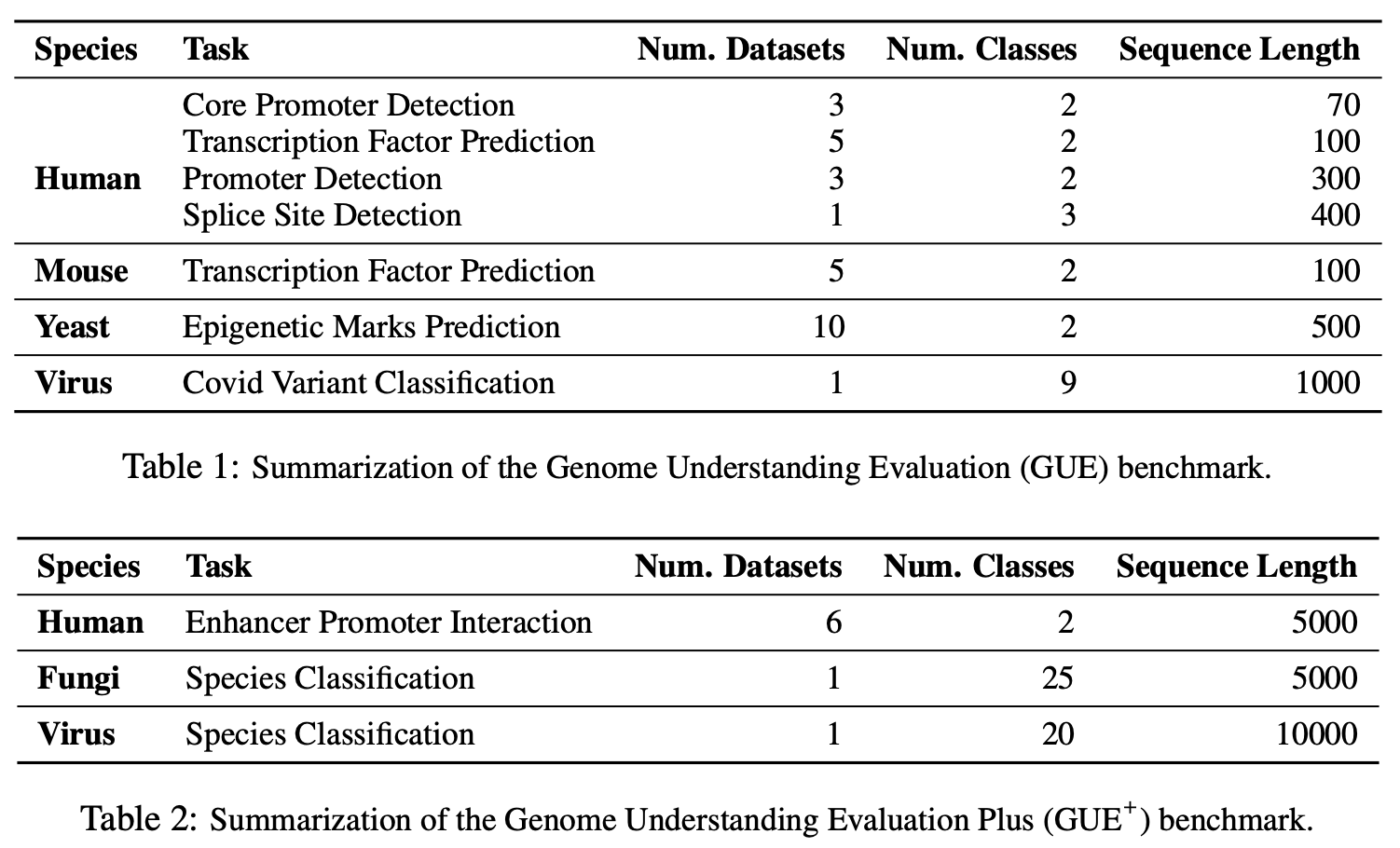
Gue ist ein umfassender Benchmark für das Genomverständnis
# create and activate virtual python environment
conda create -n dna python=3.8
conda activate dna
# (optional if you would like to use flash attention)
# install triton from source
git clone https://github.com/openai/triton.git;
cd triton/python;
pip install cmake; # build-time dependency
pip install -e .
# install required packages
python3 -m pip install -r requirements.txt
Unser Modell ist einfach mit dem Transformers -Paket zu verwenden.
So laden Sie das Modell von Suggingface (Version 4.28):
import torch
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True )
model = AutoModel . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True )So laden Sie das Modell von Suggingface (Version> 4.28):
from transformers . models . bert . configuration_bert import BertConfig
config = BertConfig . from_pretrained ( "zhihan1996/DNABERT-2-117M" )
model = AutoModel . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True , config = config )Um die Einbettung einer DNA -Sequenz zu berechnen
dna = "ACGTAGCATCGGATCTATCTATCGACACTTGGTTATCGATCTACGAGCATCTCGTTAGC"
inputs = tokenizer(dna, return_tensors = 'pt')["input_ids"]
hidden_states = model(inputs)[0] # [1, sequence_length, 768]
# embedding with mean pooling
embedding_mean = torch.mean(hidden_states[0], dim=0)
print(embedding_mean.shape) # expect to be 768
# embedding with max pooling
embedding_max = torch.max(hidden_states[0], dim=0)[0]
print(embedding_max.shape) # expect to be 768
Wir haben die Mosaicbert-Implementierung für dnabert-2 https://github.com/mosaicml/examples/tree/main/examples/benchmarks/bert leicht modifiziert. Sie sollten in der Lage sein, das Modelltraining nach den Anweisungen zu replizieren.
Oder Sie können die run_mlm.py unter https://github.com/huggingface/transformers/tree/main/examples/pytorch/glanguage-modeling verwenden https://huggingface.co/zhihan1996/dnabert-2-117m/blob/main/bert_layers.py. Es sollte ein sehr ähnliches Modell erzeugen.
Die Trainingsdaten sind hier verfügbar.
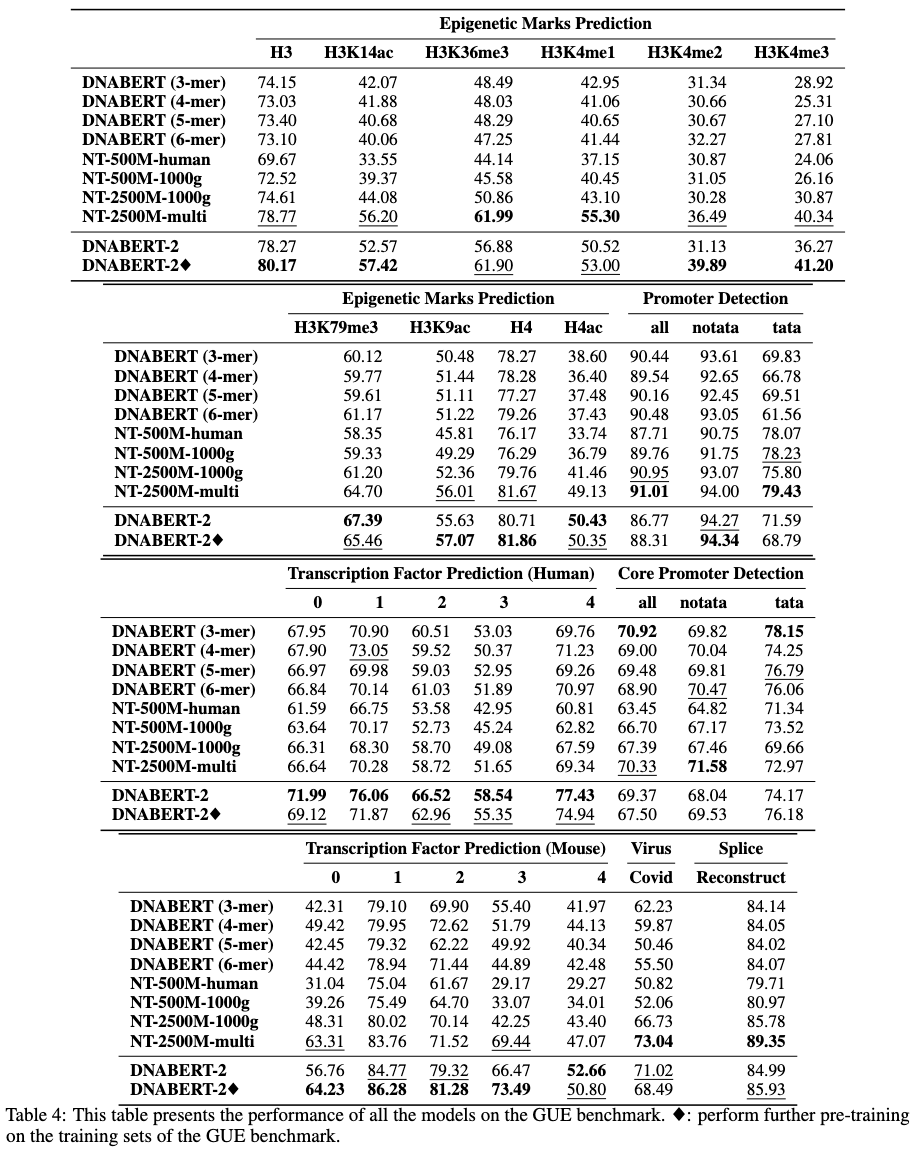
Bitte laden Sie zuerst den Gue -Datensatz von hier herunter. Führen Sie dann die Skripte aus, um alle Aufgaben zu bewerten.
Das aktuelle Skript wird so eingestellt, dass DataParallel für das Training auf 4 GPUs verwendet wird. Wenn Sie eine unterschiedliche Anzahl von GPUs haben, ändern Sie bitte die per_device_train_batch_size und gradient_accumulation_steps entsprechend, um die globale Stapelgröße auf 32 anzupassen, um die Ergebnisse im Papier zu replizieren. Wenn Sie ein verteiltes Multi-GPU-Training (z. B. mit DistributedDataParallel ) durchführen möchten, ändern Sie einfach python in torchrun --nproc_per_node ${n_gpu} .
export DATA_PATH=/path/to/GUE #(e.g., /home/user)
cd finetune
# Evaluate DNABERT-2 on GUE
sh scripts/run_dnabert2.sh DATA_PATH
# Evaluate DNABERT (e.g., DNABERT with 3-mer) on GUE
# 3 for 3-mer, 4 for 4-mer, 5 for 5-mer, 6 for 6-mer
sh scripts/run_dnabert1.sh DATA_PATH 3
# Evaluate Nucleotide Transformers on GUE
# 0 for 500m-1000g, 1 for 500m-human-ref, 2 for 2.5b-1000g, 3 for 2.5b-multi-species
sh scripts/run_nt.sh DATA_PATH 0
Hier bieten wir ein Beispiel für die Feinabstimmung DNABERT2 auf Ihren eigenen Datensätzen.
Erstellen Sie zunächst 3 csv -Dateien aus Ihrem Datensatz: train.csv , dev.csv und test.csv . Im Trainingsprozess wird das Modell im train.csv geschult und in der dev.csv -Datei bewertet. Nach dem Training wird der Kontrollpunkt mit dem kleinsten Verlust in der dev.csv -Datei geladen und auf test.csv ausgewertet. Wenn Sie keinen Validierungssatz haben, machen Sie bitte einfach den dev.csv und test.csv . CSV.
Weitere Informationen zum Datenformat finden Sie im Ordner sample_data . Jede Datei sollte im selben Format sein, wobei die erste Zeile als Dokumentkopf mit dem Namen sequence, label . Jede folgende Zeile sollte eine DNA -Sequenz und eine durch A verkettete numerische Markierung enthalten , z. B. ACGTCAGTCAGCGTACGT, 1 ).
Anschließend können Sie DNABERT-2 in Ihrem eigenen Datensatz mit dem folgenden Code beenden:
cd finetune
export DATA_PATH=$path/to/data/folder # e.g., ./sample_data
export MAX_LENGTH=100 # Please set the number as 0.25 * your sequence length.
# e.g., set it as 250 if your DNA sequences have 1000 nucleotide bases
# This is because the tokenized will reduce the sequence length by about 5 times
export LR=3e-5
# Training use DataParallel
python train.py
--model_name_or_path zhihan1996/DNABERT-2-117M
--data_path ${DATA_PATH}
--kmer -1
--run_name DNABERT2_${DATA_PATH}
--model_max_length ${MAX_LENGTH}
--per_device_train_batch_size 8
--per_device_eval_batch_size 16
--gradient_accumulation_steps 1
--learning_rate ${LR}
--num_train_epochs 5
--fp16
--save_steps 200
--output_dir output/dnabert2
--evaluation_strategy steps
--eval_steps 200
--warmup_steps 50
--logging_steps 100
--overwrite_output_dir True
--log_level info
--find_unused_parameters False
# Training use DistributedDataParallel (more efficient)
export num_gpu=4 # please change the value based on your setup
torchrun --nproc_per_node=${num_gpu} train.py
--model_name_or_path zhihan1996/DNABERT-2-117M
--data_path ${DATA_PATH}
--kmer -1
--run_name DNABERT2_${DATA_PATH}
--model_max_length ${MAX_LENGTH}
--per_device_train_batch_size 8
--per_device_eval_batch_size 16
--gradient_accumulation_steps 1
--learning_rate ${LR}
--num_train_epochs 5
--fp16
--save_steps 200
--output_dir output/dnabert2
--evaluation_strategy steps
--eval_steps 200
--warmup_steps 50
--logging_steps 100
--overwrite_output_dir True
--log_level info
--find_unused_parameters False
Wenn Sie Fragen zu unserem Papier oder unserer Codes haben, können Sie sich gerne mit einem Problem anfangen oder Zhihan Zhou ([email protected]) senden.
Wenn Sie Dnabert-2 in Ihrer Arbeit verwenden, zitieren Sie bitte unsere Zeitung:
Dnabert-2
@misc{zhou2023dnabert2,
title={DNABERT-2: Efficient Foundation Model and Benchmark For Multi-Species Genome},
author={Zhihan Zhou and Yanrong Ji and Weijian Li and Pratik Dutta and Ramana Davuluri and Han Liu},
year={2023},
eprint={2306.15006},
archivePrefix={arXiv},
primaryClass={q-bio.GN}
}
Dnabert
@article{ji2021dnabert,
author = {Ji, Yanrong and Zhou, Zhihan and Liu, Han and Davuluri, Ramana V},
title = "{DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome}",
journal = {Bioinformatics},
volume = {37},
number = {15},
pages = {2112-2120},
year = {2021},
month = {02},
issn = {1367-4803},
doi = {10.1093/bioinformatics/btab083},
url = {https://doi.org/10.1093/bioinformatics/btab083},
eprint = {https://academic.oup.com/bioinformatics/article-pdf/37/15/2112/50578892/btab083.pdf},
}