Repo berisi:
Kami menerbitkan DNABERT-S, model fondasi yang didasarkan pada DNABERT-2 yang dirancang khusus untuk menghasilkan embedding DNA yang secara alami kelompok dan memisahkan genom spesies yang berbeda dalam ruang embedding. Silakan periksa di sini jika Anda tertarik.
DNABERT-2 adalah model fondasi yang dilatih pada genom multi-spesies skala besar yang mencapai kinerja canggih
Model pra-terlatih tersedia di Huggingface sebagai zhihan1996/DNABERT-2-117M . Tautan ke HuggingFace ModelHub. Tautan untuk unduhan langsung.
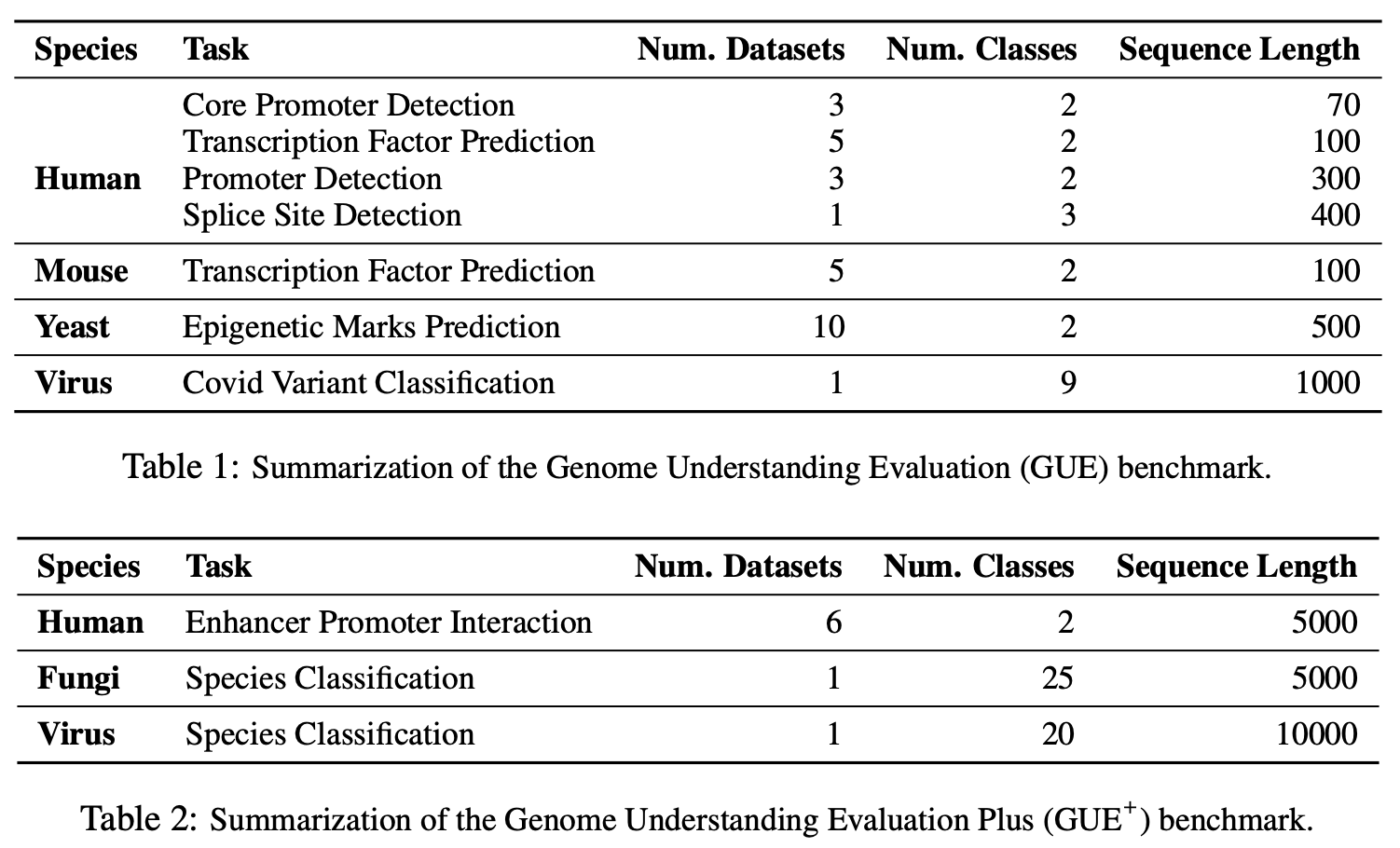
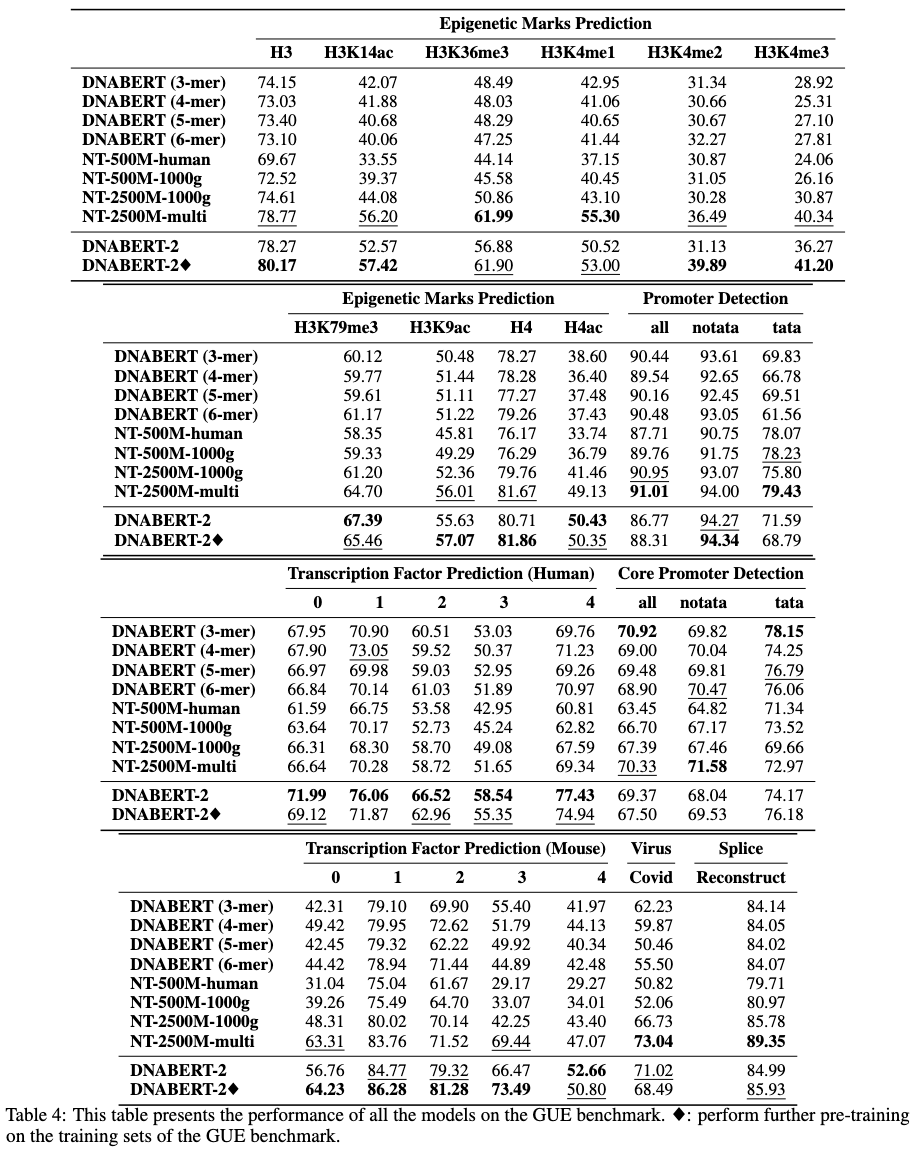
GUE adalah tolok ukur komprehensif untuk pemahaman genom yang disebutkan
# create and activate virtual python environment
conda create -n dna python=3.8
conda activate dna
# (optional if you would like to use flash attention)
# install triton from source
git clone https://github.com/openai/triton.git;
cd triton/python;
pip install cmake; # build-time dependency
pip install -e .
# install required packages
python3 -m pip install -r requirements.txt
Model kami mudah digunakan dengan paket Transformers.
Untuk memuat model dari HuggingFace (versi 4.28):
import torch
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True )
model = AutoModel . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True )Untuk memuat model dari HuggingFace (versi> 4.28):
from transformers . models . bert . configuration_bert import BertConfig
config = BertConfig . from_pretrained ( "zhihan1996/DNABERT-2-117M" )
model = AutoModel . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True , config = config )Untuk menghitung embedding urutan DNA
dna = "ACGTAGCATCGGATCTATCTATCGACACTTGGTTATCGATCTACGAGCATCTCGTTAGC"
inputs = tokenizer(dna, return_tensors = 'pt')["input_ids"]
hidden_states = model(inputs)[0] # [1, sequence_length, 768]
# embedding with mean pooling
embedding_mean = torch.mean(hidden_states[0], dim=0)
print(embedding_mean.shape) # expect to be 768
# embedding with max pooling
embedding_max = torch.max(hidden_states[0], dim=0)[0]
print(embedding_max.shape) # expect to be 768
Kami menggunakan dan sedikit memodifikasi implementasi Mosaicbert untuk dnabert-2 https://github.com/mosaicml/examples/tree/main/examples/benchmarks/bert. Anda harus dapat meniru pelatihan model mengikuti instruksi.
Atau Anda dapat menggunakan run_mlm.py di https://github.com/huggingface/transformers/tree/main/examples/pytorch/language-modeling dengan mengimpor BertModelFormaskedlm dari https://huggingface.co/zhihan1996/dnabert-2-117m/blob/main/bert_layers.py. Itu harus menghasilkan model yang sangat mirip.
Data pelatihan tersedia di sini.
Pertama -tama unduh dataset GUE dari sini. Kemudian jalankan skrip untuk mengevaluasi semua tugas.
Skrip saat ini diatur untuk menggunakan DataParallel untuk pelatihan pada 4 GPU. Jika Anda memiliki jumlah GPU yang berbeda, harap ubah per_device_train_batch_size dan gradient_accumulation_steps sesuai untuk menyesuaikan ukuran batch global menjadi 32 untuk mereplikasi hasil dalam kertas. Jika Anda ingin melakukan pelatihan multi-GPU terdistribusi (misalnya, dengan DistributedDataParallel ), cukup ubah python menjadi torchrun --nproc_per_node ${n_gpu} .
export DATA_PATH=/path/to/GUE #(e.g., /home/user)
cd finetune
# Evaluate DNABERT-2 on GUE
sh scripts/run_dnabert2.sh DATA_PATH
# Evaluate DNABERT (e.g., DNABERT with 3-mer) on GUE
# 3 for 3-mer, 4 for 4-mer, 5 for 5-mer, 6 for 6-mer
sh scripts/run_dnabert1.sh DATA_PATH 3
# Evaluate Nucleotide Transformers on GUE
# 0 for 500m-1000g, 1 for 500m-human-ref, 2 for 2.5b-1000g, 3 for 2.5b-multi-species
sh scripts/run_nt.sh DATA_PATH 0
Di sini kami memberikan contoh fine-tuning DNABERT2 pada set data Anda sendiri.
Pertama, harap hasilkan 3 file csv dari dataset Anda: train.csv , dev.csv , dan test.csv . Dalam proses pelatihan, model dilatih di train.csv dan dievaluasi pada file dev.csv . Setelah pelatihan jika selesai, pos pemeriksaan dengan kerugian terkecil pada file dev.csv dimuat dan dievaluasi pada test.csv . Jika Anda tidak memiliki set validasi, harap buat saja dev.csv dan test.csv sama.
Silakan lihat folder sample_data untuk sampel format data. Setiap file harus dalam format yang sama, dengan baris pertama sebagai kepala dokumen bernama sequence, label . Setiap baris berikut harus berisi urutan DNA dan label numerik yang disatukan oleh A , (misalnya, ACGTCAGTCAGCGTACGT, 1 ).
Kemudian, Anda dapat finetune DNABERT-2 pada dataset Anda sendiri dengan kode berikut:
cd finetune
export DATA_PATH=$path/to/data/folder # e.g., ./sample_data
export MAX_LENGTH=100 # Please set the number as 0.25 * your sequence length.
# e.g., set it as 250 if your DNA sequences have 1000 nucleotide bases
# This is because the tokenized will reduce the sequence length by about 5 times
export LR=3e-5
# Training use DataParallel
python train.py
--model_name_or_path zhihan1996/DNABERT-2-117M
--data_path ${DATA_PATH}
--kmer -1
--run_name DNABERT2_${DATA_PATH}
--model_max_length ${MAX_LENGTH}
--per_device_train_batch_size 8
--per_device_eval_batch_size 16
--gradient_accumulation_steps 1
--learning_rate ${LR}
--num_train_epochs 5
--fp16
--save_steps 200
--output_dir output/dnabert2
--evaluation_strategy steps
--eval_steps 200
--warmup_steps 50
--logging_steps 100
--overwrite_output_dir True
--log_level info
--find_unused_parameters False
# Training use DistributedDataParallel (more efficient)
export num_gpu=4 # please change the value based on your setup
torchrun --nproc_per_node=${num_gpu} train.py
--model_name_or_path zhihan1996/DNABERT-2-117M
--data_path ${DATA_PATH}
--kmer -1
--run_name DNABERT2_${DATA_PATH}
--model_max_length ${MAX_LENGTH}
--per_device_train_batch_size 8
--per_device_eval_batch_size 16
--gradient_accumulation_steps 1
--learning_rate ${LR}
--num_train_epochs 5
--fp16
--save_steps 200
--output_dir output/dnabert2
--evaluation_strategy steps
--eval_steps 200
--warmup_steps 50
--logging_steps 100
--overwrite_output_dir True
--log_level info
--find_unused_parameters False
Jika Anda memiliki pertanyaan tentang makalah atau kode kami, jangan ragu untuk memulai masalah atau email Zhihan Zhou ([email protected]).
Jika Anda menggunakan DNABERT-2 dalam pekerjaan Anda, silakan kutip kertas kami:
DNABERT-2
@misc{zhou2023dnabert2,
title={DNABERT-2: Efficient Foundation Model and Benchmark For Multi-Species Genome},
author={Zhihan Zhou and Yanrong Ji and Weijian Li and Pratik Dutta and Ramana Davuluri and Han Liu},
year={2023},
eprint={2306.15006},
archivePrefix={arXiv},
primaryClass={q-bio.GN}
}
Dnabert
@article{ji2021dnabert,
author = {Ji, Yanrong and Zhou, Zhihan and Liu, Han and Davuluri, Ramana V},
title = "{DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome}",
journal = {Bioinformatics},
volume = {37},
number = {15},
pages = {2112-2120},
year = {2021},
month = {02},
issn = {1367-4803},
doi = {10.1093/bioinformatics/btab083},
url = {https://doi.org/10.1093/bioinformatics/btab083},
eprint = {https://academic.oup.com/bioinformatics/article-pdf/37/15/2112/50578892/btab083.pdf},
}