El repositorio contiene:
Publicamos DNABERT-S, un modelo de base basado en DNABERT-2 diseñado específicamente para generar incrustación de ADN que se agrupa y segregue naturalmente el genoma de diferentes especies en el espacio de incrustación. Por favor, échale un vistazo aquí si estás interesado.
Dnabert-2 es un modelo de fundación entrenado en el genoma de múltiples especies a gran escala que logra el rendimiento de vanguardia en
Los modelos previamente capacitados están disponibles en Huggingface como zhihan1996/DNABERT-2-117M . Enlace a Huggingface ModelHub. Enlace para descargas directas.
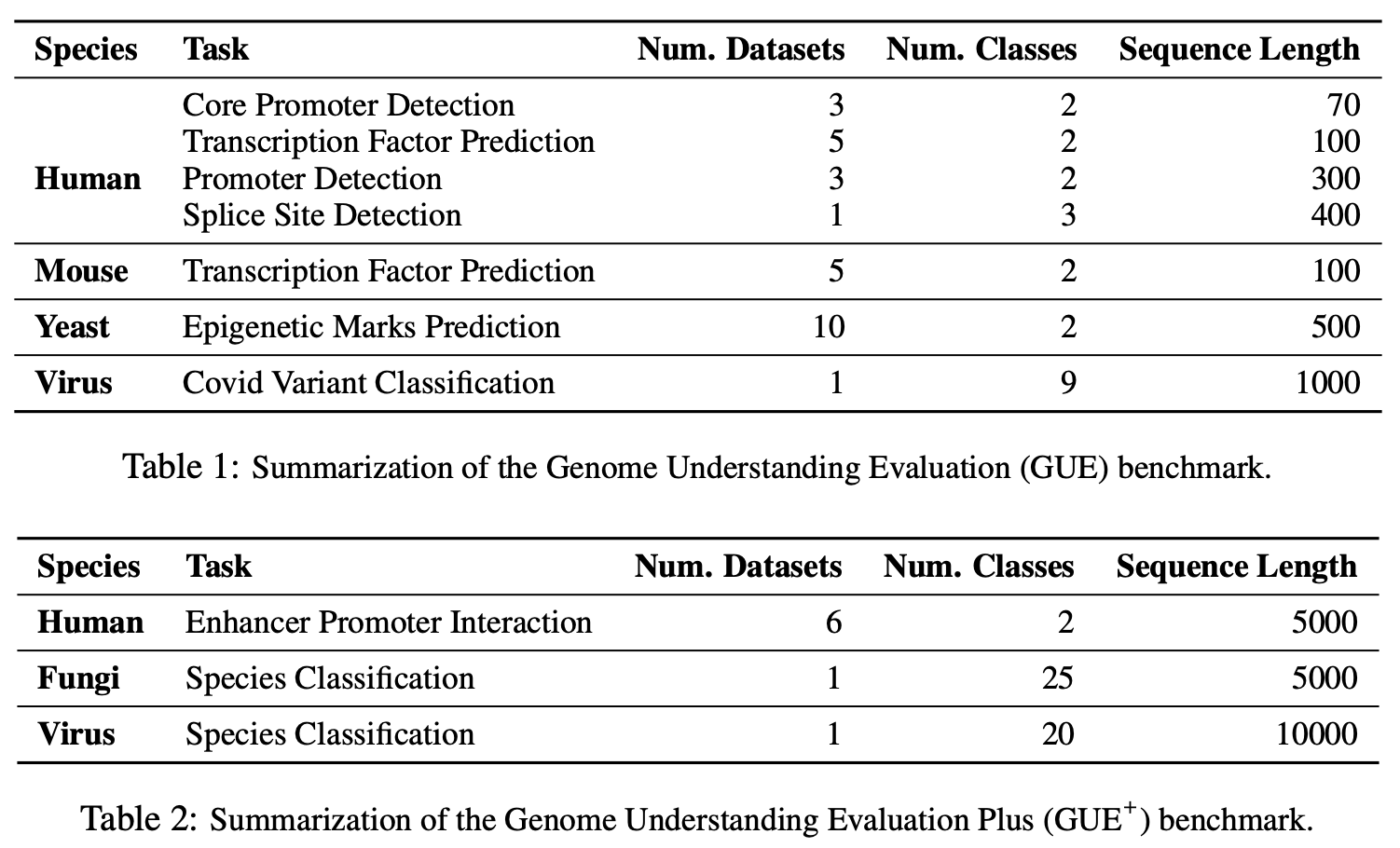
Gue es un punto de referencia integral para la comprensión del genoma que considera
# create and activate virtual python environment
conda create -n dna python=3.8
conda activate dna
# (optional if you would like to use flash attention)
# install triton from source
git clone https://github.com/openai/triton.git;
cd triton/python;
pip install cmake; # build-time dependency
pip install -e .
# install required packages
python3 -m pip install -r requirements.txt
Nuestro modelo es fácil de usar con el paquete Transformers.
Para cargar el modelo de Huggingface (versión 4.28):
import torch
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True )
model = AutoModel . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True )Para cargar el modelo de Huggingface (versión> 4.28):
from transformers . models . bert . configuration_bert import BertConfig
config = BertConfig . from_pretrained ( "zhihan1996/DNABERT-2-117M" )
model = AutoModel . from_pretrained ( "zhihan1996/DNABERT-2-117M" , trust_remote_code = True , config = config )Para calcular la incrustación de una secuencia de ADN
dna = "ACGTAGCATCGGATCTATCTATCGACACTTGGTTATCGATCTACGAGCATCTCGTTAGC"
inputs = tokenizer(dna, return_tensors = 'pt')["input_ids"]
hidden_states = model(inputs)[0] # [1, sequence_length, 768]
# embedding with mean pooling
embedding_mean = torch.mean(hidden_states[0], dim=0)
print(embedding_mean.shape) # expect to be 768
# embedding with max pooling
embedding_max = torch.max(hidden_states[0], dim=0)[0]
print(embedding_max.shape) # expect to be 768
Utilizamos y modificamos ligeramente la implementación de Mosaicbert para dnabert-2 https://github.com/mosaicml/examples/tree/main/examples/benchmarks/bert. Debería poder replicar la capacitación del modelo después de las instrucciones.
O puede usar run_mlm.py en https://github.com/huggingface/transformers/tree/main/examples/pytorch/language-modeling importando el BertModeformaskedlm de https://huggingface.co/zhihan1996/dnabert-2-117m/blob/main/bert_layers.py. Debería producir un modelo muy similar.
Los datos de capacitación están disponibles aquí.
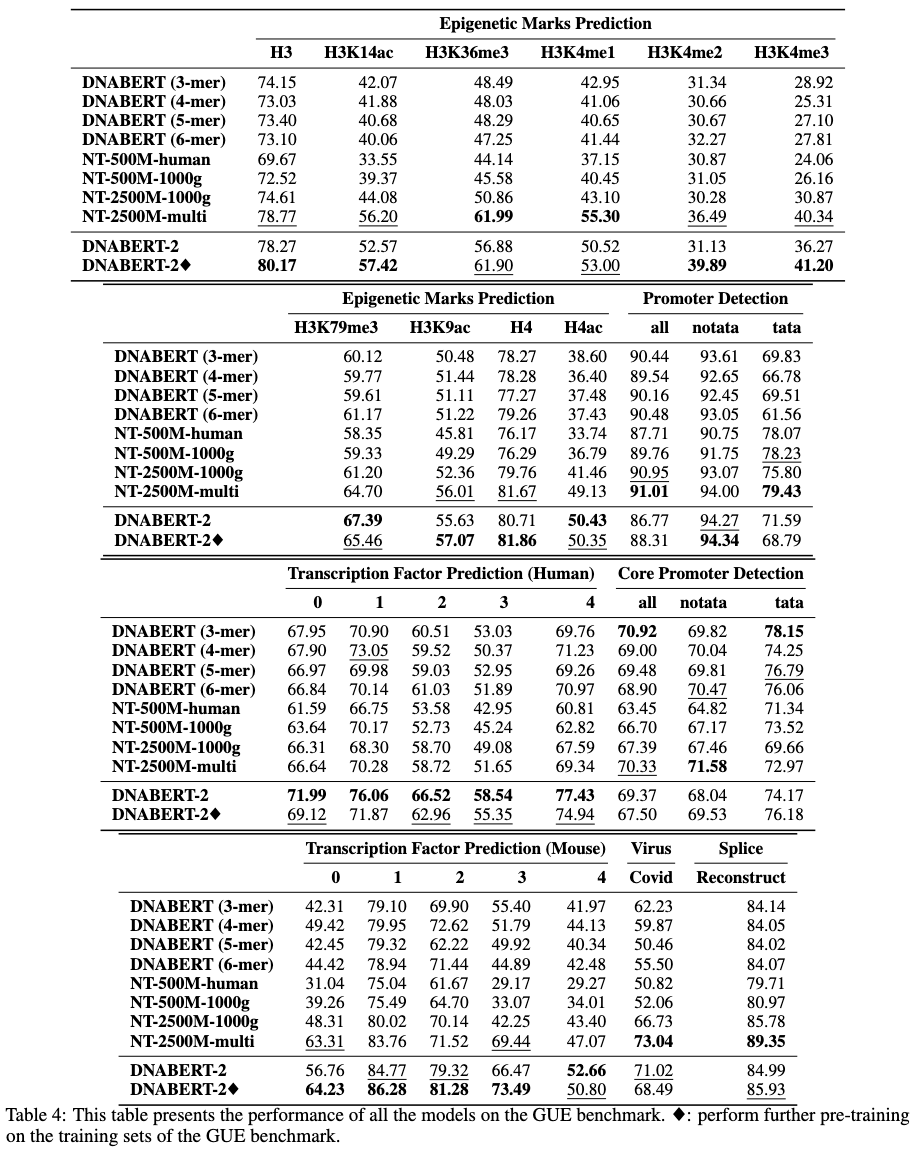
Primero descargue el conjunto de datos GUE desde aquí. Luego ejecute los scripts para evaluar todas las tareas.
El script actual está configurado para usar DataParallel para capacitación en 4 GPU. Si tiene un número diferente de GPU, cambie el per_device_train_batch_size y gradient_accumulation_steps en consecuencia para ajustar el tamaño de lotes global a 32 para replicar los resultados en el documento. Si desea realizar una capacitación distribuida de múltiples GPU (por ejemplo, con DistributedDataParallel ), simplemente cambie python a torchrun --nproc_per_node ${n_gpu} .
export DATA_PATH=/path/to/GUE #(e.g., /home/user)
cd finetune
# Evaluate DNABERT-2 on GUE
sh scripts/run_dnabert2.sh DATA_PATH
# Evaluate DNABERT (e.g., DNABERT with 3-mer) on GUE
# 3 for 3-mer, 4 for 4-mer, 5 for 5-mer, 6 for 6-mer
sh scripts/run_dnabert1.sh DATA_PATH 3
# Evaluate Nucleotide Transformers on GUE
# 0 for 500m-1000g, 1 for 500m-human-ref, 2 for 2.5b-1000g, 3 for 2.5b-multi-species
sh scripts/run_nt.sh DATA_PATH 0
Aquí proporcionamos un ejemplo de DNABERT2 ajustado en sus propios conjuntos de datos.
Primero, genere 3 archivos csv a partir de su conjunto de datos: train.csv , dev.csv y test.csv . En el proceso de capacitación, el modelo está capacitado en train.csv y se evalúa en el archivo dev.csv . Después de la capacitación si se termina, el punto de control con la pérdida más pequeña en el archivo dev.csv se carga y se evalúa en test.csv . Si no tiene un conjunto de validación, simplemente haga el dev.csv y test.csv lo mismo.
Consulte la carpeta sample_data para obtener una muestra de formato de datos. Cada archivo debe estar en el mismo formato, con la primera fila como cabezal de documento llamado sequence, label . Cada fila siguiente debe contener una secuencia de ADN y una etiqueta numérica concatenada por A , (por ejemplo, ACGTCAGTCAGCGTACGT, 1 ).
Luego, puede Finetune Dnabert-2 en su propio conjunto de datos con el siguiente código:
cd finetune
export DATA_PATH=$path/to/data/folder # e.g., ./sample_data
export MAX_LENGTH=100 # Please set the number as 0.25 * your sequence length.
# e.g., set it as 250 if your DNA sequences have 1000 nucleotide bases
# This is because the tokenized will reduce the sequence length by about 5 times
export LR=3e-5
# Training use DataParallel
python train.py
--model_name_or_path zhihan1996/DNABERT-2-117M
--data_path ${DATA_PATH}
--kmer -1
--run_name DNABERT2_${DATA_PATH}
--model_max_length ${MAX_LENGTH}
--per_device_train_batch_size 8
--per_device_eval_batch_size 16
--gradient_accumulation_steps 1
--learning_rate ${LR}
--num_train_epochs 5
--fp16
--save_steps 200
--output_dir output/dnabert2
--evaluation_strategy steps
--eval_steps 200
--warmup_steps 50
--logging_steps 100
--overwrite_output_dir True
--log_level info
--find_unused_parameters False
# Training use DistributedDataParallel (more efficient)
export num_gpu=4 # please change the value based on your setup
torchrun --nproc_per_node=${num_gpu} train.py
--model_name_or_path zhihan1996/DNABERT-2-117M
--data_path ${DATA_PATH}
--kmer -1
--run_name DNABERT2_${DATA_PATH}
--model_max_length ${MAX_LENGTH}
--per_device_train_batch_size 8
--per_device_eval_batch_size 16
--gradient_accumulation_steps 1
--learning_rate ${LR}
--num_train_epochs 5
--fp16
--save_steps 200
--output_dir output/dnabert2
--evaluation_strategy steps
--eval_steps 200
--warmup_steps 50
--logging_steps 100
--overwrite_output_dir True
--log_level info
--find_unused_parameters False
Si tiene alguna pregunta sobre nuestro documento o códigos, no dude en iniciar un problema o enviar un correo electrónico a Zhihan Zhou (zhihanzhou20202.northwestern.edu).
Si usa Dnabert-2 en su trabajo, por favor cita nuestro documento:
Dnabert-2
@misc{zhou2023dnabert2,
title={DNABERT-2: Efficient Foundation Model and Benchmark For Multi-Species Genome},
author={Zhihan Zhou and Yanrong Ji and Weijian Li and Pratik Dutta and Ramana Davuluri and Han Liu},
year={2023},
eprint={2306.15006},
archivePrefix={arXiv},
primaryClass={q-bio.GN}
}
Dnabert
@article{ji2021dnabert,
author = {Ji, Yanrong and Zhou, Zhihan and Liu, Han and Davuluri, Ramana V},
title = "{DNABERT: pre-trained Bidirectional Encoder Representations from Transformers model for DNA-language in genome}",
journal = {Bioinformatics},
volume = {37},
number = {15},
pages = {2112-2120},
year = {2021},
month = {02},
issn = {1367-4803},
doi = {10.1093/bioinformatics/btab083},
url = {https://doi.org/10.1093/bioinformatics/btab083},
eprint = {https://academic.oup.com/bioinformatics/article-pdf/37/15/2112/50578892/btab083.pdf},
}