

SCIKIT-LEARN 모델은 진화 알고리즘을 사용하여 하이퍼 파라미터 튜닝 및 기능 선택을 모델링합니다.
이는 하이퍼 파라미터 튜닝을위한 그리드 검색 및 무작위 그리드 검색과 같은 Scikit-Learn 내부의 인기있는 방법에 대한 대안이며, RFE (Recursive Feature Emination)에서 기능 선택을 위해 모델을 선택하십시오.
Sklearn-Genetic-OPT는 DEAP (Python의 분산 진화 알고리즘) 패키지의 진화 알고리즘을 사용하여 교차 검증 점수를 최적화하는 (최대 또는 최소) 회귀 및 분류 문제에 사용될 수 있습니다.
문서는 여기에서 확인할 수 있습니다
교육 진행 상황을 시각화하십시오.
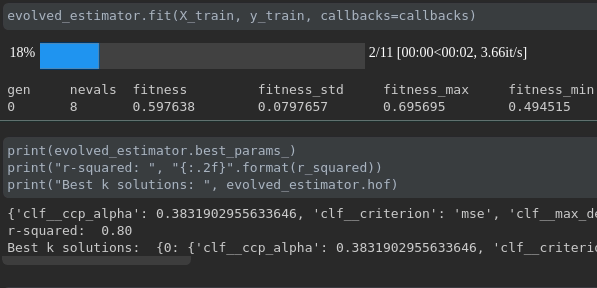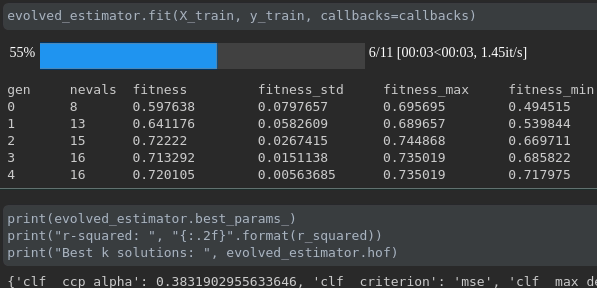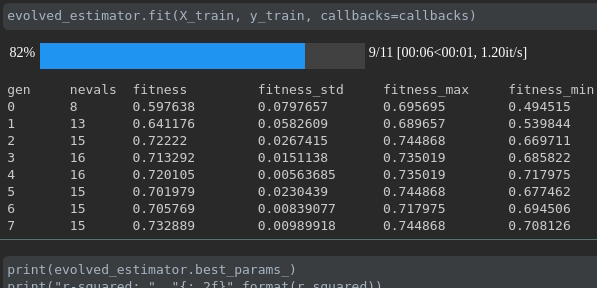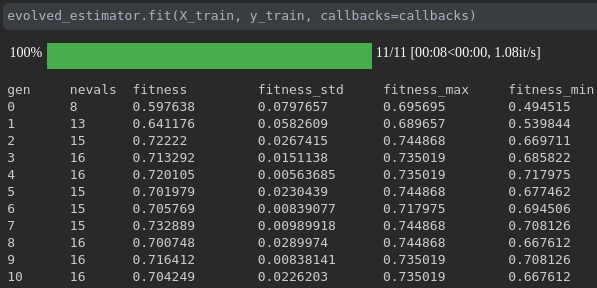
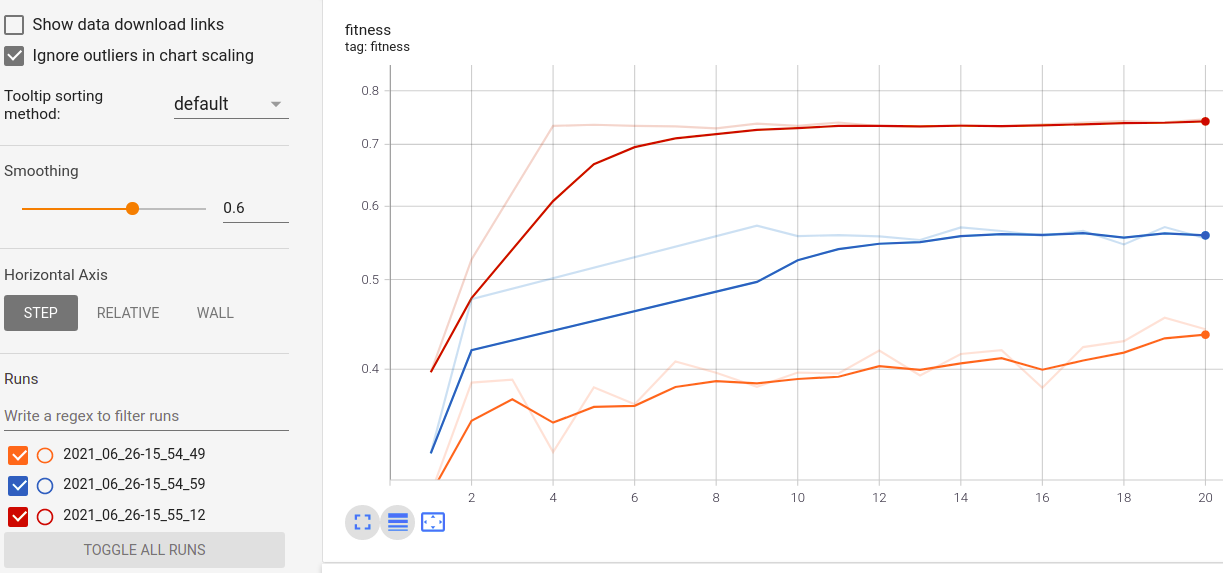
실시간 메트릭 시각화 및 실행에 대한 비교 :
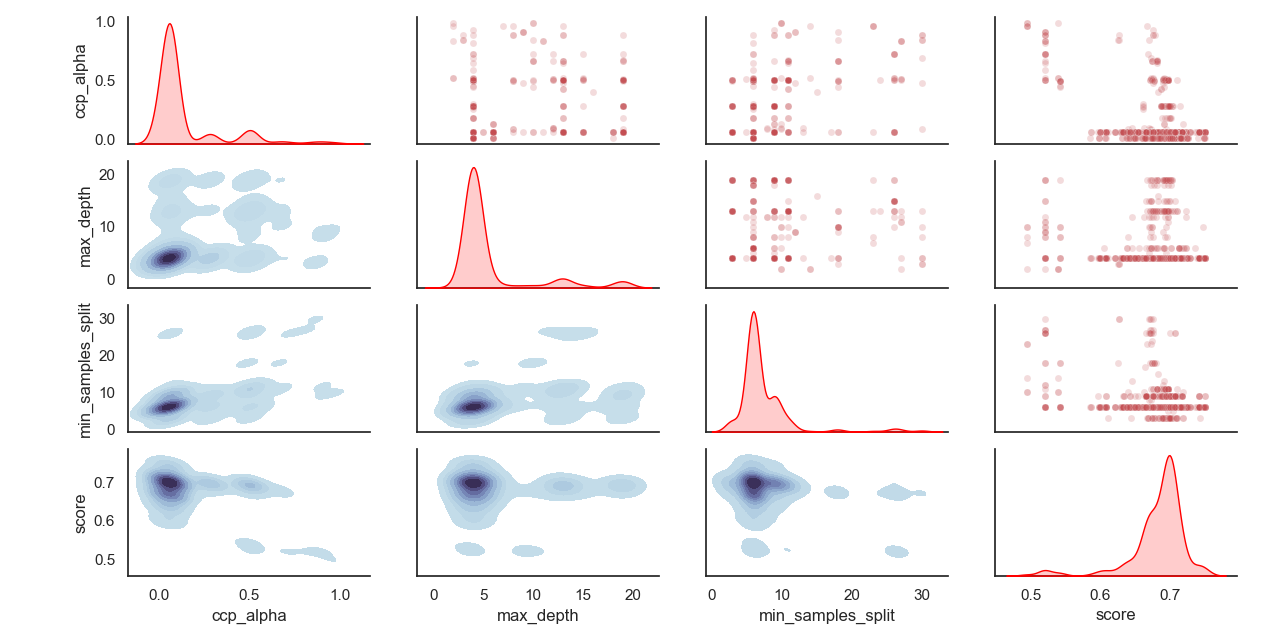
초 파라미터의 샘플링 분포 :
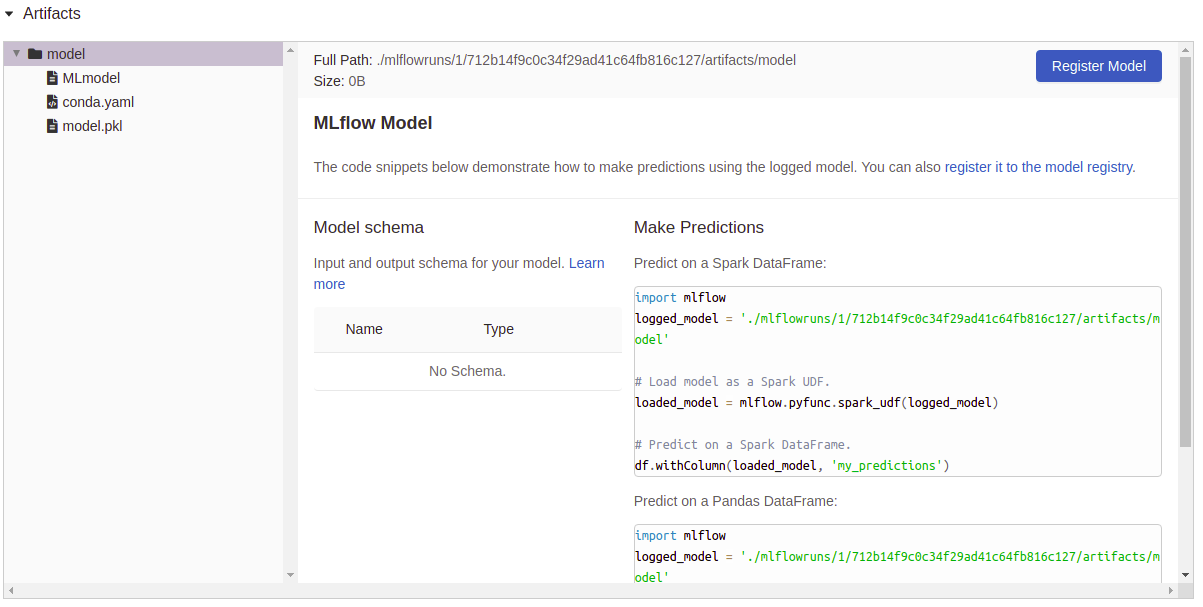
아티팩트 로깅 :
Sklearn-Genetic-opt를 설치하십시오
ENV 사용 내부에 가상 ENV를 사용하여 Sklearn-Genetic을 설치하는 것이 좋습니다.
PIP Sklearn-Genetic-opt를 설치하십시오
플로팅, 텐서 보드 및 MLFLOW 로깅 기능을 포함한 모든 기능을 얻으려면 모든 추가 패키지를 설치하십시오.
PIP Sklearn-Genetic-OPT 설치 [모두]
from sklearn_genetic import GASearchCV
from sklearn_genetic . space import Continuous , Categorical , Integer
from sklearn . ensemble import RandomForestClassifier
from sklearn . model_selection import train_test_split , StratifiedKFold
from sklearn . datasets import load_digits
from sklearn . metrics import accuracy_score
data = load_digits ()
n_samples = len ( data . images )
X = data . images . reshape (( n_samples , - 1 ))
y = data [ 'target' ]
X_train , X_test , y_train , y_test = train_test_split ( X , y , test_size = 0.33 , random_state = 42 )
clf = RandomForestClassifier ()
# Defines the possible values to search
param_grid = { 'min_weight_fraction_leaf' : Continuous ( 0.01 , 0.5 , distribution = 'log-uniform' ),
'bootstrap' : Categorical ([ True , False ]),
'max_depth' : Integer ( 2 , 30 ),
'max_leaf_nodes' : Integer ( 2 , 35 ),
'n_estimators' : Integer ( 100 , 300 )}
# Seed solutions
warm_start_configs = [
{ "min_weight_fraction_leaf" : 0.02 , "bootstrap" : True , "max_depth" : None , "n_estimators" : 100 },
{ "min_weight_fraction_leaf" : 0.4 , "bootstrap" : True , "max_depth" : 5 , "n_estimators" : 200 },
]
cv = StratifiedKFold ( n_splits = 3 , shuffle = True )
evolved_estimator = GASearchCV ( estimator = clf ,
cv = cv ,
scoring = 'accuracy' ,
population_size = 20 ,
generations = 35 ,
param_grid = param_grid ,
n_jobs = - 1 ,
verbose = True ,
use_cache = True ,
warm_start_configs = warm_start_configs ,
keep_top_k = 4 )
# Train and optimize the estimator
evolved_estimator . fit ( X_train , y_train )
# Best parameters found
print ( evolved_estimator . best_params_ )
# Use the model fitted with the best parameters
y_predict_ga = evolved_estimator . predict ( X_test )
print ( accuracy_score ( y_test , y_predict_ga ))
# Saved metadata for further analysis
print ( "Stats achieved in each generation: " , evolved_estimator . history )
print ( "Best k solutions: " , evolved_estimator . hof ) from sklearn_genetic import GAFeatureSelectionCV , ExponentialAdapter
from sklearn . model_selection import train_test_split
from sklearn . svm import SVC
from sklearn . datasets import load_iris
from sklearn . metrics import accuracy_score
import numpy as np
data = load_iris ()
X , y = data [ "data" ], data [ "target" ]
# Add random non-important features
noise = np . random . uniform ( 5 , 10 , size = ( X . shape [ 0 ], 5 ))
X = np . hstack (( X , noise ))
X_train , X_test , y_train , y_test = train_test_split ( X , y , test_size = 0.33 , random_state = 0 )
clf = SVC ( gamma = 'auto' )
mutation_scheduler = ExponentialAdapter ( 0.8 , 0.2 , 0.01 )
crossover_scheduler = ExponentialAdapter ( 0.2 , 0.8 , 0.01 )
evolved_estimator = GAFeatureSelectionCV (
estimator = clf ,
scoring = "accuracy" ,
population_size = 30 ,
generations = 20 ,
mutation_probability = mutation_scheduler ,
crossover_probability = crossover_scheduler ,
n_jobs = - 1 )
# Train and select the features
evolved_estimator . fit ( X_train , y_train )
# Features selected by the algorithm
features = evolved_estimator . support_
print ( features )
# Predict only with the subset of selected features
y_predict_ga = evolved_estimator . predict ( X_test )
print ( accuracy_score ( y_test , y_predict_ga ))
# Transform the original data to the selected features
X_reduced = evolved_estimator . transform ( X_test )Sklearn-Genetic-opt의 변화에 대한 메모는 Changelog를 참조하십시오.
명령으로 최신 개발 버전을 확인할 수 있습니다.
git 클론 https://github.com/rodrigo-arenas/sklearn-genetic-opt.git
개발 종속성 설치 :
PIP 설치 -r dev -requirements.txt
최신 개발중인 문서를 확인하십시오 : https://sklearn-genetic-opt.readthedocs.io/en/latest/
기부금은 환영받는 것 이상입니다! 진행중인 프로젝트에는 몇 가지 기회가 있으므로 도와주고 싶다면 연락하십시오. 현재 문제와 기여 가이드를 확인하십시오.
이 프로젝트를 돕고있는 사람들에게 큰 감사를드립니다!
설치 후 소스 디렉토리 외부에서 테스트 스위트를 시작할 수 있습니다.
pytest sklearn_genetic