

SCIKIT-LEARNモデルのハイパーパラメーターの調整と機能の選択。進化的アルゴリズムを使用します。
これは、グリッド検索やハイパーパラメータチューニングのランダム化グリッド検索など、Scikit-Learn内の一般的な方法に代わるものであり、RFE(再帰機能除去)から、機能選択のためにモデルから選択します。
Sklearn-genetic-OPTは、DEAP(Pythonの分散進化アルゴリズム)パッケージの進化アルゴリズムを使用して、交差検証スコアを最適化(最大またはmin)するハイパーパラメーターのセットを選択します。回帰と分類の問題の両方に使用できます。
ドキュメントはこちらから入手できます
トレーニングの進捗状況を視覚化します。
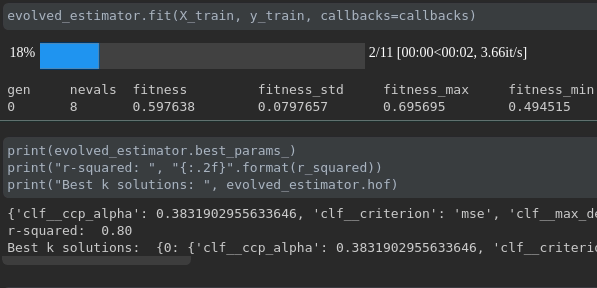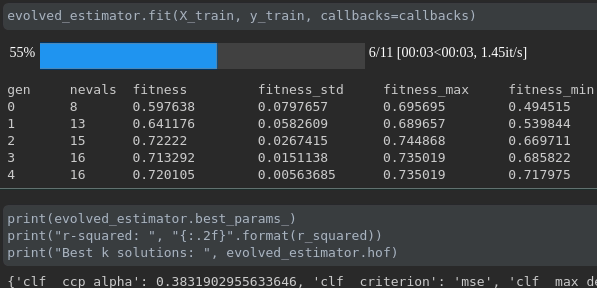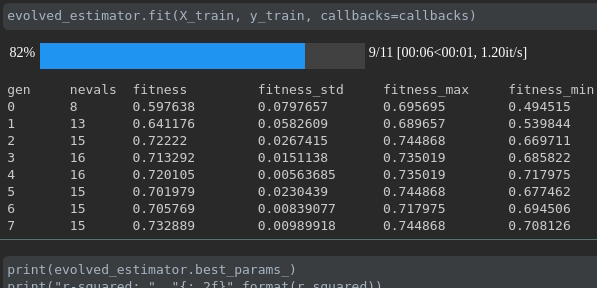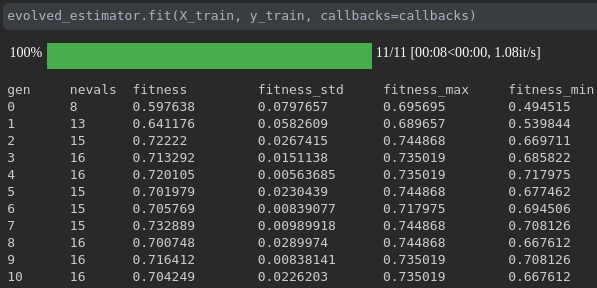
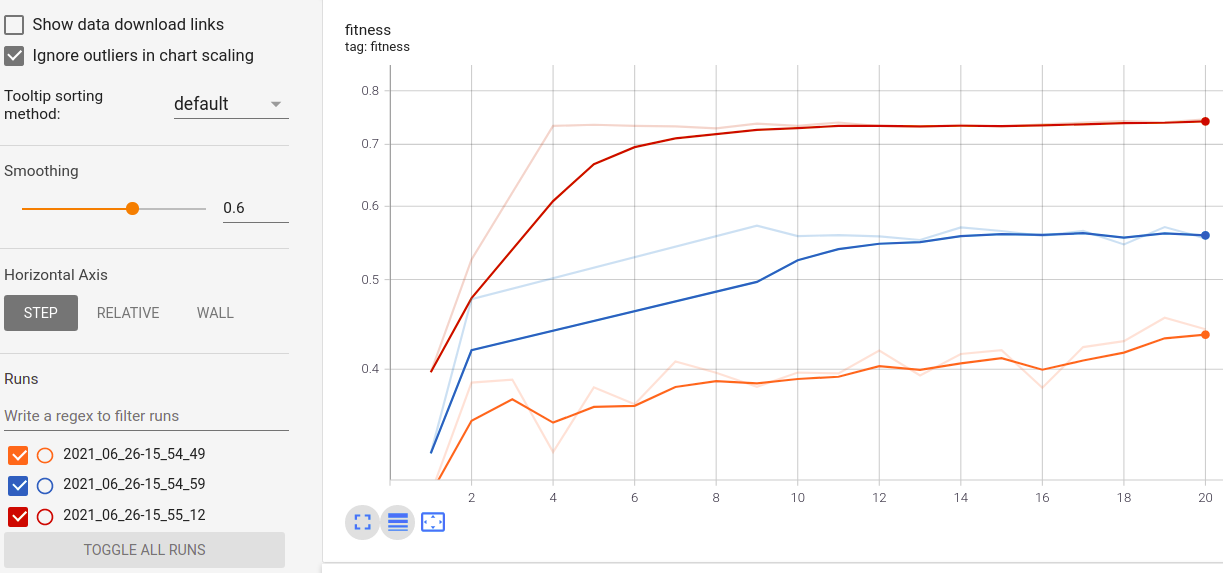
ラン全体のリアルタイムメトリックの視覚化と比較:
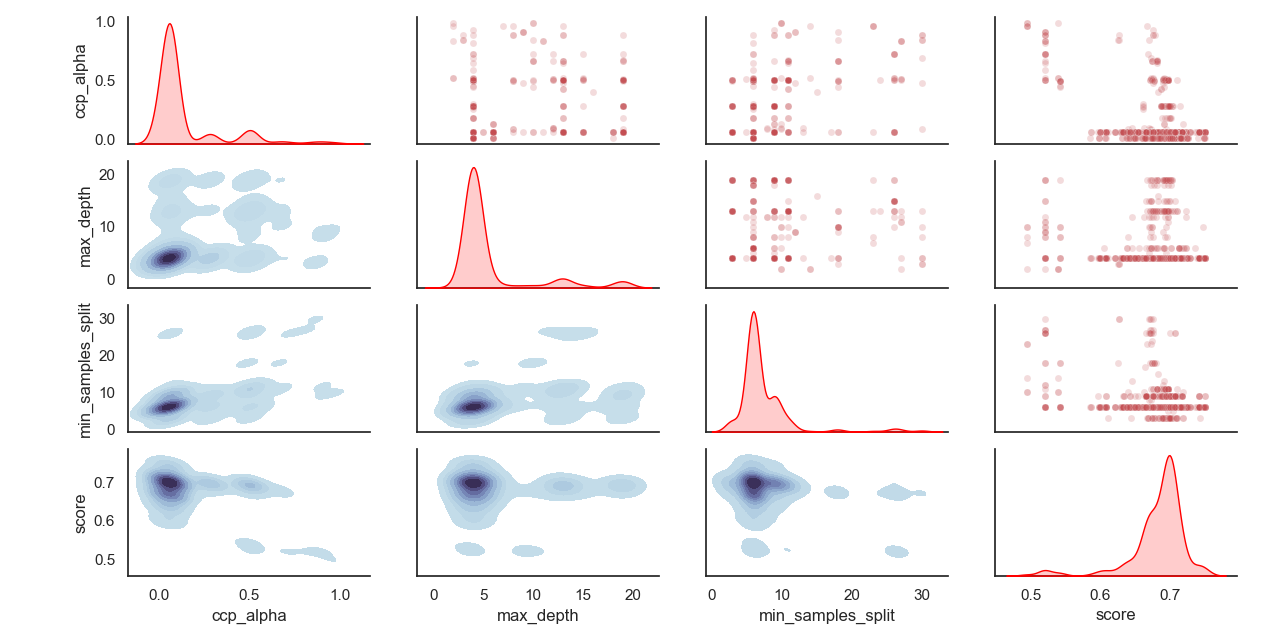
ハイパーパラメーターのサンプリングされた分布:
アーティファクトロギング:
sklearn-genetic-optをインストールします
virtual envを使用してsklearn-geneticをインストールすることをお勧めします。
PIPインストールSklearn-genetic-opt
プロット、テンソルボード、MLFLOWロギング機能など、すべての機能を取得する場合は、すべての追加パッケージをインストールします。
PIPインストールSklearn-genetic-opt [すべて]
from sklearn_genetic import GASearchCV
from sklearn_genetic . space import Continuous , Categorical , Integer
from sklearn . ensemble import RandomForestClassifier
from sklearn . model_selection import train_test_split , StratifiedKFold
from sklearn . datasets import load_digits
from sklearn . metrics import accuracy_score
data = load_digits ()
n_samples = len ( data . images )
X = data . images . reshape (( n_samples , - 1 ))
y = data [ 'target' ]
X_train , X_test , y_train , y_test = train_test_split ( X , y , test_size = 0.33 , random_state = 42 )
clf = RandomForestClassifier ()
# Defines the possible values to search
param_grid = { 'min_weight_fraction_leaf' : Continuous ( 0.01 , 0.5 , distribution = 'log-uniform' ),
'bootstrap' : Categorical ([ True , False ]),
'max_depth' : Integer ( 2 , 30 ),
'max_leaf_nodes' : Integer ( 2 , 35 ),
'n_estimators' : Integer ( 100 , 300 )}
# Seed solutions
warm_start_configs = [
{ "min_weight_fraction_leaf" : 0.02 , "bootstrap" : True , "max_depth" : None , "n_estimators" : 100 },
{ "min_weight_fraction_leaf" : 0.4 , "bootstrap" : True , "max_depth" : 5 , "n_estimators" : 200 },
]
cv = StratifiedKFold ( n_splits = 3 , shuffle = True )
evolved_estimator = GASearchCV ( estimator = clf ,
cv = cv ,
scoring = 'accuracy' ,
population_size = 20 ,
generations = 35 ,
param_grid = param_grid ,
n_jobs = - 1 ,
verbose = True ,
use_cache = True ,
warm_start_configs = warm_start_configs ,
keep_top_k = 4 )
# Train and optimize the estimator
evolved_estimator . fit ( X_train , y_train )
# Best parameters found
print ( evolved_estimator . best_params_ )
# Use the model fitted with the best parameters
y_predict_ga = evolved_estimator . predict ( X_test )
print ( accuracy_score ( y_test , y_predict_ga ))
# Saved metadata for further analysis
print ( "Stats achieved in each generation: " , evolved_estimator . history )
print ( "Best k solutions: " , evolved_estimator . hof ) from sklearn_genetic import GAFeatureSelectionCV , ExponentialAdapter
from sklearn . model_selection import train_test_split
from sklearn . svm import SVC
from sklearn . datasets import load_iris
from sklearn . metrics import accuracy_score
import numpy as np
data = load_iris ()
X , y = data [ "data" ], data [ "target" ]
# Add random non-important features
noise = np . random . uniform ( 5 , 10 , size = ( X . shape [ 0 ], 5 ))
X = np . hstack (( X , noise ))
X_train , X_test , y_train , y_test = train_test_split ( X , y , test_size = 0.33 , random_state = 0 )
clf = SVC ( gamma = 'auto' )
mutation_scheduler = ExponentialAdapter ( 0.8 , 0.2 , 0.01 )
crossover_scheduler = ExponentialAdapter ( 0.2 , 0.8 , 0.01 )
evolved_estimator = GAFeatureSelectionCV (
estimator = clf ,
scoring = "accuracy" ,
population_size = 30 ,
generations = 20 ,
mutation_probability = mutation_scheduler ,
crossover_probability = crossover_scheduler ,
n_jobs = - 1 )
# Train and select the features
evolved_estimator . fit ( X_train , y_train )
# Features selected by the algorithm
features = evolved_estimator . support_
print ( features )
# Predict only with the subset of selected features
y_predict_ga = evolved_estimator . predict ( X_test )
print ( accuracy_score ( y_test , y_predict_ga ))
# Transform the original data to the selected features
X_reduced = evolved_estimator . transform ( X_test )sklearn-genetic-optの変更に関するメモについては、changelogを参照してください
コマンドで最新の開発バージョンを確認できます。
git clone https://github.com/rodrigo-arenas/sklearn-genetic-opt.git
開発依存関係をインストールします。
PIPインストール-R dev -requirements.txt
最新の開発中のドキュメントを確認してください:https://sklearn-genetic-opt.readthedocs.io/en/latest/
貢献は大歓迎です!進行中のプロジェクトにはいくつかの機会がありますので、お手伝いしたい場合はご連絡ください。現在の問題と貢献ガイドを確認してください。
このプロジェクトを手伝ってくれた人々に感謝します!
インストール後、ソースディレクトリの外部からテストスイートを起動できます。
pytest sklearn_genetic