

Scikit-learn modela la sintonización de hiperparámetros y la selección de características, utilizando algoritmos evolutivos.
Esto está destinado a ser una alternativa a los métodos populares dentro de Scikit-Learn, como la búsqueda de la cuadrícula y la búsqueda aleatoria de la cuadrícula para el ajuste de los hiperparámetros, y desde RFE (eliminación de características recursivas), seleccione del modelo para la selección de características.
Sklearn-Genetic-OPT utiliza algoritmos evolutivos del paquete DEAP (algoritmos evolutivos distribuidos en Python) para elegir el conjunto de hiperparámetros que optimiza (máximo o min) las puntuaciones de validación cruzada, se puede usar para problemas de regresión y clasificación.
La documentación está disponible aquí
Visualice el progreso de su entrenamiento:
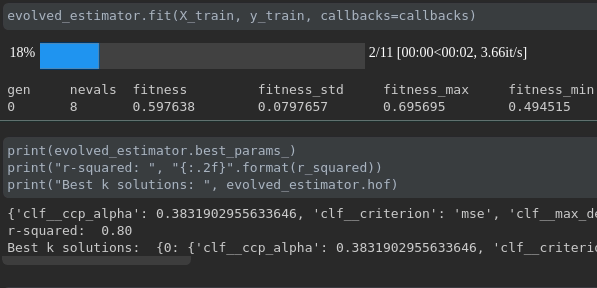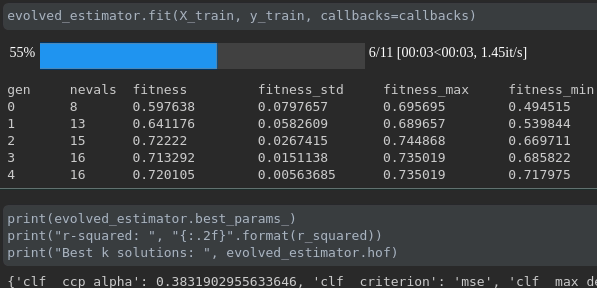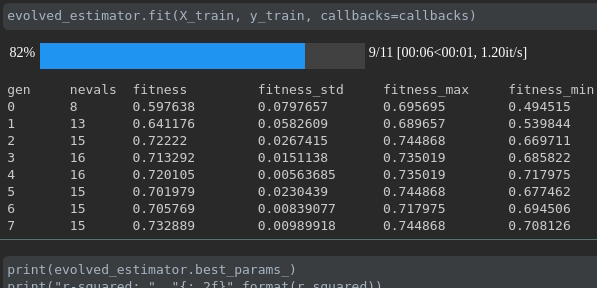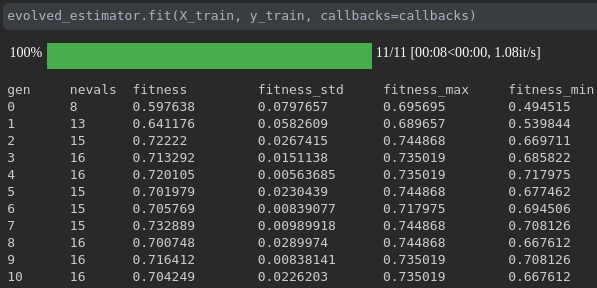
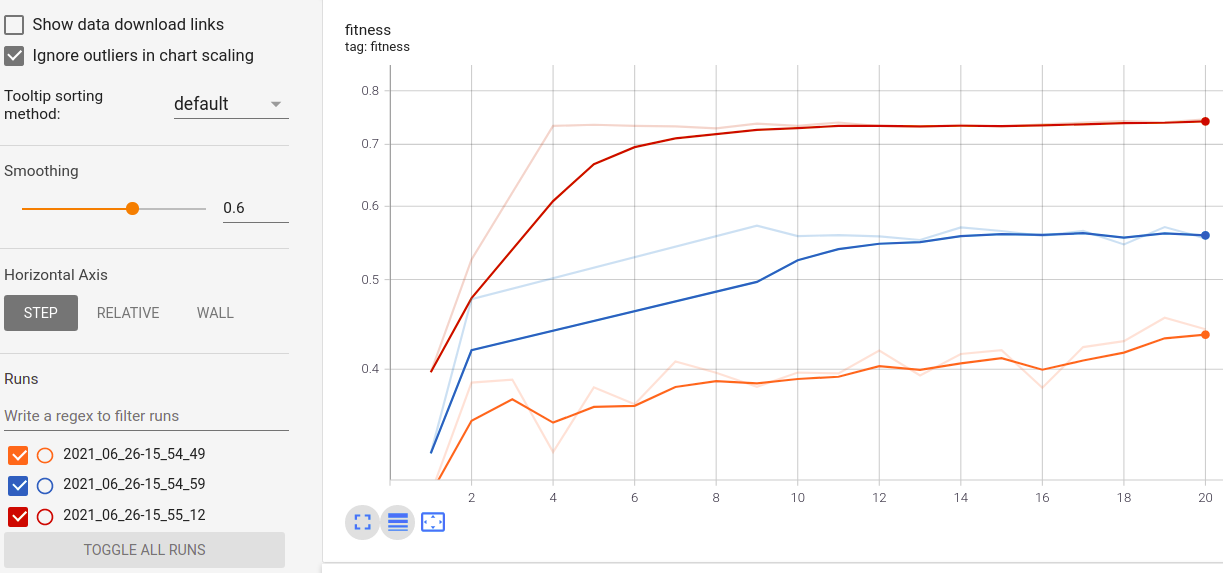
Visualización y comparación de métricas en tiempo real a través de ejecuciones:
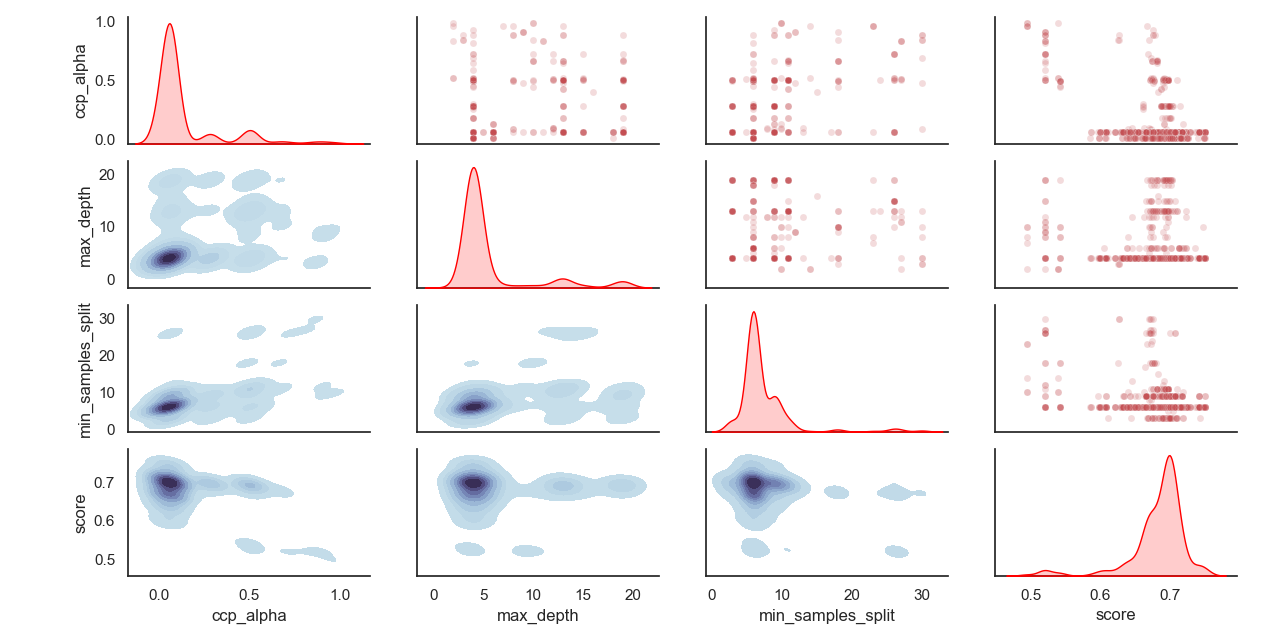
Distribución de hiperparámetros muestreados:
Registro de artefactos:
Instalar sklearn-genetic-opt
Se recomienda instalar Sklearn-Genetic utilizando un env de vista virtual, dentro del uso de env:
PIP install sklearn-Genetic-opt
Si desea obtener todas las funciones, incluidas las capacidades de trazado, TensorBoard y Mlflow de registro, instale todos los paquetes adicionales:
PIP install sklearn-Genetic-opt [all]
from sklearn_genetic import GASearchCV
from sklearn_genetic . space import Continuous , Categorical , Integer
from sklearn . ensemble import RandomForestClassifier
from sklearn . model_selection import train_test_split , StratifiedKFold
from sklearn . datasets import load_digits
from sklearn . metrics import accuracy_score
data = load_digits ()
n_samples = len ( data . images )
X = data . images . reshape (( n_samples , - 1 ))
y = data [ 'target' ]
X_train , X_test , y_train , y_test = train_test_split ( X , y , test_size = 0.33 , random_state = 42 )
clf = RandomForestClassifier ()
# Defines the possible values to search
param_grid = { 'min_weight_fraction_leaf' : Continuous ( 0.01 , 0.5 , distribution = 'log-uniform' ),
'bootstrap' : Categorical ([ True , False ]),
'max_depth' : Integer ( 2 , 30 ),
'max_leaf_nodes' : Integer ( 2 , 35 ),
'n_estimators' : Integer ( 100 , 300 )}
# Seed solutions
warm_start_configs = [
{ "min_weight_fraction_leaf" : 0.02 , "bootstrap" : True , "max_depth" : None , "n_estimators" : 100 },
{ "min_weight_fraction_leaf" : 0.4 , "bootstrap" : True , "max_depth" : 5 , "n_estimators" : 200 },
]
cv = StratifiedKFold ( n_splits = 3 , shuffle = True )
evolved_estimator = GASearchCV ( estimator = clf ,
cv = cv ,
scoring = 'accuracy' ,
population_size = 20 ,
generations = 35 ,
param_grid = param_grid ,
n_jobs = - 1 ,
verbose = True ,
use_cache = True ,
warm_start_configs = warm_start_configs ,
keep_top_k = 4 )
# Train and optimize the estimator
evolved_estimator . fit ( X_train , y_train )
# Best parameters found
print ( evolved_estimator . best_params_ )
# Use the model fitted with the best parameters
y_predict_ga = evolved_estimator . predict ( X_test )
print ( accuracy_score ( y_test , y_predict_ga ))
# Saved metadata for further analysis
print ( "Stats achieved in each generation: " , evolved_estimator . history )
print ( "Best k solutions: " , evolved_estimator . hof ) from sklearn_genetic import GAFeatureSelectionCV , ExponentialAdapter
from sklearn . model_selection import train_test_split
from sklearn . svm import SVC
from sklearn . datasets import load_iris
from sklearn . metrics import accuracy_score
import numpy as np
data = load_iris ()
X , y = data [ "data" ], data [ "target" ]
# Add random non-important features
noise = np . random . uniform ( 5 , 10 , size = ( X . shape [ 0 ], 5 ))
X = np . hstack (( X , noise ))
X_train , X_test , y_train , y_test = train_test_split ( X , y , test_size = 0.33 , random_state = 0 )
clf = SVC ( gamma = 'auto' )
mutation_scheduler = ExponentialAdapter ( 0.8 , 0.2 , 0.01 )
crossover_scheduler = ExponentialAdapter ( 0.2 , 0.8 , 0.01 )
evolved_estimator = GAFeatureSelectionCV (
estimator = clf ,
scoring = "accuracy" ,
population_size = 30 ,
generations = 20 ,
mutation_probability = mutation_scheduler ,
crossover_probability = crossover_scheduler ,
n_jobs = - 1 )
# Train and select the features
evolved_estimator . fit ( X_train , y_train )
# Features selected by the algorithm
features = evolved_estimator . support_
print ( features )
# Predict only with the subset of selected features
y_predict_ga = evolved_estimator . predict ( X_test )
print ( accuracy_score ( y_test , y_predict_ga ))
# Transform the original data to the selected features
X_reduced = evolved_estimator . transform ( X_test )Consulte The ChangeLog para obtener notas sobre los cambios de Sklearn-Genetic-opt
Puede consultar la última versión de desarrollo con el comando:
Git clon https://github.com/rodrigo-arenas/sklearn-genetic-opt.git
Instale las dependencias de desarrollo:
PIP install -r dev -requirements.txt
Consulte la última documentación de desarrollo en el desarrollo: https://sklearn-genetic-opt.readthedocs.io/en/latest/
¡Las contribuciones son más que bienvenidas! Hay varias oportunidades en el proyecto en curso, así que póngase en contacto si desea ayudar. Asegúrese de verificar los problemas actuales y también la guía de contribución.
¡Muchas gracias a las personas que están ayudando con este proyecto!
Después de la instalación, puede iniciar el conjunto de pruebas desde fuera del directorio de origen:
pytest sklearn_genetic