

Scikit-Learn Modèles Hyperparameters Taping et Sélection de fonctionnalités, en utilisant des algorithmes évolutifs.
Ceci est censé être une alternative aux méthodes populaires à l'intérieur de Scikit-Learn telles que la recherche de grille et la recherche randomisée de grille pour le réglage des hyperparamètres et à partir de RFE (élimination des fonctionnalités récursives), sélectionnez parmi le modèle pour la sélection des fonctionnalités.
Sklearn-Genetic-OPT utilise des algorithmes évolutifs du package DEAP (algorithmes évolutifs distribués dans Python) pour choisir l'ensemble des hyperparamètres qui optimise (Max ou Min) les scores de validation croisée, il peut être utilisé pour les problèmes de régression et de classification.
La documentation est disponible ici
Visualisez la progression de votre formation:
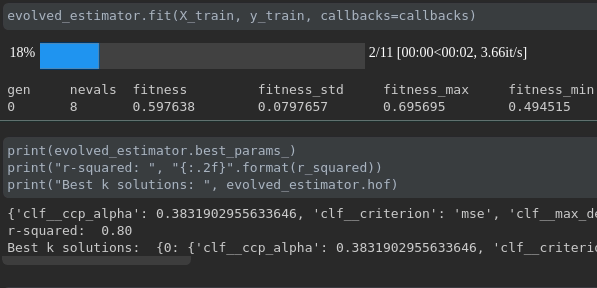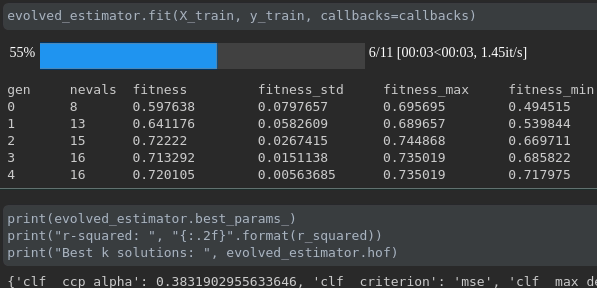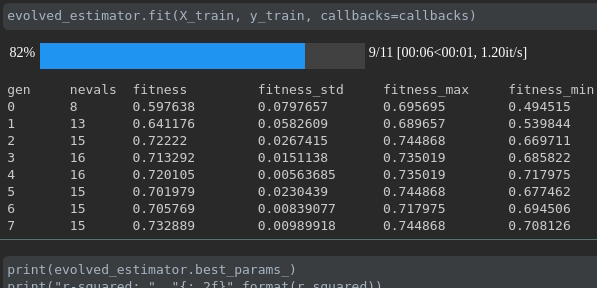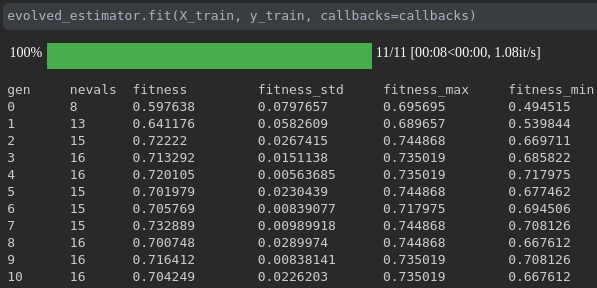
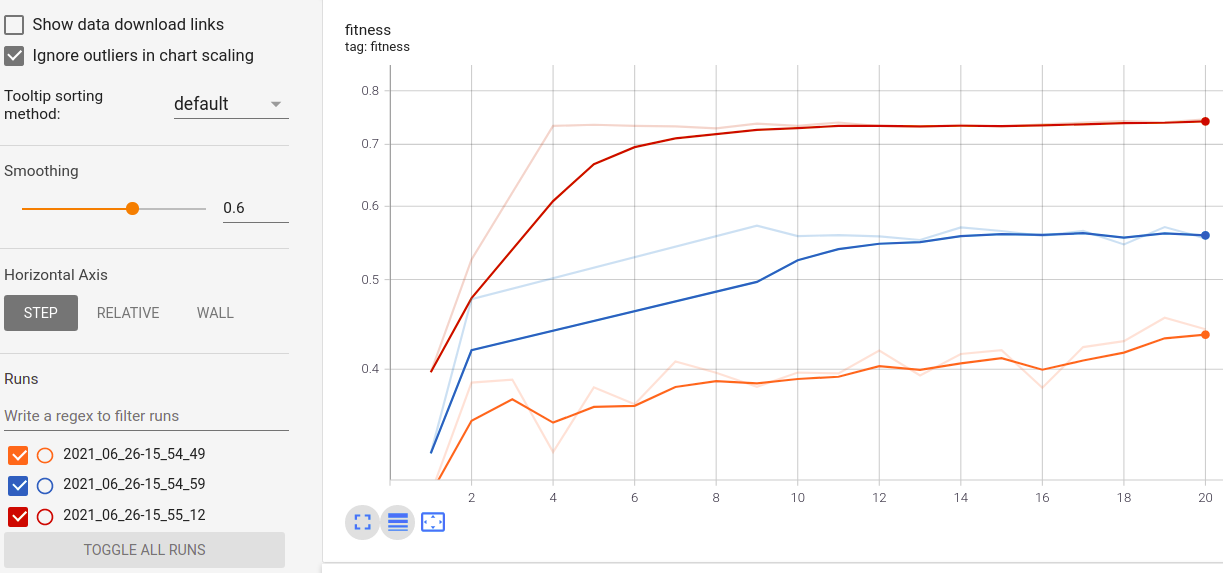
Visualisation et comparaison des métriques en temps réel entre les courses:
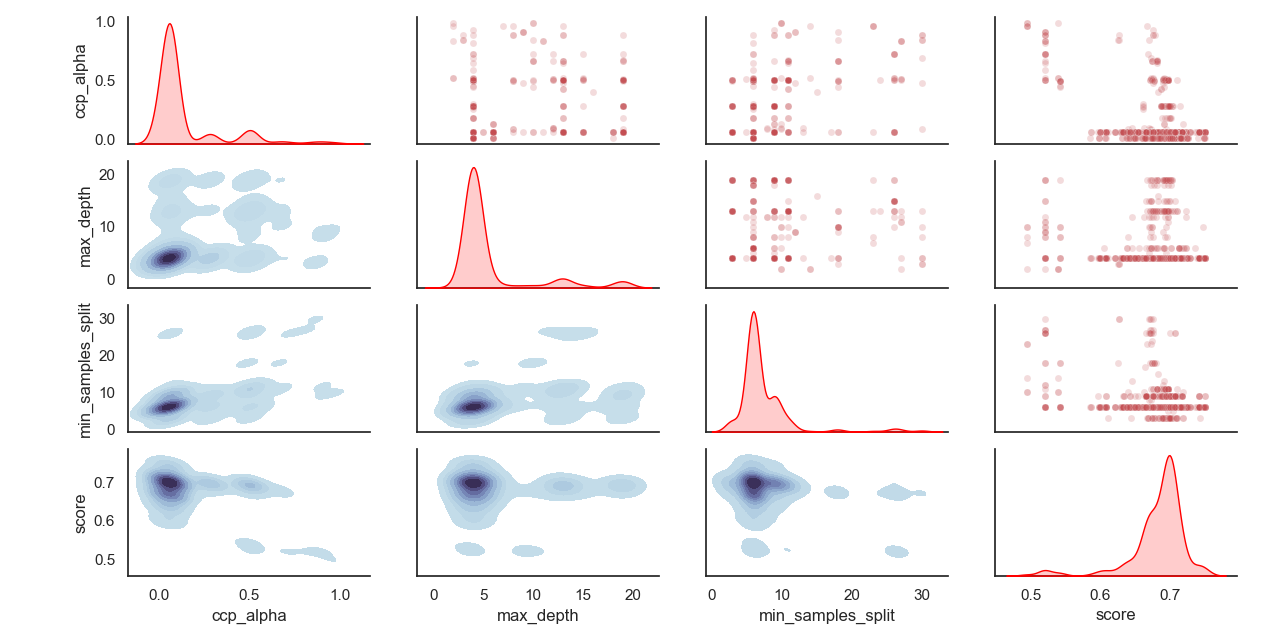
Distribution échantillonnée des hyperparamètres:
Logotage des artefacts:
Installer Sklearn-Genetic-Opt
Il est conseillé d'installer Sklearn-Génétique à l'aide d'un ENV virtuel, à l'intérieur de l'utilisation Env:
PIP installe Sklearn-Genetic-Opt
Si vous souhaitez obtenir toutes les fonctionnalités, y compris le traçage, les capacités de journalisation Tensorboard et MLFlow, installez tous les packages supplémentaires:
PIP installe Sklearn-Genetic-opt [tous]
from sklearn_genetic import GASearchCV
from sklearn_genetic . space import Continuous , Categorical , Integer
from sklearn . ensemble import RandomForestClassifier
from sklearn . model_selection import train_test_split , StratifiedKFold
from sklearn . datasets import load_digits
from sklearn . metrics import accuracy_score
data = load_digits ()
n_samples = len ( data . images )
X = data . images . reshape (( n_samples , - 1 ))
y = data [ 'target' ]
X_train , X_test , y_train , y_test = train_test_split ( X , y , test_size = 0.33 , random_state = 42 )
clf = RandomForestClassifier ()
# Defines the possible values to search
param_grid = { 'min_weight_fraction_leaf' : Continuous ( 0.01 , 0.5 , distribution = 'log-uniform' ),
'bootstrap' : Categorical ([ True , False ]),
'max_depth' : Integer ( 2 , 30 ),
'max_leaf_nodes' : Integer ( 2 , 35 ),
'n_estimators' : Integer ( 100 , 300 )}
# Seed solutions
warm_start_configs = [
{ "min_weight_fraction_leaf" : 0.02 , "bootstrap" : True , "max_depth" : None , "n_estimators" : 100 },
{ "min_weight_fraction_leaf" : 0.4 , "bootstrap" : True , "max_depth" : 5 , "n_estimators" : 200 },
]
cv = StratifiedKFold ( n_splits = 3 , shuffle = True )
evolved_estimator = GASearchCV ( estimator = clf ,
cv = cv ,
scoring = 'accuracy' ,
population_size = 20 ,
generations = 35 ,
param_grid = param_grid ,
n_jobs = - 1 ,
verbose = True ,
use_cache = True ,
warm_start_configs = warm_start_configs ,
keep_top_k = 4 )
# Train and optimize the estimator
evolved_estimator . fit ( X_train , y_train )
# Best parameters found
print ( evolved_estimator . best_params_ )
# Use the model fitted with the best parameters
y_predict_ga = evolved_estimator . predict ( X_test )
print ( accuracy_score ( y_test , y_predict_ga ))
# Saved metadata for further analysis
print ( "Stats achieved in each generation: " , evolved_estimator . history )
print ( "Best k solutions: " , evolved_estimator . hof ) from sklearn_genetic import GAFeatureSelectionCV , ExponentialAdapter
from sklearn . model_selection import train_test_split
from sklearn . svm import SVC
from sklearn . datasets import load_iris
from sklearn . metrics import accuracy_score
import numpy as np
data = load_iris ()
X , y = data [ "data" ], data [ "target" ]
# Add random non-important features
noise = np . random . uniform ( 5 , 10 , size = ( X . shape [ 0 ], 5 ))
X = np . hstack (( X , noise ))
X_train , X_test , y_train , y_test = train_test_split ( X , y , test_size = 0.33 , random_state = 0 )
clf = SVC ( gamma = 'auto' )
mutation_scheduler = ExponentialAdapter ( 0.8 , 0.2 , 0.01 )
crossover_scheduler = ExponentialAdapter ( 0.2 , 0.8 , 0.01 )
evolved_estimator = GAFeatureSelectionCV (
estimator = clf ,
scoring = "accuracy" ,
population_size = 30 ,
generations = 20 ,
mutation_probability = mutation_scheduler ,
crossover_probability = crossover_scheduler ,
n_jobs = - 1 )
# Train and select the features
evolved_estimator . fit ( X_train , y_train )
# Features selected by the algorithm
features = evolved_estimator . support_
print ( features )
# Predict only with the subset of selected features
y_predict_ga = evolved_estimator . predict ( X_test )
print ( accuracy_score ( y_test , y_predict_ga ))
# Transform the original data to the selected features
X_reduced = evolved_estimator . transform ( X_test )Voir le Changelog pour les notes sur les changements de Sklearn-Genetic-Opt
Vous pouvez vérifier la dernière version de développement avec la commande:
git clone https://github.com/rodrigo-arenas/sklearn-genetic-opt.git
Installez les dépendances de développement:
pip install -r dev-requiments.txt
Consultez la dernière documentation en développement intérieur: https://sklearn-genetic-opt.readthedocs.io/en/latest/
Les contributions sont plus que les bienvenues! Il existe plusieurs opportunités sur le projet en cours, alors contactez-nous si vous souhaitez vous aider. Assurez-vous de vérifier les problèmes actuels ainsi que le guide de contribution.
Un grand merci aux personnes qui aident ce projet!
Après l'installation, vous pouvez lancer la suite de test depuis l'extérieur du répertoire source:
pytest sklearn_genetic