

Scikit-Learn-Modelle Hyperparameter-Tuning- und Feature-Auswahl unter Verwendung evolutionärer Algorithmen.
Dies soll eine Alternative zu beliebten Methoden innerhalb von Scikit-Learn sein, wie z. B. Rastersuche und randomisierte Gittersuche nach Hyperparametern und aus RFE (rekursive Feature-Eliminierung), wählen Sie vom Modell für die Auswahl der Merkmale aus.
Sklearn-Genetic-opt verwendet evolutionäre Algorithmen aus dem DEAP-Paket (verteilte evolutionäre Algorithmen in Python), um den Satz von Hyperparametern auszuwählen, die (max oder min) die Quervalidierungswerte optimieren, und es kann sowohl für Regressions- als auch für Klassifizierungsprobleme verwendet werden.
Dokumentation ist hier verfügbar
Visualisieren Sie den Fortschritt Ihres Trainings:
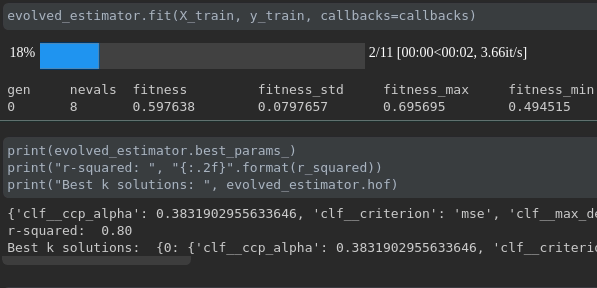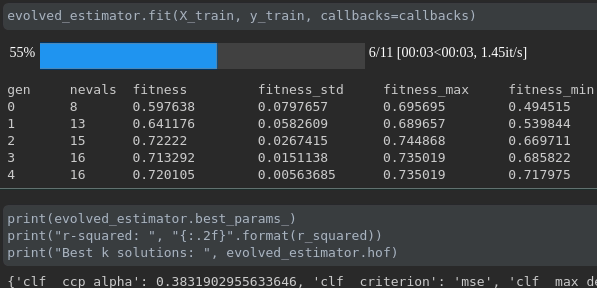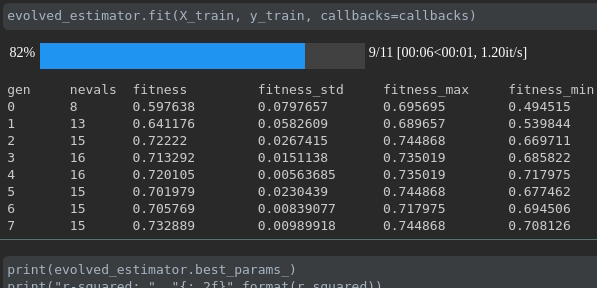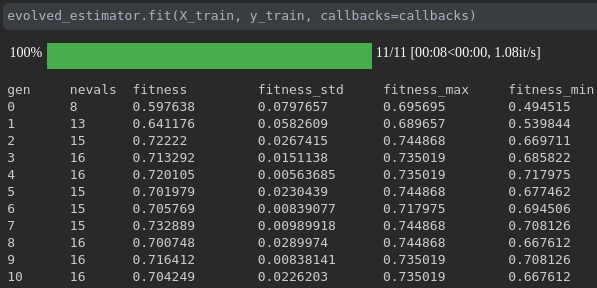
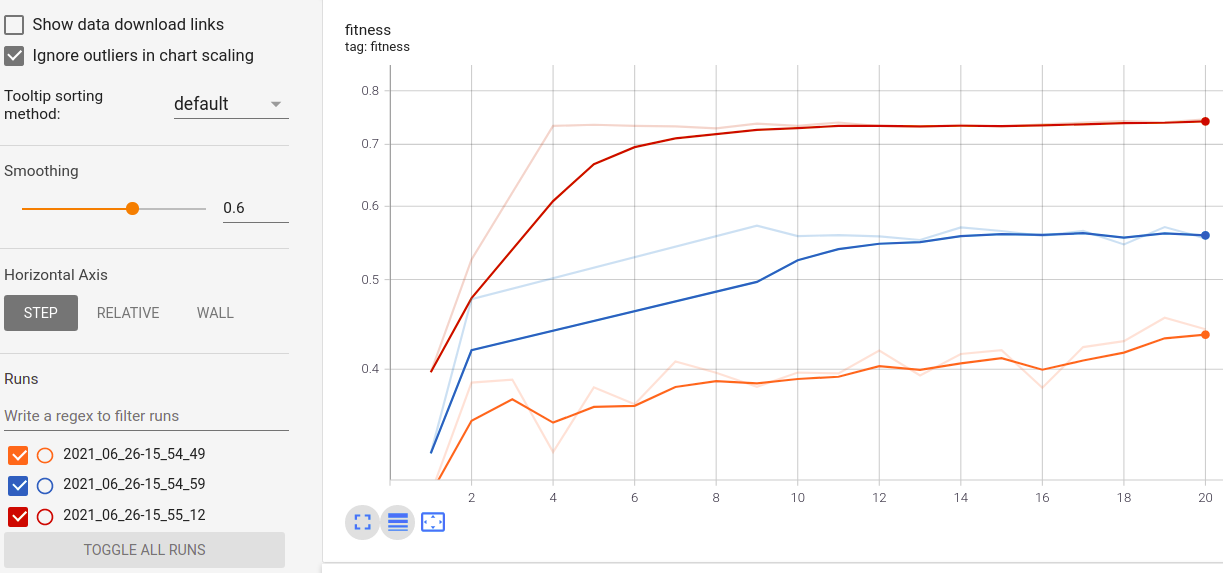
Visualisierung und Vergleich von Echtzeitmetriken über Läufe hinweg:
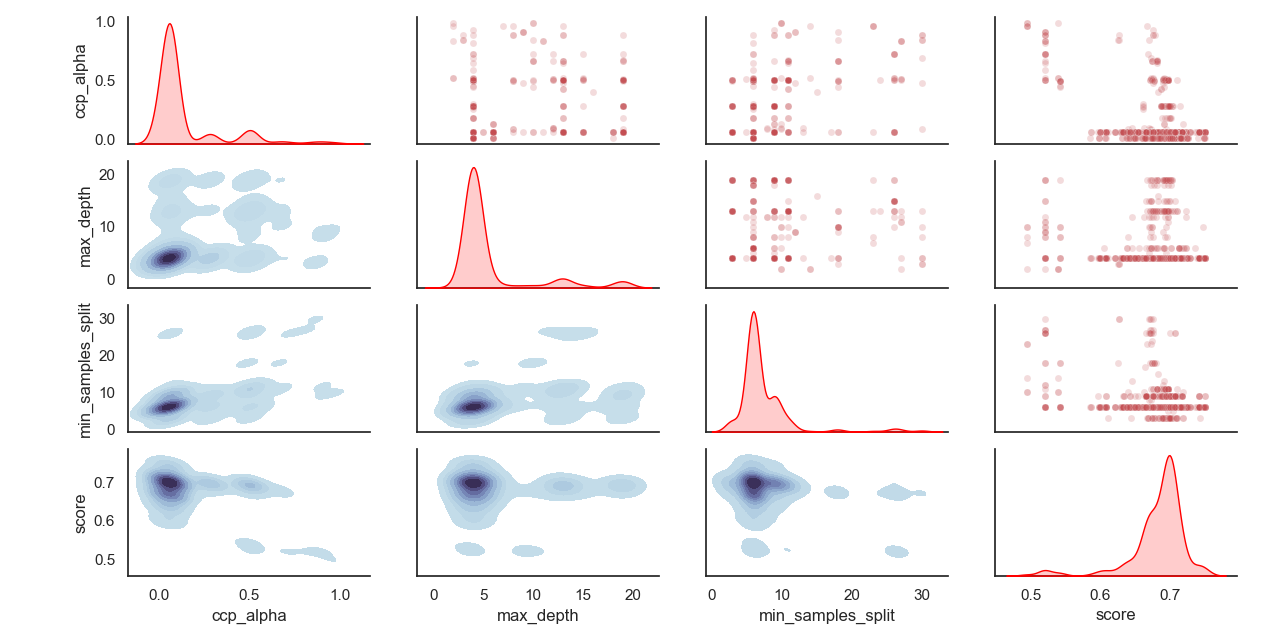
Abgesandte Verteilung von Hyperparametern:
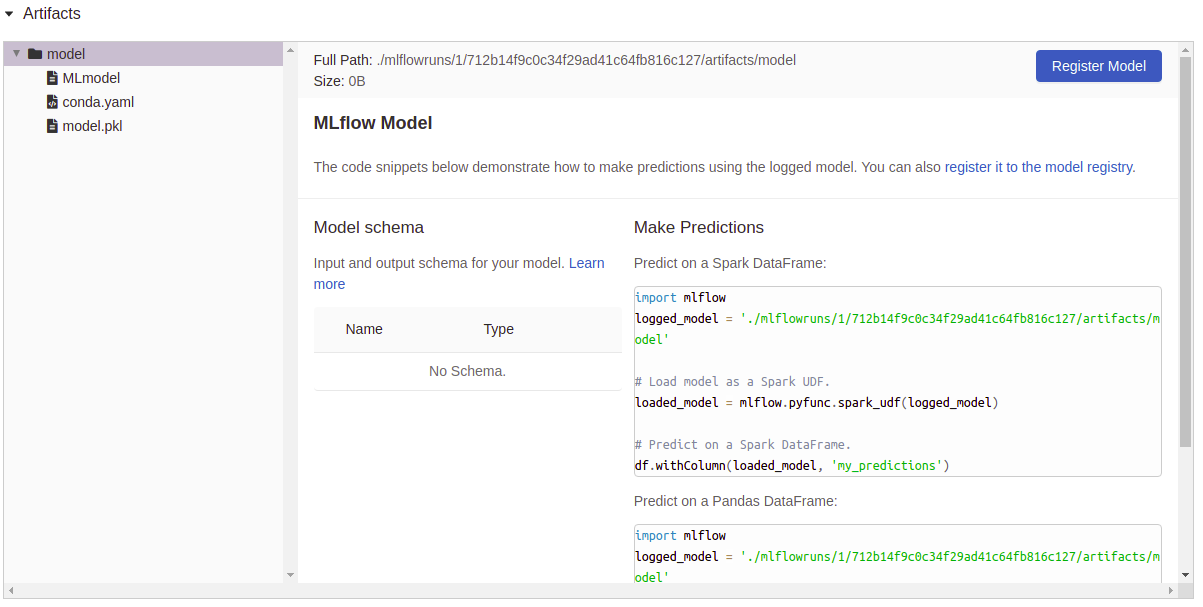
Artefakte Protokollierung:
Installieren Sie die Sklearn-Genetic-opt
Es wird empfohlen, Sklearn-Genetic mit einer virtuellen Umwelt innerhalb der Env-Verwendung zu installieren:
PIP Installieren Sie die Sklearn-Genetic-opt
Wenn Sie alle Funktionen abrufen möchten, einschließlich der Aufplotten-, Tensorboard- und MLFlow -Protokollierungsfunktionen, installieren Sie alle zusätzlichen Pakete:
PIP Installieren Sie die Sklearn-Genetic-opt [alle]
from sklearn_genetic import GASearchCV
from sklearn_genetic . space import Continuous , Categorical , Integer
from sklearn . ensemble import RandomForestClassifier
from sklearn . model_selection import train_test_split , StratifiedKFold
from sklearn . datasets import load_digits
from sklearn . metrics import accuracy_score
data = load_digits ()
n_samples = len ( data . images )
X = data . images . reshape (( n_samples , - 1 ))
y = data [ 'target' ]
X_train , X_test , y_train , y_test = train_test_split ( X , y , test_size = 0.33 , random_state = 42 )
clf = RandomForestClassifier ()
# Defines the possible values to search
param_grid = { 'min_weight_fraction_leaf' : Continuous ( 0.01 , 0.5 , distribution = 'log-uniform' ),
'bootstrap' : Categorical ([ True , False ]),
'max_depth' : Integer ( 2 , 30 ),
'max_leaf_nodes' : Integer ( 2 , 35 ),
'n_estimators' : Integer ( 100 , 300 )}
# Seed solutions
warm_start_configs = [
{ "min_weight_fraction_leaf" : 0.02 , "bootstrap" : True , "max_depth" : None , "n_estimators" : 100 },
{ "min_weight_fraction_leaf" : 0.4 , "bootstrap" : True , "max_depth" : 5 , "n_estimators" : 200 },
]
cv = StratifiedKFold ( n_splits = 3 , shuffle = True )
evolved_estimator = GASearchCV ( estimator = clf ,
cv = cv ,
scoring = 'accuracy' ,
population_size = 20 ,
generations = 35 ,
param_grid = param_grid ,
n_jobs = - 1 ,
verbose = True ,
use_cache = True ,
warm_start_configs = warm_start_configs ,
keep_top_k = 4 )
# Train and optimize the estimator
evolved_estimator . fit ( X_train , y_train )
# Best parameters found
print ( evolved_estimator . best_params_ )
# Use the model fitted with the best parameters
y_predict_ga = evolved_estimator . predict ( X_test )
print ( accuracy_score ( y_test , y_predict_ga ))
# Saved metadata for further analysis
print ( "Stats achieved in each generation: " , evolved_estimator . history )
print ( "Best k solutions: " , evolved_estimator . hof ) from sklearn_genetic import GAFeatureSelectionCV , ExponentialAdapter
from sklearn . model_selection import train_test_split
from sklearn . svm import SVC
from sklearn . datasets import load_iris
from sklearn . metrics import accuracy_score
import numpy as np
data = load_iris ()
X , y = data [ "data" ], data [ "target" ]
# Add random non-important features
noise = np . random . uniform ( 5 , 10 , size = ( X . shape [ 0 ], 5 ))
X = np . hstack (( X , noise ))
X_train , X_test , y_train , y_test = train_test_split ( X , y , test_size = 0.33 , random_state = 0 )
clf = SVC ( gamma = 'auto' )
mutation_scheduler = ExponentialAdapter ( 0.8 , 0.2 , 0.01 )
crossover_scheduler = ExponentialAdapter ( 0.2 , 0.8 , 0.01 )
evolved_estimator = GAFeatureSelectionCV (
estimator = clf ,
scoring = "accuracy" ,
population_size = 30 ,
generations = 20 ,
mutation_probability = mutation_scheduler ,
crossover_probability = crossover_scheduler ,
n_jobs = - 1 )
# Train and select the features
evolved_estimator . fit ( X_train , y_train )
# Features selected by the algorithm
features = evolved_estimator . support_
print ( features )
# Predict only with the subset of selected features
y_predict_ga = evolved_estimator . predict ( X_test )
print ( accuracy_score ( y_test , y_predict_ga ))
# Transform the original data to the selected features
X_reduced = evolved_estimator . transform ( X_test )Weitere Informationen zu den Änderungen der Sklearn-Genetic-opt finden Sie im Changelog
Sie können die neueste Entwicklungsversion mit dem Befehl überprüfen:
Git Clone https://github.com/rodrigo-arenas/sklearn-genetic-opt.git
Installieren Sie die Entwicklungsabhängigkeiten:
PIP install -r Dev -requirements.txt
Sehen Sie sich die neueste Dokumentation zur Entwicklung an: https://sklearn-genetic-opt.readthedocs.io/en/latest/
Beiträge sind mehr als willkommen! Das laufende Projekt gibt mehrere Möglichkeiten. Bitte setzen Sie sich mit uns in Verbindung, wenn Sie helfen möchten. Überprüfen Sie die aktuellen Probleme und auch den Beitragsanleitung.
Vielen Dank an die Leute, die bei diesem Projekt helfen!
Nach der Installation können Sie die Testsuite von außerhalb des Quellverzeichnisses starten:
PyTest Sklearn_Genetic