
Virheat adalah alat untuk memvisualisasikan VCFS sebagai mutasi heatmap dan peta ke gen masing -masing.
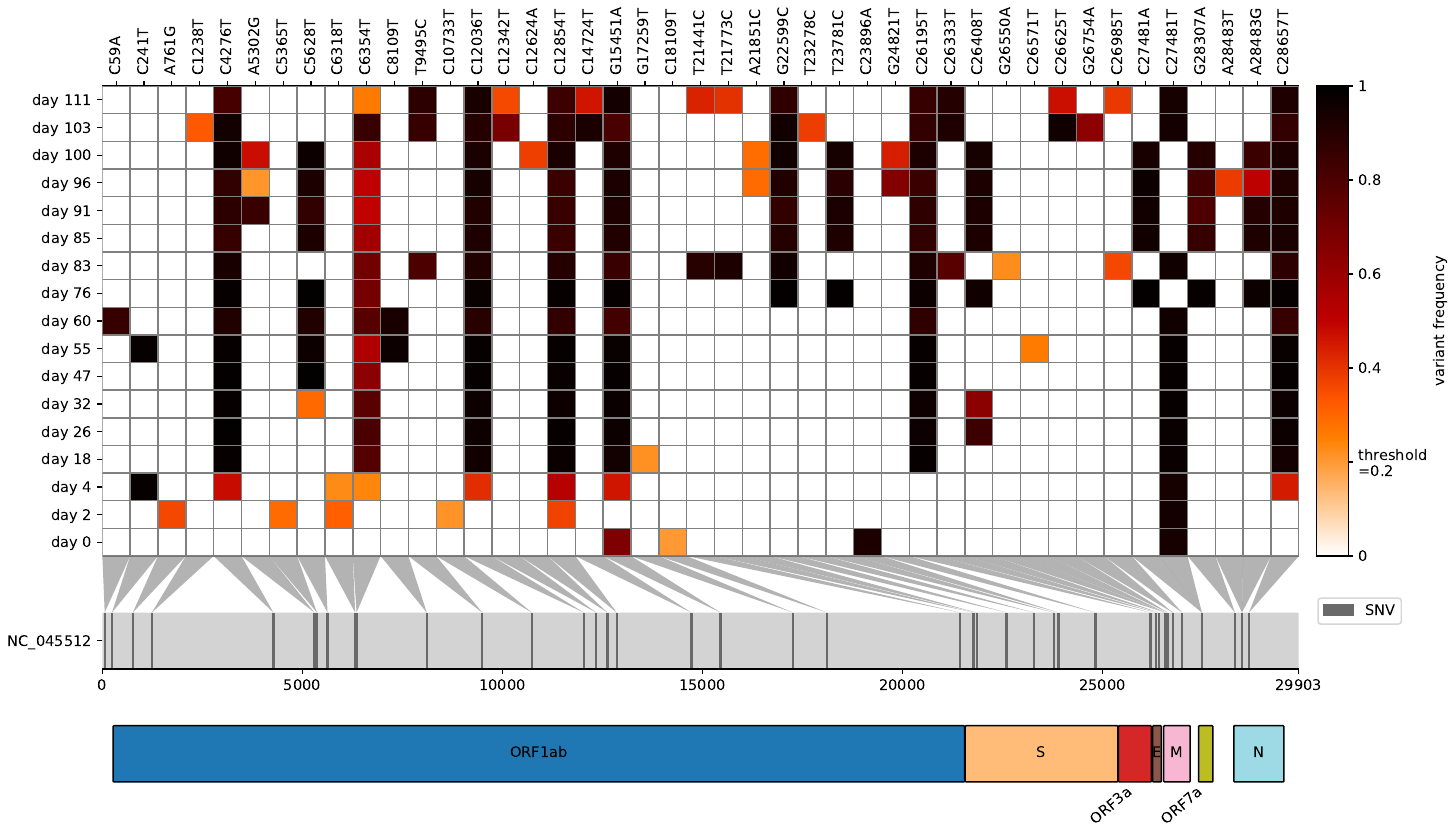
Pernah ingin melihat frekuensi varian yang kental setelah memetakan bacaan mentah Anda ke genom referensi virus/bakteri dan membandingkan beberapa file VCF secara bersamaan? Dari Virheat untuk Anda. Anda tidak hanya dapat memvisualisasikan heatmap tetapi juga membaca dalam file GFF3 yang memungkinkan Anda menampilkan gen yang menyimpan mutasi. Skrip ringan ini terinspirasi oleh Snipit dan plot frekuensi varian saya, mendapatkan fitur visualisasi terbaik dari keduanya.
--scores 
pip install virheatconda install -c bioconda virheatgit clone https://github.com/jonas-fuchs/virHEAT
cd virHEAT
pip install -r requirements.txt
# or
pip install .Itu sudah. Untuk memeriksa apakah itu berhasil:
virheat -vAnda harus melihat versi virheat saat ini.
usage: virheat < folder containing vcfs > < output dir > -l or -g [additional arguments]
Argumen:
positional arguments:
input folder containing input files and output folder
options:
-h, --help show this help message and exit
-r ref_id, --reference ref_id
reference identifier
--name virHEAT_plot.pdf
plot name and file type (pdf, png, svg, jpg). Default: virHEAT_plot.pdf
-l None, --genome-length None
length of the genome (needed if gff3 is not provided)
-g None, --gff3-path None
path to gff3 (needed if length is not provided)
-a [gene ...], --gff3-annotations [gene ...]
annotations to display from gff3 file (standard: gene). Multiple possible.
--gene-arrows, --no-gene-arrows
show genes in arrow format (only if the -g argument is provided) (default: False)
-t 0, --threshold 0 display frequencies above this threshold (0-1)
--delete, --no-delete
delete mutations that are present in all samples and their maximum frequency divergence is smaller than 0.5 (default: True)
-n None, --delete-n None
do not show mutations that occur n times or less (default: Do not delete)
-z start stop, --zoom start stop
restrict the plot to a specific genomic region.
--sort, --no-sort sort sample names alphanumerically (default: False)
--min-cov 20 display mutations covered at least x time (only if per base cov tsv files are provided)
-s scores_file pos_col score_col score_name, --scores scores_file pos_col score_col score_name
specify scores to be added to the plot by providing a CSV file containing scores, along with its column for amino-acid positions, its column for scores, and descriptive score names (e.g., expression, binding, antibody escape, etc.).
This option can be used multiple times to include multiple sets of scores.
-v, --version show program's version number and exit
Anda perlu memberikan panjang genom referensi Anda atau jika Anda ingin mendapatkan anotasi urutan yang Anda perlukan untuk menyediakan file GFF3.
Selain itu, Anda juga dapat menganalisis apakah mutasi cukup tertutup dan menampilkan sel-sel yang tidak tertutup dalam warna abu-abu. Untuk itu, buat file TSV cakupan per dasar untuk setiap file BAM dengan Qualimap dan berikan di folder yang sama dengan file VCF. Beri mereka nama yang sama dengan file VCF Anda.
Selain itu, ada opsi untuk memasukkan visualisasi skor tambahan (misalnya, skor MAVE untuk afinitas mengikat, tingkat ekspresi, pelarian antibodi, dll.) Yang dipetakan ke mutasi pada heatmap. Untuk memanfaatkan fitur ini, gunakan argumen -s atau - -scores, dan berikan argumen berikut: 1) jalur ke file CSV yang berisi skor; 2) nama kolom dalam file ini yang berisi posisi mutasi dalam notasi klasik (misalnya, T430Y); 3) nama kolom dalam file ini yang berisi skor itu sendiri; 4) Nama skor deskriptif yang akan digunakan sebagai label di plot. Beberapa set skor dapat dimasukkan secara bersamaan dengan mengulangi opsi -s atau - -scores dengan argumen yang berbeda. Misalnya input dan kemungkinan data output, silakan merujuk ke file yang terletak di folder example_data/example_mave_data.
Penafian penting: Kode ini berada di bawah lisensi GPLV3. Kode itu tanpa jaminan apa pun; bahkan tanpa jaminan tersirat dari dapat diperjualbelikan atau kebugaran untuk tujuan tertentu. Program ini adalah perangkat lunak gratis: Anda dapat mendistribusikannya kembali dan/atau memodifikasinya berdasarkan ketentuan lisensi publik umum GNU seperti yang diterbitkan oleh Yayasan Perangkat Lunak Gratis, baik versi 3 lisensi, atau (pada opsi Anda) versi selanjutnya.