
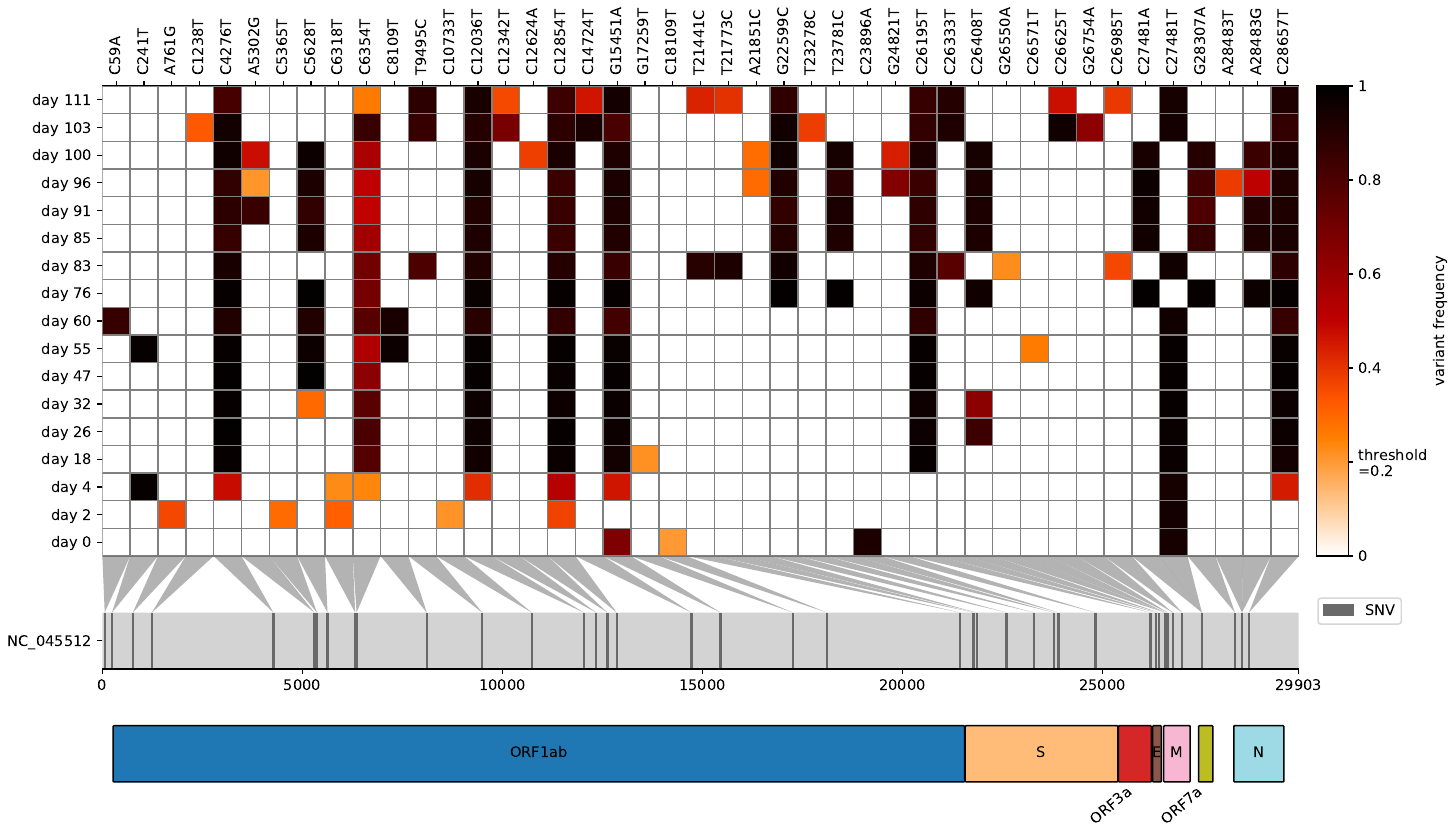
Virheat es una herramienta para visualizar VCFS como un mapa de calor y mapear mutaciones a los genes respectivos.
¿Alguna vez quiso tener una mirada condensada a las frecuencias de variantes después de mapear sus lecturas sin procesar con un genoma de referencia viral/bacteriana y comparar múltiples archivos VCF al mismo tiempo? Que Virheat es para ti. No solo puede visualizar el mapa de calor, sino también leer en un archivo GFF3 que le permite mostrar genes que albergan una mutación. Este guión liviano se inspiró en Snipit y mi trama de frecuencia variante, obteniendo las mejores características de visualización de ambas.
--scores 
pip install virheatconda install -c bioconda virheatgit clone https://github.com/jonas-fuchs/virHEAT
cd virHEAT
pip install -r requirements.txt
# or
pip install .Eso ya era todo. Para verificar si funcionó:
virheat -vDebería ver la versión actual de Virheat.
usage: virheat < folder containing vcfs > < output dir > -l or -g [additional arguments]
Argumentos:
positional arguments:
input folder containing input files and output folder
options:
-h, --help show this help message and exit
-r ref_id, --reference ref_id
reference identifier
--name virHEAT_plot.pdf
plot name and file type (pdf, png, svg, jpg). Default: virHEAT_plot.pdf
-l None, --genome-length None
length of the genome (needed if gff3 is not provided)
-g None, --gff3-path None
path to gff3 (needed if length is not provided)
-a [gene ...], --gff3-annotations [gene ...]
annotations to display from gff3 file (standard: gene). Multiple possible.
--gene-arrows, --no-gene-arrows
show genes in arrow format (only if the -g argument is provided) (default: False)
-t 0, --threshold 0 display frequencies above this threshold (0-1)
--delete, --no-delete
delete mutations that are present in all samples and their maximum frequency divergence is smaller than 0.5 (default: True)
-n None, --delete-n None
do not show mutations that occur n times or less (default: Do not delete)
-z start stop, --zoom start stop
restrict the plot to a specific genomic region.
--sort, --no-sort sort sample names alphanumerically (default: False)
--min-cov 20 display mutations covered at least x time (only if per base cov tsv files are provided)
-s scores_file pos_col score_col score_name, --scores scores_file pos_col score_col score_name
specify scores to be added to the plot by providing a CSV file containing scores, along with its column for amino-acid positions, its column for scores, and descriptive score names (e.g., expression, binding, antibody escape, etc.).
This option can be used multiple times to include multiple sets of scores.
-v, --version show program's version number and exit
Debe proporcionar la longitud de su genoma de referencia o si desea obtener la anotación de secuencia, deberá proporcionar el archivo GFF3.
Además, también puede analizar si las mutaciones están suficientemente cubiertas y muestran células no cubiertas en gris. Para eso, primero cree un archivo TSV de cobertura por base para cada archivo BAM con QualImap y proporcione en la misma carpeta que los archivos VCF. Déles el mismo nombre que sus archivos VCF.
Además, existe una opción para incluir visualizaciones de puntajes adicionales (p. Ej., Puntuaciones de mazos para afinidad de unión, nivel de expresión, escape de anticuerpos, etc.) asignadas a mutaciones en el mapa de calor. Para utilizar esta característica, use el argumento -s o --corsos, y proporcione los siguientes argumentos: 1) ruta al archivo CSV que contiene puntajes; 2) el nombre de la columna en este archivo que contiene posiciones de mutación en la notación clásica (por ejemplo, T430Y); 3) el nombre de la columna en este archivo que contiene los puntajes en sí mismo; 4) Un nombre de puntaje descriptivo que se utilizará como etiquetas en la trama. Se pueden incluir múltiples conjuntos de puntajes simultáneamente repitiendo la opción -s o -cora con diferentes argumentos. Por ejemplo, la entrada y los posibles datos de salida, consulte los archivos ubicados en la carpeta Ejemplo_Data/Ejemplo_Mave_Data.
Descargo de responsabilidad importante: el código está bajo la licencia GPLV3. El código no tiene ninguna garantía; Sin siquiera la garantía implícita de comerciabilidad o estado físico para un propósito particular. Este programa es un software gratuito: puede redistribuirlo y/o modificarlo bajo los términos de la Licencia Pública General de GNU publicada por Free Software Foundation, ya sea la versión 3 de la licencia o (a su opción) cualquier versión posterior.