
Virheat هي أداة لتصور VCFs كطفرات خريطة الحرارة وخريطة الخريطة للجينات المعنية.
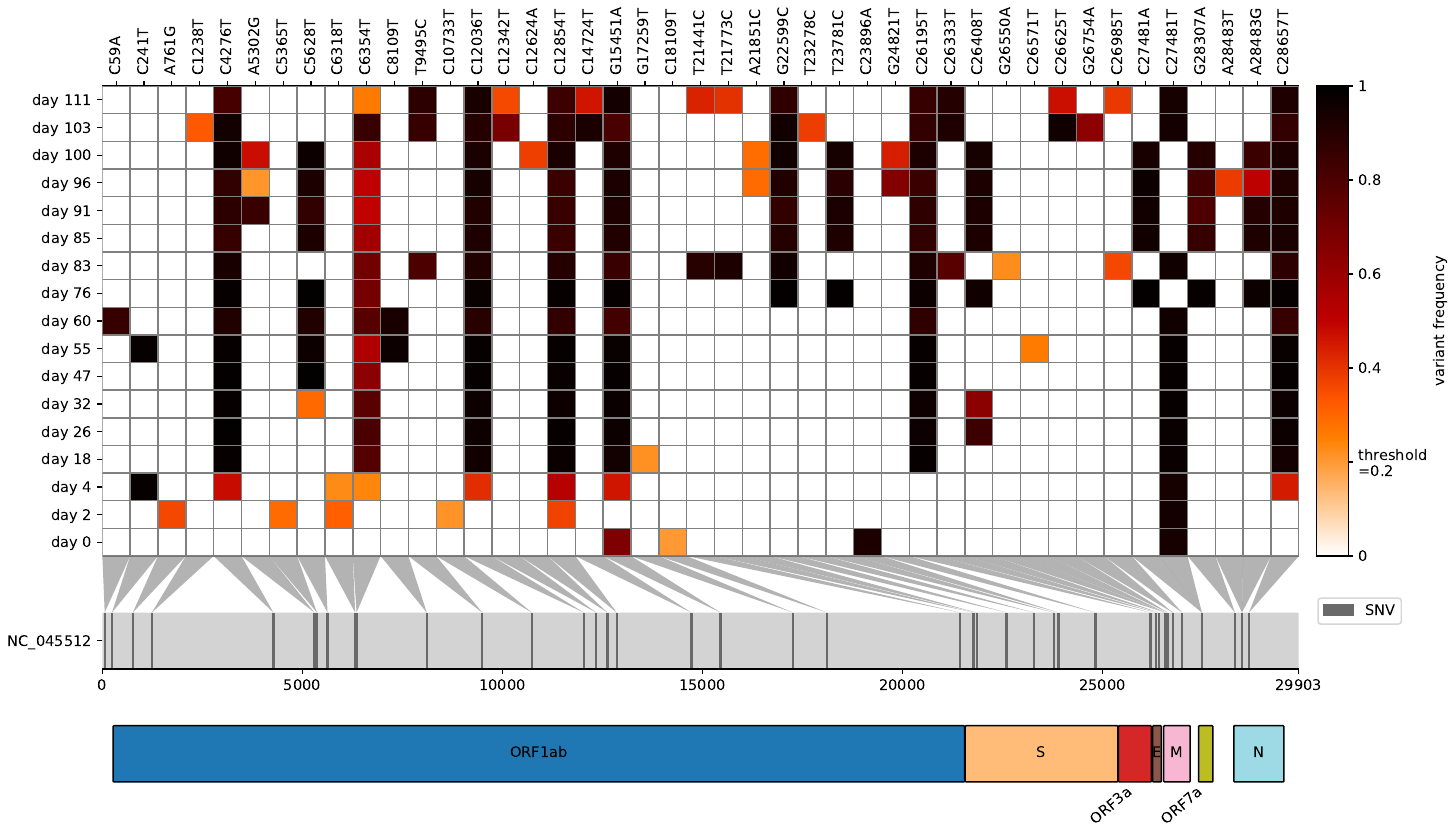
هل أرادت يومًا أن تتم إلقاء نظرة مكثفة على الترددات المتغيرة بعد تعيين قراءاتك الخام إلى جينوم مرجعي فيروسي/بكتيري ومقارنة ملفات VCF متعددة في نفس الوقت؟ من Virheat لك. لا يمكنك فقط تصور خريطة الحرارة ولكن أيضًا القراءة في ملف GFF3 الذي يتيح لك عرض الجينات التي تؤوي طفرة. استلهم هذا البرنامج النصي خفيف الوزن من Snipit ومؤامرة التردد المتنوعة الخاصة بي ، وحصل على أفضل ميزات التصور لكليهما.
--scores 
pip install virheatconda install -c bioconda virheatgit clone https://github.com/jonas-fuchs/virHEAT
cd virHEAT
pip install -r requirements.txt
# or
pip install .كان ذلك بالفعل. للتحقق مما إذا كان يعمل:
virheat -vيجب أن ترى نسخة virheat الحالية.
usage: virheat < folder containing vcfs > < output dir > -l or -g [additional arguments]
الحجج:
positional arguments:
input folder containing input files and output folder
options:
-h, --help show this help message and exit
-r ref_id, --reference ref_id
reference identifier
--name virHEAT_plot.pdf
plot name and file type (pdf, png, svg, jpg). Default: virHEAT_plot.pdf
-l None, --genome-length None
length of the genome (needed if gff3 is not provided)
-g None, --gff3-path None
path to gff3 (needed if length is not provided)
-a [gene ...], --gff3-annotations [gene ...]
annotations to display from gff3 file (standard: gene). Multiple possible.
--gene-arrows, --no-gene-arrows
show genes in arrow format (only if the -g argument is provided) (default: False)
-t 0, --threshold 0 display frequencies above this threshold (0-1)
--delete, --no-delete
delete mutations that are present in all samples and their maximum frequency divergence is smaller than 0.5 (default: True)
-n None, --delete-n None
do not show mutations that occur n times or less (default: Do not delete)
-z start stop, --zoom start stop
restrict the plot to a specific genomic region.
--sort, --no-sort sort sample names alphanumerically (default: False)
--min-cov 20 display mutations covered at least x time (only if per base cov tsv files are provided)
-s scores_file pos_col score_col score_name, --scores scores_file pos_col score_col score_name
specify scores to be added to the plot by providing a CSV file containing scores, along with its column for amino-acid positions, its column for scores, and descriptive score names (e.g., expression, binding, antibody escape, etc.).
This option can be used multiple times to include multiple sets of scores.
-v, --version show program's version number and exit
تحتاج إما إلى توفير طول الجينوم المرجعي أو إذا كنت ترغب في الحصول على شرح التسلسل ، فستحتاج إلى توفير ملف GFF3.
بالإضافة إلى ذلك ، يمكنك أيضًا تحليل ما إذا كانت الطفرات مغطاة بشكل كاف وعرض خلايا غير مغطاة باللون الرمادي. لذلك ، قم أولاً بإنشاء ملفات TSV لكل تغطية قاعدة لكل ملف BAM مع QUALIMAP وقم بتوفيره في نفس المجلد مثل ملفات VCF. امنحهم نفس اسم ملفات VCF الخاصة بك.
علاوة على ذلك ، هناك خيار لتضمين تصورات الدرجات الإضافية (على سبيل المثال ، درجات Mave لارتباط الربط ، مستوى التعبير ، هروب الأجسام المضادة ، إلخ) المعينة على الطفرات على خريطة الحرارة. لاستخدام هذه الميزة ، استخدم وسيطة -S أو -الدرجات ، وتوفير الوسائط التالية: 1) المسار إلى ملف CSV يحتوي على درجات ؛ 2) اسم العمود في هذا الملف الذي يحتوي على مواضع طفرة في التدوين الكلاسيكي (على سبيل المثال ، T430Y) ؛ 3) اسم العمود في هذا الملف الذي يحتوي على الدرجات نفسها ؛ 4) اسم النتيجة الوصفي الذي سيتم استخدامه كعلامات في المؤامرة. يمكن تضمين مجموعات نقاط متعددة في وقت واحد عن طريق تكرار خيار -S أو -الدرجات مع وسيطات مختلفة. على سبيل المثال الإدخال وبيانات الإخراج المحتملة ، يرجى الرجوع إلى الملفات الموجودة في مجلد Imexame_Data/example_mave_data.
إخلاء المسؤولية المهم: الكود تحت رخصة GPLV3. الكود بدون أي ضمان ؛ بدون حتى الضمان الضمني للتسويق أو اللياقة لغرض معين. هذا البرنامج هو برنامج مجاني: يمكنك إعادة توزيعه و/أو تعديله بموجب شروط رخصة GNU العامة العامة كما تم نشرها من قبل مؤسسة البرمجيات المجانية ، إما الإصدار 3 من الترخيص ، أو (في خيارك) أي إصدار لاحق.