
Environnement de développement Web tout-en-un pour l'apprentissage automatique
Démarrage • Fonctionnalités et captures d'écran • Support • Signaler un bogue • FAQ • Problèmes connus • Contribution
L'espace de travail ML est un IDE Web tout-en-un spécialisé pour l'apprentissage automatique et la science des données. Il est simple à déployer et vous fait démarrer en quelques minutes pour construire des solutions ML de manière productive sur vos propres machines. Cet espace de travail est l'outil ultime pour les développeurs préchargés avec une variété de bibliothèques de science des données populaires (par exemple, Tensorflow, Pytorch, Keras, Sklearn) et Dev Tools (par exemple, Jupyter, VS Code, Tensorboard) parfaitement configuré, optimisée et intégrée.
L'espace de travail nécessite que Docker soit installé sur votre machine (guide d'installation).
Le déploiement d'une instance d'espace de travail unique est aussi simple que:
docker run -p 8080:8080 mltooling/ml-workspace:0.13.2Voilà, c'était facile! Maintenant, Docker tirera la dernière image de l'espace de travail sur votre machine. Cela peut prendre quelques minutes, selon votre vitesse Internet. Une fois l'espace de travail démarré, vous pouvez y accéder via http: // localhost: 8080.
Si vous êtes démarré sur une autre machine ou avec un autre port, assurez-vous d'utiliser l'IP / DNS de la machine et / ou le port exposé.
Pour déployer une seule instance pour une utilisation productive, nous vous recommandons d'appliquer au moins les options suivantes:
docker run -d
-p 8080:8080
--name " ml-workspace "
-v " ${PWD} :/workspace "
--env AUTHENTICATE_VIA_JUPYTER= " mytoken "
--shm-size 512m
--restart always
mltooling/ml-workspace:0.13.2 Cette commande exécute le conteneur en arrière-plan ( -d ), monte votre répertoire de travail actuel dans le dossier /workspace ( -v ), sécurise l'espace de travail via un jeton fourni ( --env AUTHENTICATE_VIA_JUPYTER ), fournit 512Mb de mémoire partagée ( --shm-size ) pour empêcher les accidents inattendus (voir les problèmes connus), et maintient le conteneur des conteneurs, même sur les repos du système ( --restart always ). Vous pouvez trouver des options supplémentaires pour Docker Exécuter ici et des options de configuration de l'espace de travail dans la section ci-dessous.
L'espace de travail offre une variété d'options de configuration qui peuvent être utilisées en définissant des variables d'environnement (via Docker Run Option: --env ).
| Variable | Description | Défaut |
|---|---|---|
| Workspace_base_url | L'URL de base sous laquelle Jupyter et tous les autres outils seront accessibles. | / / |
| Workspace_ssl_enabled | Activer ou désactiver SSL. Lorsqu'il est défini sur true, le certificat (cert.crt) doit être monté sur /resources/ssl ou, sinon, le conteneur génère un certificat auto-signé. | FAUX |
| Workspace_auth_user | Nom d'utilisateur de base Auth. Pour activer Basic Auth, l'utilisateur et le mot de passe doivent être définis. Nous vous recommandons d'utiliser l' AUTHENTICATE_VIA_JUPYTER pour sécuriser l'espace de travail. | |
| Workspace_auth_password | Mot de passe utilisateur de base AUTH. Pour activer Basic Auth, l'utilisateur et le mot de passe doivent être définis. Nous vous recommandons d'utiliser l' AUTHENTICATE_VIA_JUPYTER pour sécuriser l'espace de travail. | |
| Workspace_port | Configure le port interne à conteneur principal du proxy de l'espace de travail. Pour la plupart des scénarios, cette configuration ne doit pas être modifiée et la configuration du port via Docker doit être utilisée à la place de l'espace de travail doit être accessible à partir d'un port différent. | 8080 |
| Config_backup_enabled | Sauvegarder et restaurer automatiquement la configuration de l'utilisateur dans le dossier persistant /workspace , tels que le .ssh, .jupyter ou .gitconfig du répertoire domestique des utilisateurs. | vrai |
| Shared_links_enabled | Activer ou désactiver la capacité de partager des ressources via des liens externes. Ceci est utilisé pour activer le partage de fichiers, l'accès aux ports internes de l'espace de travail et la configuration SSH basée sur les commandes. Tous les liens partagés sont protégés via un jeton. Cependant, il existe certains risques car le jeton ne peut pas être facilement invalidé après le partage et n'expire pas. | vrai |
| Include_tutorial | Si true , une sélection de cahiers de tutoriel et d'introduction est ajouté au dossier /workspace au démarrage du conteneur, mais seulement si le dossier est vide. | vrai |
| Max_num_threads | Le nombre de threads utilisés pour les calculs lors de l'utilisation de diverses bibliothèques communes (MKL, OpenBlas, OMP, Numba, ...). Vous pouvez également utiliser auto pour permettre à l'espace de travail de déterminer dynamiquement le nombre de threads en fonction des ressources CPU disponibles. Cette configuration peut être écrasée par l'utilisateur à partir de l'espace de travail. Généralement, il est bon de le définir ou en dessous du nombre de processeurs disponibles pour l'espace de travail. | auto |
| Configuration de Jupyter: | ||
| Shutdown_inactive_kernels | Arrêtez automatiquement les noyaux inactifs après un délai d'attente donné (pour nettoyer la mémoire ou les ressources GPU). La valeur peut être un délai d'expiration en secondes ou défini sur true avec une valeur par défaut de 48h. | FAUX |
| Authenticiate_via_jupyter | Si true , toutes les demandes HTTP seront authentifiées contre le serveur Jupyter, ce qui signifie que la méthode d'authentification configurée avec Jupyter sera également utilisée pour tous les autres outils. Cela peut être désactivé avec false . Toute autre valeur activera cette authentification et sera appliquée sous forme de jeton via NotebookApp.Token Configuration de Jupyter. | FAUX |
| Cahier_args | Ajouter et écraser les options de configuration du jupyter via des args de ligne de commande. Reportez-vous à cet aperçu de toutes les options. | |
Pour persister les données, vous devez monter un volume dans /workspace (via Docker Run Option: -v ).
Le répertoire de travail par défaut dans le conteneur est /workspace , qui est également le répertoire racine de l'instance de jupyter. Le répertoire /workspace est destiné à être utilisé pour tous les artefacts de travail importants. Les données dans d'autres répertoires du serveur (par exemple, /root ) peuvent se perdre lors des redémarrages des conteneurs.
Nous recommandons fortement d'activer l'authentification via l'une des deux options suivantes. Pour les deux options, l'utilisateur devra s'authentifier pour accéder à l'un des outils préinstallés.
L'authentification ne fonctionne que pour tous les outils accessibles via le port d'espace de travail principal (par défaut:
8080). Cela fonctionne pour tous les outils préinstallés et la fonction de ports d'accès. Si vous exposez un autre port du conteneur, assurez-vous de le sécuriser avec l'authentification également!
Activez l'authentification basée sur les jetons en fonction de l'implémentation d'authentification de Jupyter via la variable AUTHENTICATE_VIA_JUPYTER :
docker run -p 8080:8080 --env AUTHENTICATE_VIA_JUPYTER= " mytoken " mltooling/ml-workspace:0.13.2 Vous pouvez également utiliser <generated> pour permettre à Jupyter de générer un jeton aléatoire qui est imprimé sur les journaux de conteneurs. Une valeur de true ne définira aucun jeton mais activera que chaque demande à un outil de l'espace de travail sera vérifiée avec l'instance de jupyter si l'utilisateur est authentifié. Ceci est utilisé pour des outils comme JupyterHub, qui configure sa propre façon d'authentification.
Activez l'authentification de base via la variable WORKSPACE_AUTH_USER et WORKSPACE_AUTH_PASSWORD :
docker run -p 8080:8080 --env WORKSPACE_AUTH_USER= " user " --env WORKSPACE_AUTH_PASSWORD= " pwd " mltooling/ml-workspace:0.13.2 L'authentification de base est configurée via le proxy Nginx et pourrait être plus performante par rapport à l'autre option, car avec AUTHENTICATE_VIA_JUPYTER chaque demande à tout outil de l'espace de travail vérifiera via l'instance de jupyter si l'utilisateur (basé sur les cookies de demande) est authentifié.
Nous vous recommandons d'activer SSL afin que l'espace de travail soit accessible via HTTPS (communication chiffrée). Le cryptage SSL peut être activé via la variable WORKSPACE_SSL_ENABLED .
Lorsqu'elle est définie sur true , le fichier cert.crt et cert.key doit être monté sur /resources/ssl ou, si les fichiers de certificat n'existent pas, le conteneur génère des certificats auto-signés. Par exemple, si le /path/with/certificate/files sur le système local contient un certificat valide pour le domaine hôte ( cert.crt et cert.key FICHIER), il peut être utilisé à partir de l'espace de travail comme indiqué ci-dessous:
docker run
-p 8080:8080
--env WORKSPACE_SSL_ENABLED= " true "
-v /path/with/certificate/files:/resources/ssl:ro
mltooling/ml-workspace:0.13.2 Si vous souhaitez héberger l'espace de travail sur un domaine public, nous vous recommandons d'utiliser Let's Encrypt pour obtenir un certificat de confiance pour votre domaine. Pour utiliser le certificat généré (par exemple, via l'outil certitbot) pour l'espace de travail, le privkey.pem correspond au fichier cert.key et au fichier fullchain.pem au fichier cert.crt .
Lorsque vous activez la prise en charge SSL, vous devez accéder à l'espace de travail sur
https://, pas surhttp://.
Par défaut, le conteneur d'espace de travail n'a pas de contraintes de ressources et peut utiliser autant de ressources donné que le planificateur de noyau de l'hôte le permet. Docker fournit des moyens de contrôler la quantité de mémoire, ou CPU, un conteneur peut utiliser, en définissant des drapeaux de configuration d'exécution de la commande docker run.
L'espace de travail nécessite au moins 2 CPU et 500 Mo pour fonctionner stable et être utilisable.
Par exemple, la commande suivante restreint l'espace de travail à utiliser uniquement un maximum de 8 processeurs, 16 Go de mémoire et 1 Go de mémoire partagée (voir des problèmes connus):
docker run -p 8080:8080 --cpus=8 --memory=16g --shm-size=1G mltooling/ml-workspace:0.13.2Pour plus d'options et de documentation sur les contraintes de ressources, veuillez vous référer au guide officiel Docker.
Si un proxy est requis, vous pouvez passer la configuration de proxy via les variables d'environnement HTTP_PROXY , HTTPS_PROXY et NO_PROXY .
En plus de l'image principale de l'espace de travail ( mltooling/ml-workspace ), nous fournissons d'autres saveurs d'image qui étendent les fonctionnalités ou minimisent la taille de l'image pour prendre en charge une variété de cas d'utilisation.
La saveur minimale ( mltooling/ml-workspace-minimal ) est notre plus petite image qui contient la plupart des outils et fonctionnalités décrits dans la section des fonctionnalités sans la plupart des bibliothèques Python qui sont préinstallées dans notre image principale. Toute bibliothèque Python ou outil exclu peut être installée manuellement pendant l'exécution par l'utilisateur.
docker run -p 8080:8080 mltooling/ml-workspace-minimal:0.13.2 La saveur R ( mltooling/ml-workspace-r ) est basée sur notre image d'espace de travail par défaut et l'étend avec le noyau R-Interpreter, R-Jupyter, le serveur RStudio (accès via Open Tool -> RStudio ) et une variété de colis populaires à partir de l'écosystème R.
docker run -p 8080:8080 mltooling/ml-workspace-r:0.12.1 La saveur de Spark ( mltooling/ml-workspace-spark ) est basée sur notre image d'espace de travail R-Florn et l'étend avec le noyau Spark-Spark-Jupyter, Zeppelin Notebook (Access via Open Tool -> Zeppelin ), Pyspark, Hadoop, Java Kernel et quelques bibliothèques supplémentaires et jupyter extensions.
docker run -p 8080:8080 mltooling/ml-workspace-spark:0.12.1Actuellement, la saveur GPU ne prend en charge que CUDA 11.2. Un support pour d'autres versions CUDA pourrait être ajouté à l'avenir.
La saveur du GPU ( mltooling/ml-workspace-gpu ) est basée sur notre image d'espace de travail par défaut et l'étend avec les versions CUDA 10.1 et pratiquées par GPU de diverses bibliothèques d'apprentissage automatique (par exemple, Tensorflow, Pytorch, CNTK, Jax). Cette image GPU a les exigences supplémentaires suivantes pour le système:
>=460.32.03 (instructions).docker run -p 8080:8080 --gpus all mltooling/ml-workspace-gpu:0.13.2docker run -p 8080:8080 --runtime nvidia --env NVIDIA_VISIBLE_DEVICES= " all " mltooling/ml-workspace-gpu:0.13.2La saveur du GPU est également livrée avec quelques options de configuration supplémentaires, comme expliqué ci-dessous:
| Variable | Description | Défaut |
|---|---|---|
| Nvidia_visible_devices | Contrôle les GPU accessibles à l'intérieur de l'espace de travail. Par défaut, tous les GPU de l'hôte sont accessibles dans l'espace de travail. Vous pouvez soit utiliser all , none ou spécifier une liste séparée par des virgules des ID de périphérique (par exemple, 0,1 ). Vous pouvez trouver la liste des ID de périphérique disponibles en exécutant nvidia-smi sur la machine hôte. | tous |
| Cuda_visible_devices | Les contrôles des applications GPUS CUDA exécutées à l'intérieur de l'espace de travail verront. Par défaut, tous les GPU auxquels l'espace de travail a accès sera visible. Pour restreindre les applications, fournissez une liste séparée par des virgules des ID de périphérique interne (par exemple, 0,2 ) en fonction des appareils disponibles dans l'espace de travail (exécutez nvidia-smi ). Par rapport à NVIDIA_VISIBLE_DEVICES , l'utilisateur de l'espace de travail pourra toujours accéder à d'autres GPU en écrasant cette configuration à partir de l'espace de travail. | |
| Tf_force_gpu_allow_growth | Par défaut, la majorité de la mémoire GPU sera allouée par la première exécution d'un graphique TensorFlow. Bien que ce comportement puisse être souhaitable pour les pipelines de production, il est moins souhaitable pour une utilisation interactive. Utilisez true pour activer l'allocation de mémoire GPU dynamique ou false pour instruire TensorFlow pour allouer toute la mémoire lors de l'exécution. | vrai |
L'espace de travail est conçu comme un environnement de développement à utilisateur unique. Pour une configuration multi-utilisateurs, nous recommandons le déploiement? ML Hub. ML Hub est basé sur JupyterHub avec la tâche pour engendrer, gérer et proxy Workspace Workspace pour plusieurs utilisateurs.
ML Hub facilite la configuration d'un environnement multi-utilisateurs sur un seul serveur (via Docker) ou un cluster (via Kubernetes) et prend en charge une variété de scénarios d'utilisation et de fournisseurs d'authentification. Vous pouvez essayer ML Hub via:
docker run -p 8080:8080 -v /var/run/docker.sock:/var/run/docker.sock mltooling/ml-hub:latestPour plus d'informations et de documentation sur ML Hub, veuillez consulter le site GitHub.
Ce projet est maintenu par Benjamin Räthlein, Lukas Masuch et Jan Kalkan. Veuillez comprendre que nous ne pourrons pas fournir un support individuel par e-mail. Nous pensons également que l'aide est beaucoup plus précieuse si elle est partagée publiquement afin que davantage de personnes puissent en bénéficier.
| Taper | Canal |
|---|---|
| Rapports de bogues | |
| ? Demandes de fonctionnalités | |
| ? Questions d'utilisation | |
| ? Annonces | |
| ❓ Autres demandes |
Jupyter • GUI de bureau • Code vs • JupyterLab • Intégration GIT • Partage de fichiers • Ports d'accès • Tensorboard • Extensibilité • Surveillance matérielle • Accès SSH • Développement à distance • Exécution du travail
L'espace de travail est équipé d'une sélection d'outils de développement open source de classe les meilleurs pour aider avec le flux de travail d'apprentissage automatique. Beaucoup de ces outils peuvent être démarrés à partir du menu Open Tool à partir de Jupyter (l'application principale de l'espace de travail):
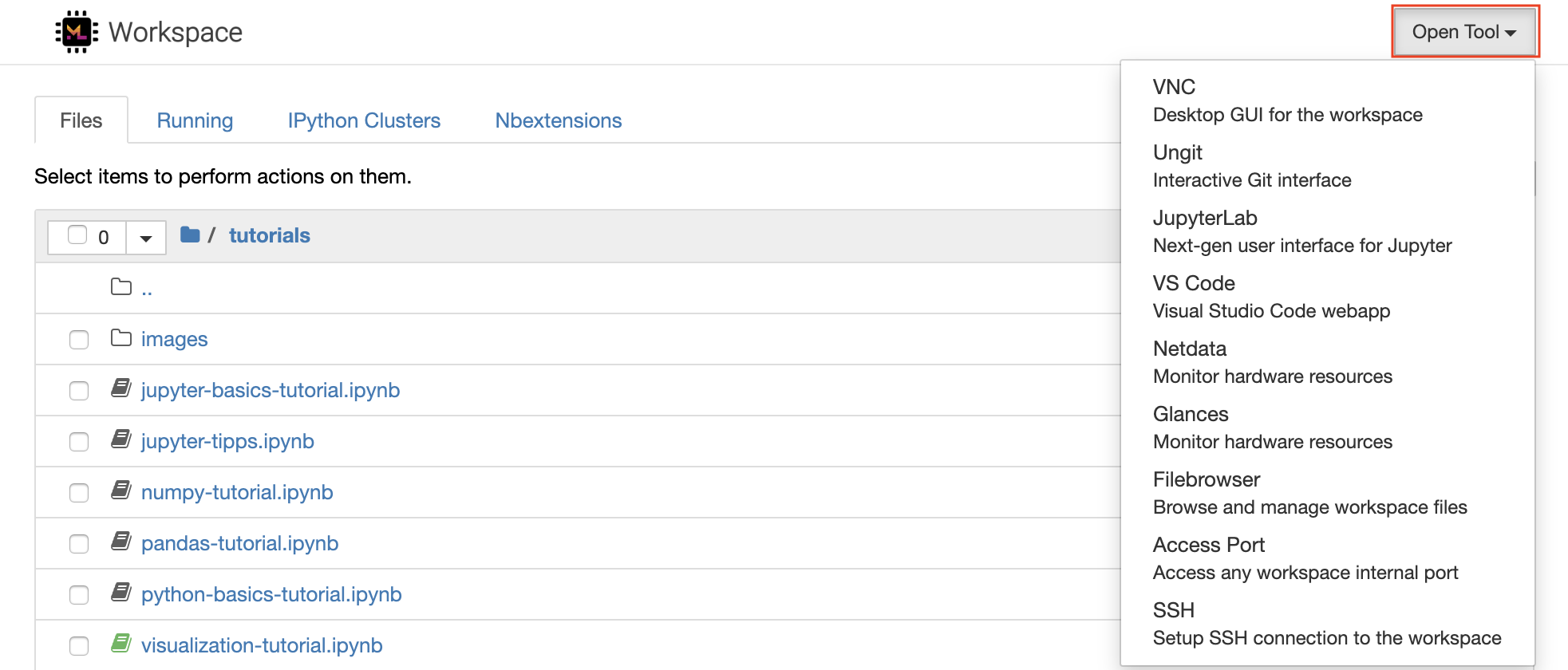
Dans votre espace de travail, vous avez des privilèges Root & Sudo complets pour installer n'importe quelle bibliothèque ou outil dont vous avez besoin via Terminal (par exemple,
pip,apt-get,condaounpm). Vous pouvez trouver plus de façons d'étendre l'espace de travail dans la section Extensibilité
Jupyter Notebook est un environnement interactif basé sur le Web pour l'écriture et l'exécution de code. Les principaux éléments constitutifs de Jupyter sont le navigateur de fichiers, l'éditeur de carnet et les noyaux. Le File-Browser fournit un gestionnaire de fichiers interactif pour tous les ordinateurs portables, fichiers et dossiers du répertoire /workspace .
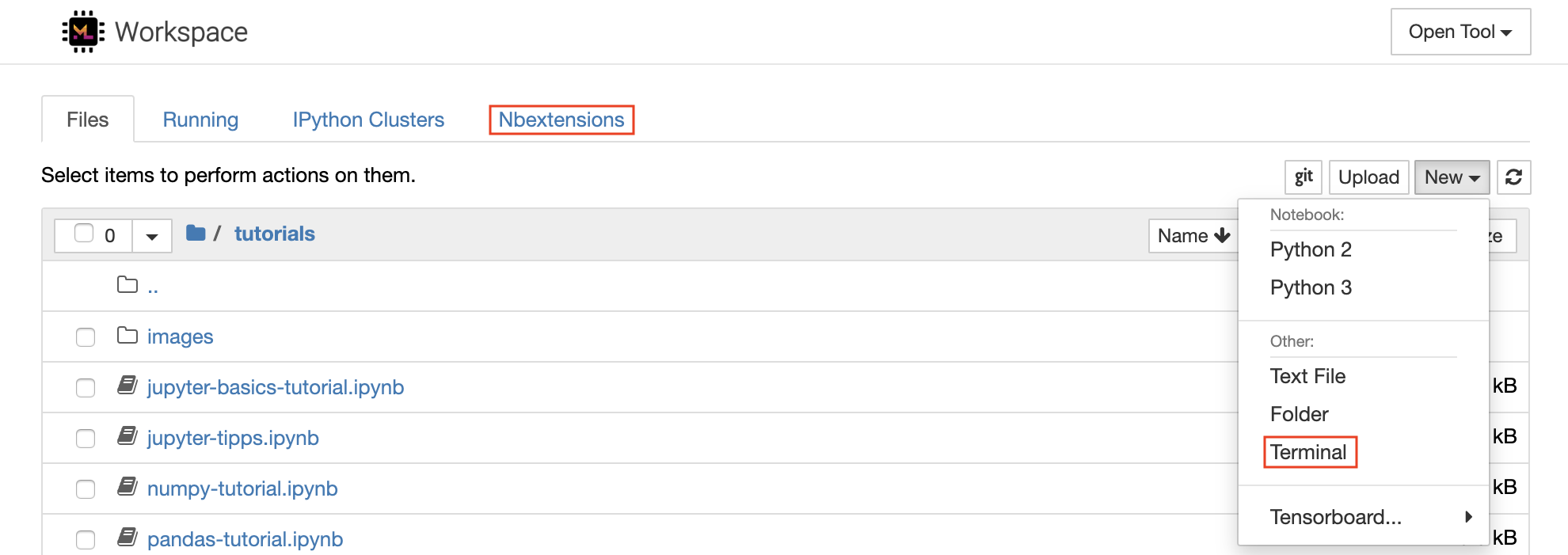
Un nouveau cahier peut être créé en cliquant sur le New bouton déroulant en haut de la liste et en sélectionnant le noyau de langue souhaité.
Vous pouvez également engendrer des instances de terminal interactif en sélectionnant
New -> Terminaldans le navigateur de fichiers.
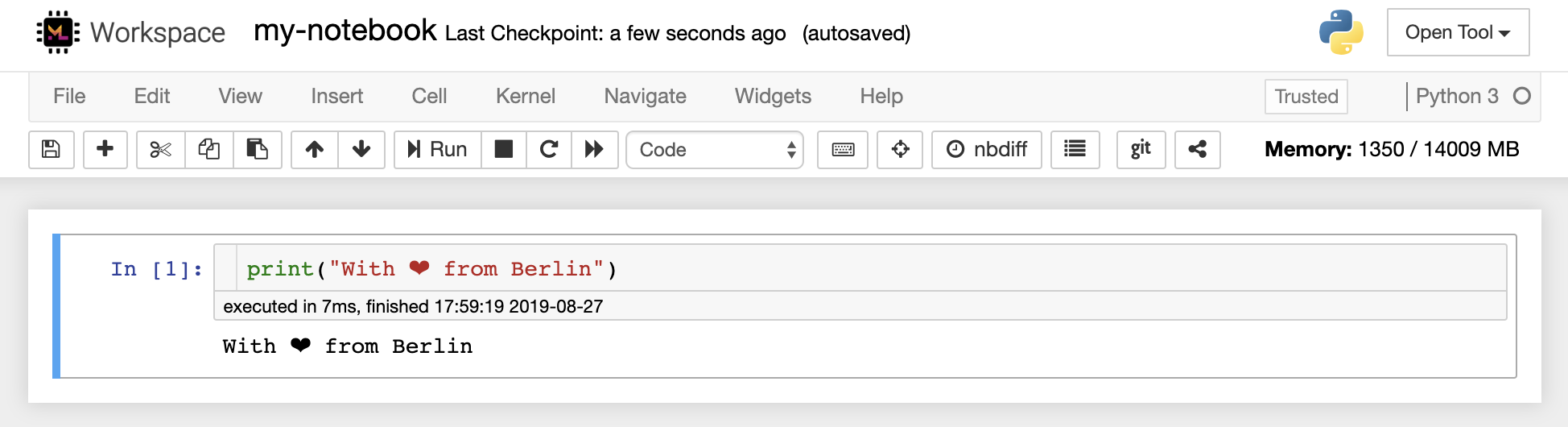
L'éditeur de carnet permet aux utilisateurs d'auteur des documents qui incluent le code en direct, le texte Markdown, les commandes de shell, les équations de latex, les widgets interactifs, les parcelles et les images. Ces documents de carnet fournissent un enregistrement complet et autonome d'un calcul qui peut être converti en divers formats et partagé avec d'autres.
Cet espace de travail a une variété d' extensions de jupyter tierces activées. Vous pouvez configurer ces extensions dans l'onglet NBExtensions Configurator:
nbextensionssur le navigateur de fichier
Le cahier permet d'exécuter le code dans une gamme de différents langages de programmation. Pour chaque document de cahier ouvre un utilisateur, l'application Web démarre un noyau qui exécute le code pour ce ordinateur portable et renvoie la sortie. Cet espace de travail a un noyau Python 3 préinstallé. Des noyaux supplémentaires peuvent être installés pour accéder à d'autres langues (par exemple, R, Scala, GO) ou des ressources informatiques supplémentaires (par exemple, GPU, CPU, mémoire).
Python 2 est rejeté et nous ne recommandons pas de l'utiliser. Cependant, vous pouvez toujours installer un noyau Python 2.7 via cette commande:
/bin/bash /resources/tools/python-27.sh
Cet espace de travail offre un accès VNC basé sur HTTP à l'espace de travail via Novnc. Ainsi, vous pouvez accéder et travailler dans l'espace de travail avec une interface graphique de bureau entièrement tracée. Pour accéder à cette interface graphique de bureau, accédez à Open Tool , sélectionnez VNC et cliquez sur le bouton Connect . Dans le cas, vous êtes demandé un mot de passe, utilisez vncpassword .
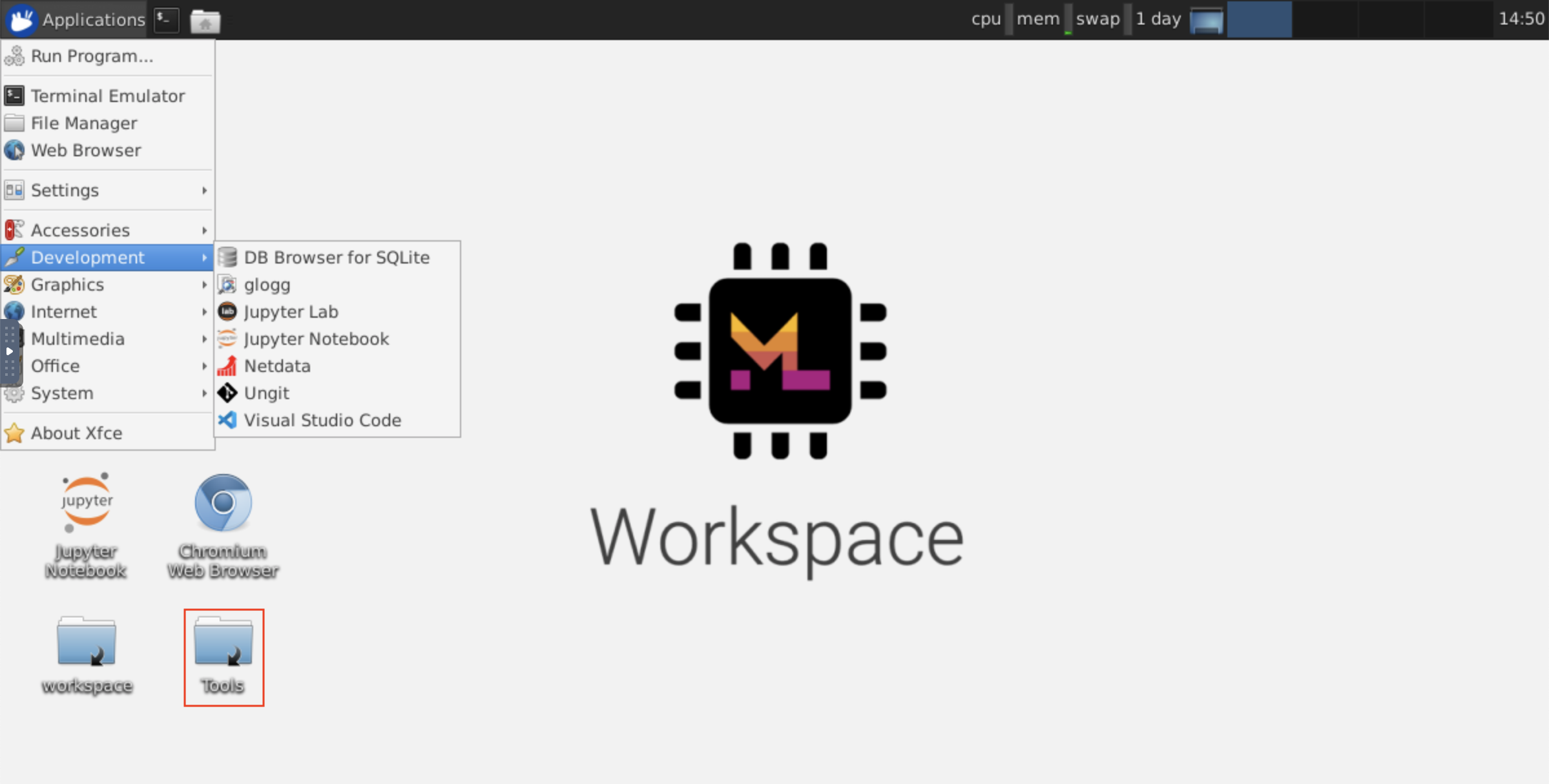
Une fois que vous êtes connecté, vous verrez une interface graphique de bureau qui vous permet d'installer et d'utiliser des navigateurs Web à part entière ou tout autre outil disponible pour Ubuntu. Dans le dossier Tools sur le bureau, vous trouverez une collection de scripts d'installation qui rend simple pour installer certains des outils de développement les plus couramment utilisés, tels que Atom, PyCharm, R-Runtime, R-Studio ou Postman (il suffit de double-cliquez sur le script).
Presse-papiers: Si vous souhaitez partager le presse-papiers entre votre machine et l'espace de travail, vous pouvez utiliser la fonctionnalité de copie-coller comme décrit ci-dessous:
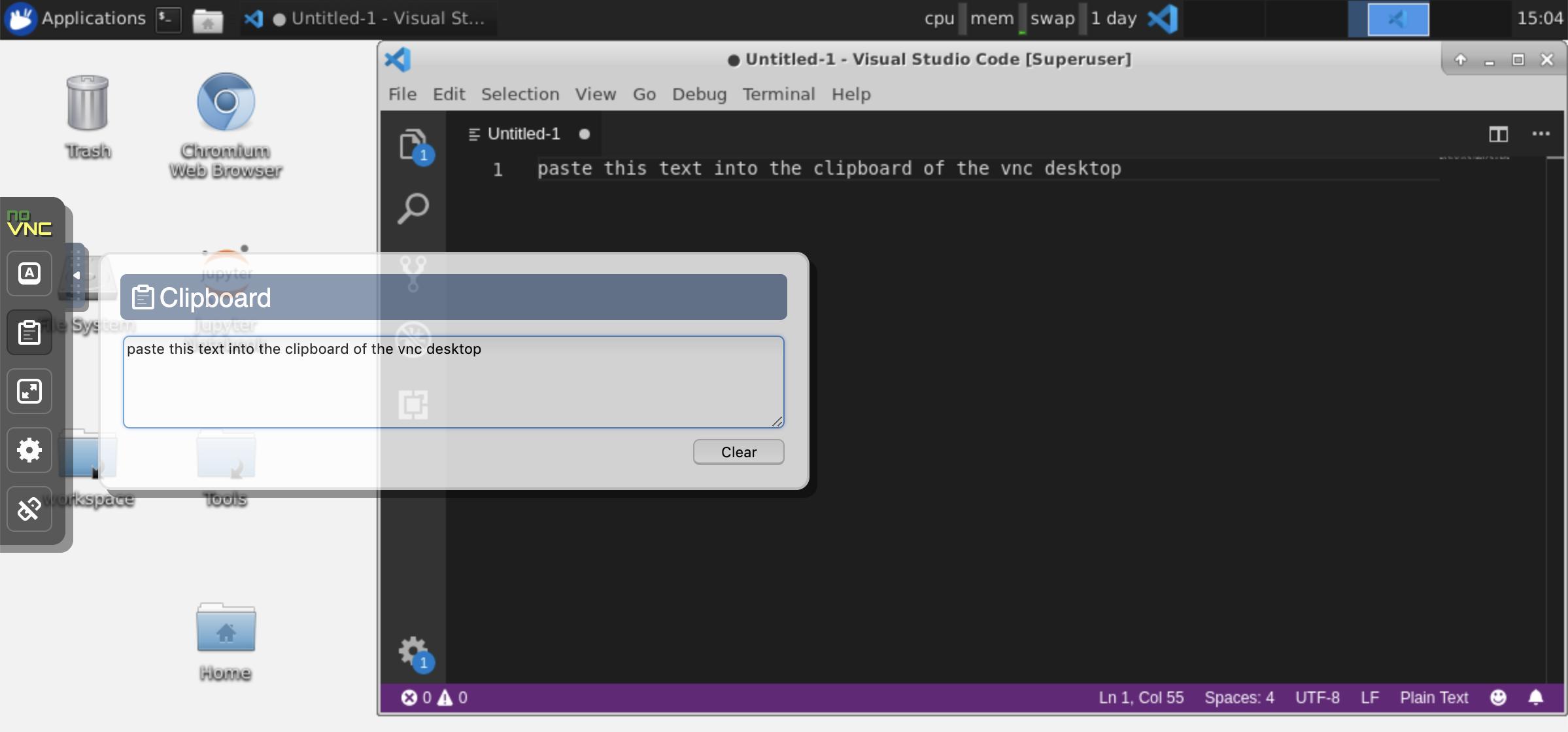
Tâches de longue durée: utilisez l'interface graphique de bureau pour les exécutions de jupyter à longue durée. En exécutant des cahiers à partir du navigateur de votre interface graphique de bureau d'espace de travail, toutes les sorties seront synchronisées avec le cahier même si vous avez déconnecté votre navigateur à partir du cahier.
Visual Studio Code ( Open Tool -> VS Code ) est un éditeur de code léger mais puissant open source avec une prise en charge intégrée pour une variété de langues et un riche écosystème d'extensions. Il combine la simplicité d'un éditeur de code source avec de puissants outils de développeur, comme l'achèvement et le débogage du code Intellisense. L'espace de travail intègre VS Code en tant qu'application Web accessible via le navigateur basé sur le projet de serveur de code impressionnant. Il vous permet de personnaliser chaque fonctionnalité à votre goût et d'installer n'importe quel nombre d'extensions de tiers.
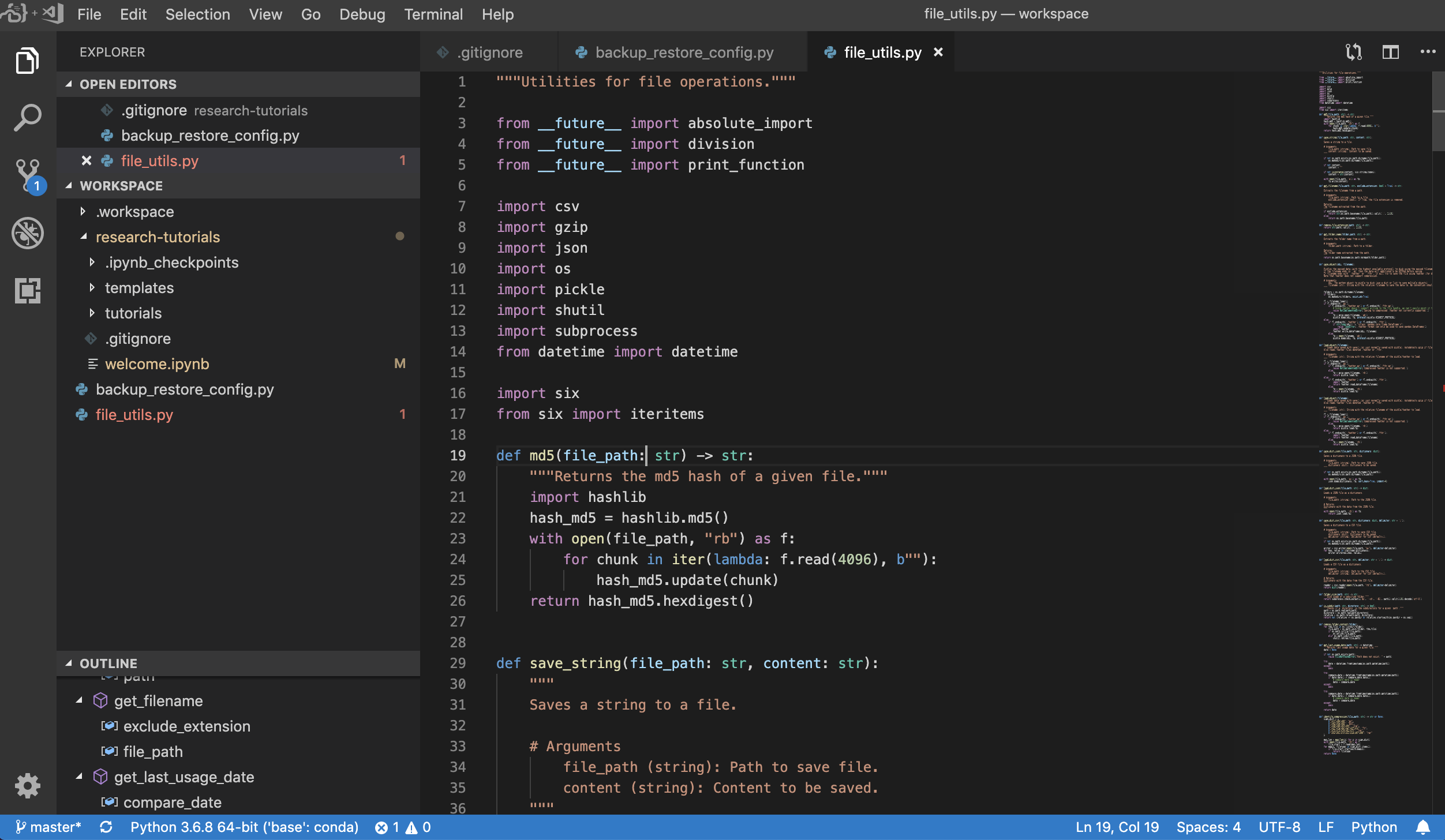
L'espace de travail fournit également une intégration de code vs dans Jupyter vous permettant d'ouvrir une instance de code vs pour tout dossier sélectionné, comme indiqué ci-dessous:

JupyterLab ( Open Tool -> JupyterLab ) est l'interface utilisateur de nouvelle génération pour Project Jupyter. Il propose tous les éléments constitutifs familiers du cahier Jupyter classique (cahier, terminal, éditeur de texte, navigateur de fichiers, sorties riches, etc.) dans une interface utilisateur flexible et puissante. Cette instance JupyterLab est préinstallée avec quelques extensions utiles telles que le jupyterlab-toc, le jupyterlab-git et le juptyterlab-tensorboard.
Le contrôle des versions est un aspect crucial de la collaboration productive. Pour rendre ce processus aussi fluide que possible, nous avons intégré une extension de jupyter sur mesure spécialisée sur la poussée de cahiers uniques, un client Git basé sur le Web à part entière (Ungit), un outil pour ouvrir et modifier des documents texte brut (par exemple, .py , .md ) en tant que cahiers (JupyText), ainsi qu'un outil de merging de carnet (nbdime). De plus, JupyterLab et VS Code fournissent également des clients GIT basés sur GUI.
Pour les référentiels de clonage via https , nous vous recommandons de naviguer vers le dossier racine souhaité et de cliquer sur le bouton git comme indiqué ci-dessous:
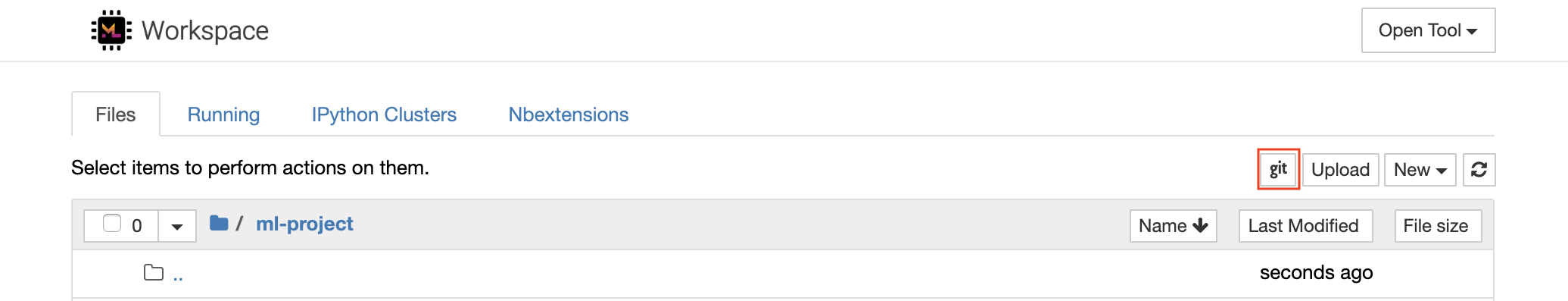
Cela peut demander certains paramètres requis et, par la suite, ouvre UNGIT, un client GIT basé sur le Web avec une interface utilisateur propre et intuitive qui permet de synchroniser vos artefacts de code. Dans Ungit, vous pouvez cloner n'importe quel référentiel. Si l'authentification est requise, vous recevrez vos informations d'identification.
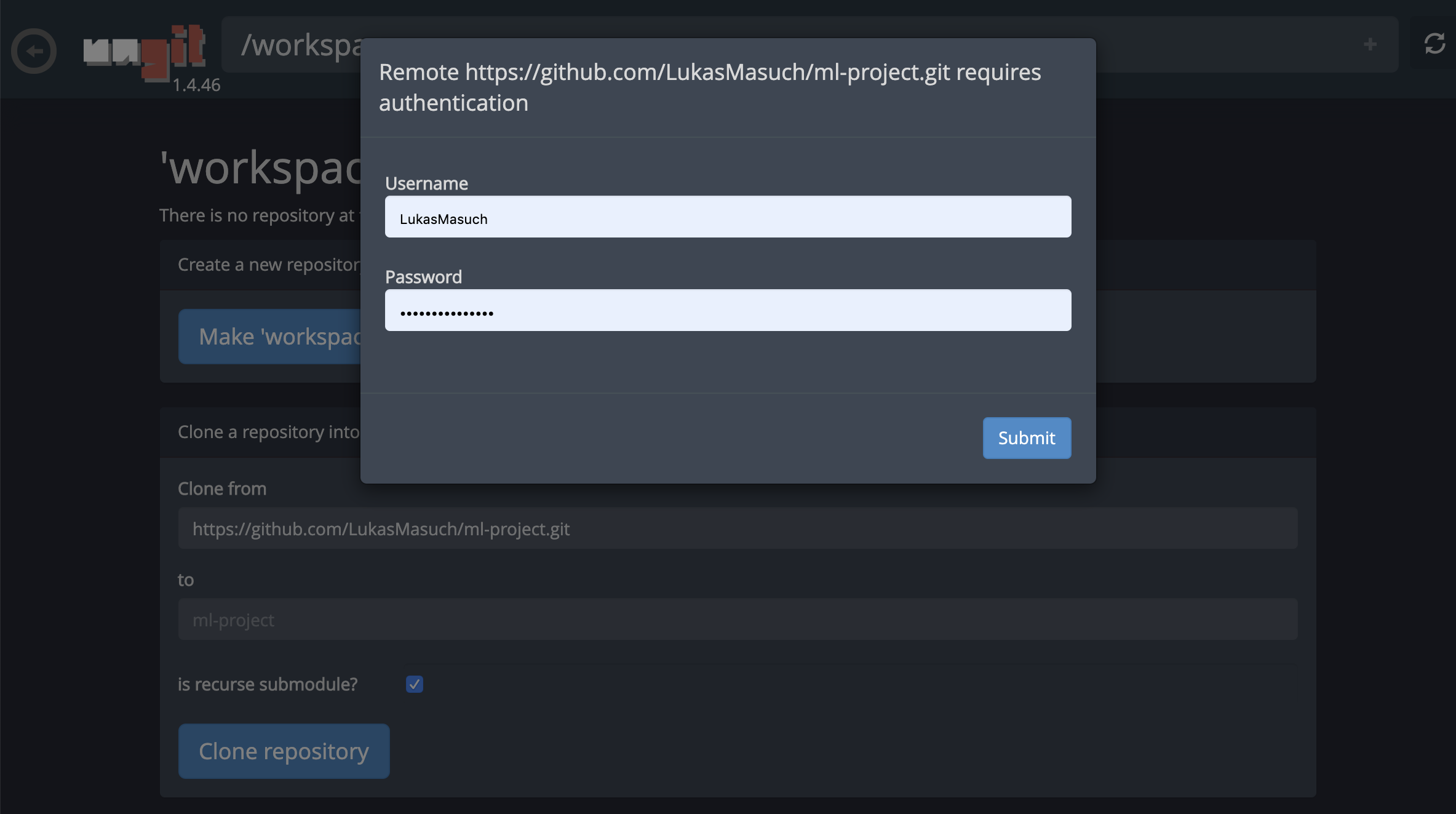
Pour engager et pousser un seul ordinateur portable vers un référentiel GIT distant, nous vous recommandons d'utiliser le plugin GIT intégré dans Jupyter, comme indiqué ci-dessous:
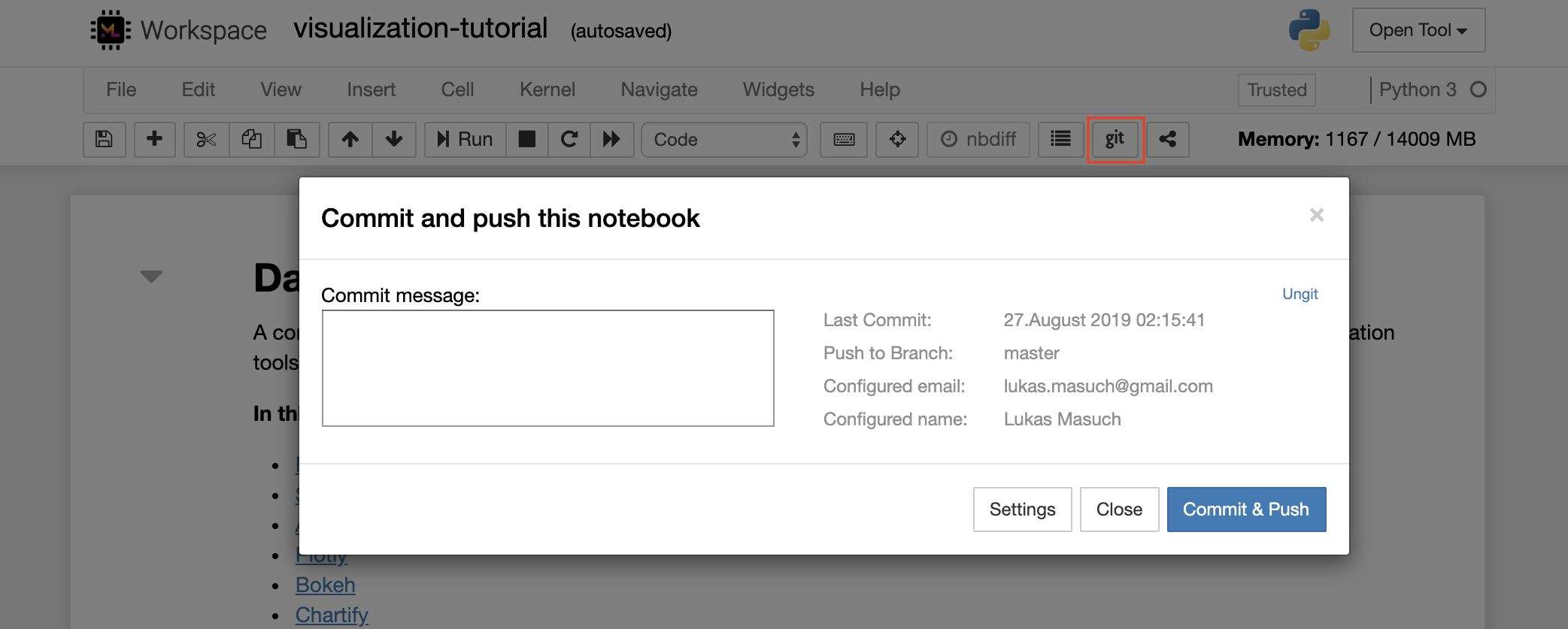
Pour des opérations GIT plus avancées, nous vous recommandons d'utiliser ungit. Avec Ungit, vous pouvez effectuer la plupart des actions GIT communes telles que Push, Pull, Merge, Branch, Tag, Checkout et bien d'autres.
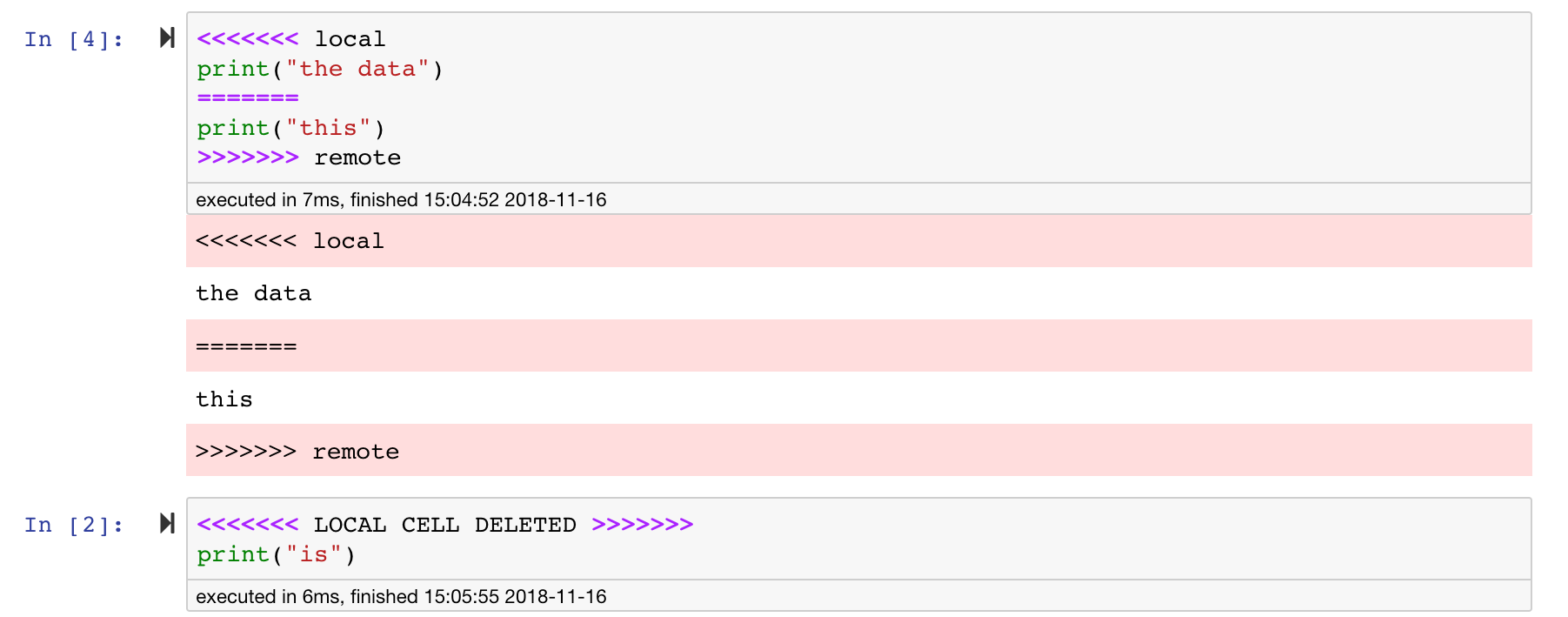
Les cahiers Jupyter sont excellents, mais ce sont souvent d'énormes fichiers, avec un format de fichier JSON très spécifique. Pour permettre une diffusion et une fusion transparentes via GIT, cet espace de travail est préinstallé avec NBDIME. NBDIME comprend la structure des documents de carnet et, par conséquent, prend automatiquement des décisions intelligentes lors de la diffusion et de la fusion des cahiers. Dans le cas où vous avez des conflits fusionnés, NBDIME s'assurera que le cahier est toujours lisible par Jupyter, comme indiqué ci-dessous:
De plus, l'espace de travail est pré-installé avec JupyText, un plugin Jupyter qui lit et écrit des ordinateurs portables en tant que fichiers texte brut. Cela vous permet d'ouvrir, de modifier et d'exécuter des scripts ou des fichiers de marque (par exemple, .py , .md ) comme ordinateurs portables dans Jupyter. Dans la capture d'écran suivante, nous avons ouvert un fichier Markdown via Jupyter:
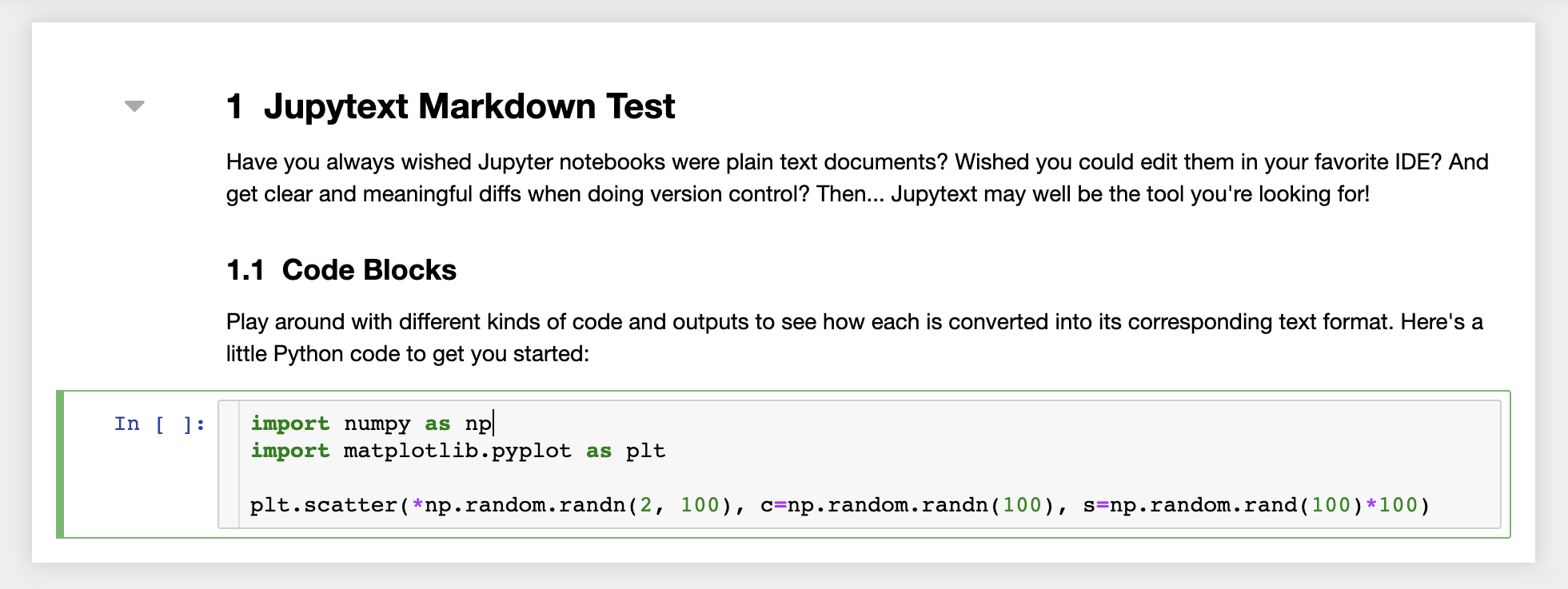
En combinaison avec Git, JupyText permet une histoire claire et une fusion facile des conflits de version. Avec ces deux outils, collaborer sur des cahiers Jupyter avec GIT devient simple.
L'espace de travail dispose d'une fonctionnalité pour partager n'importe quel fichier ou dossier avec quiconque via un lien protégé de jeton. Pour partager les données via un lien, sélectionnez n'importe quel fichier ou dossier dans l'arborescence du répertoire Jupyter et cliquez sur le bouton Partager comme indiqué dans la capture d'écran suivante:
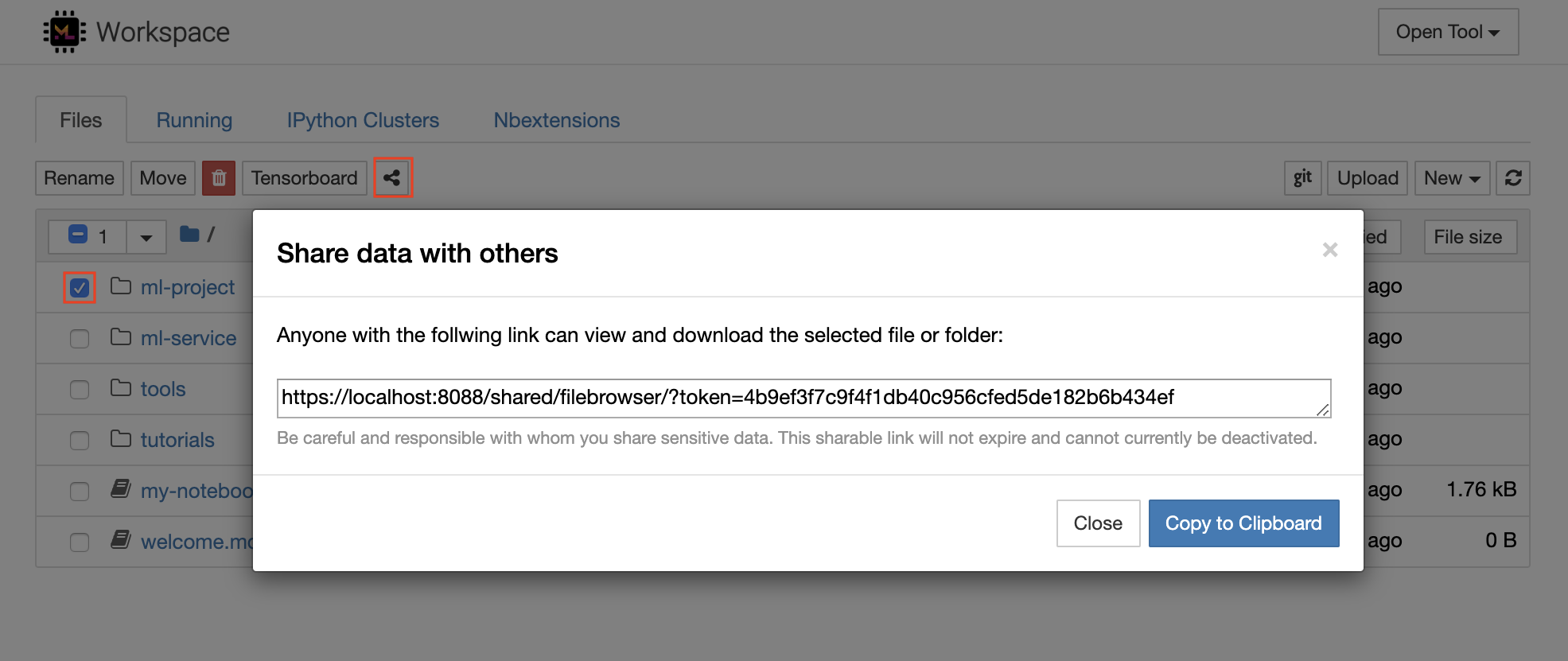
Cela générera un lien unique protégé via un jeton qui donne à toute personne ayant l'accès au lien pour afficher et télécharger les données sélectionnées via l'interface utilisateur FileBrowser:
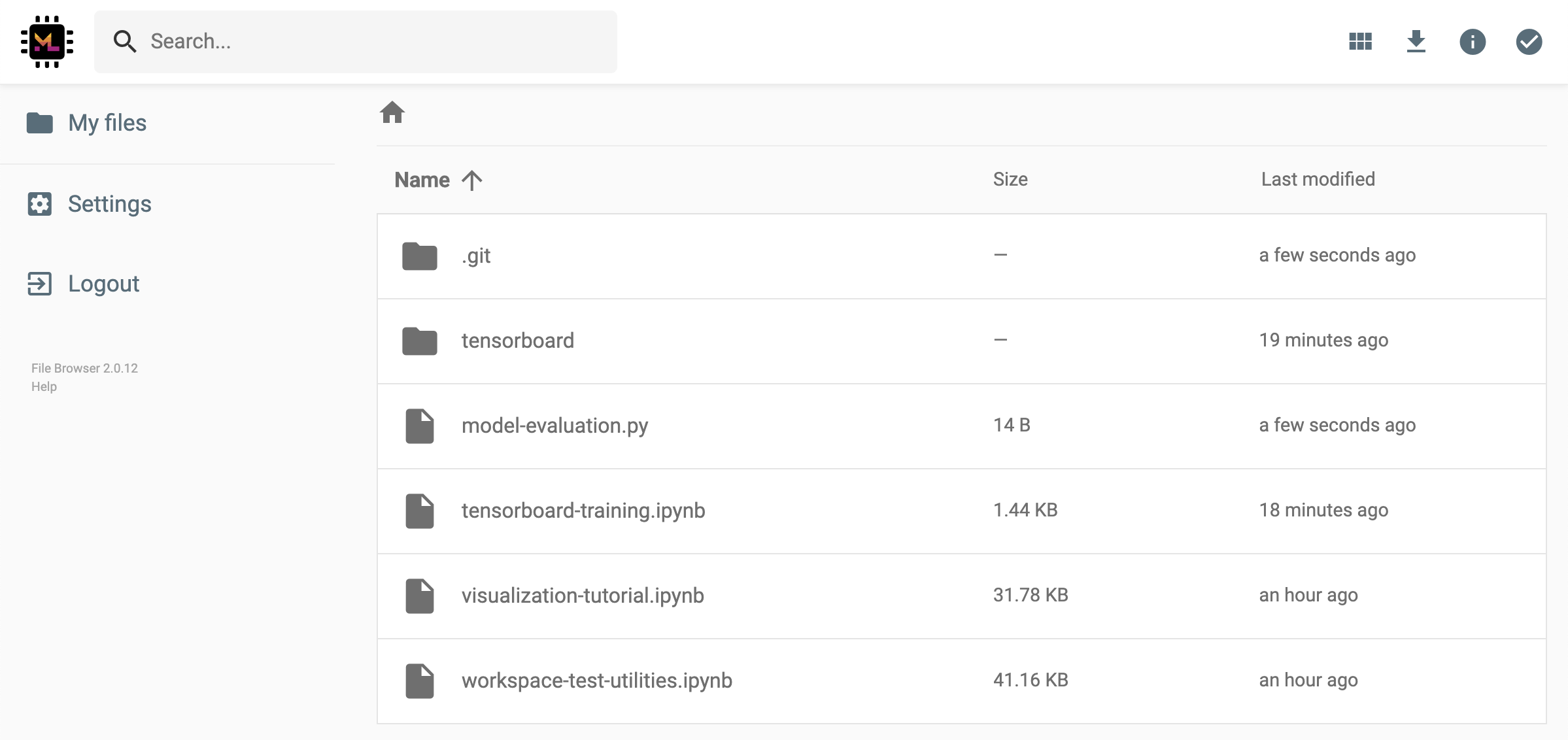
Pour désactiver ou gérer (par exemple, fournir des autorisations d'édition) des liens partagés, ouvrez le FileBrowser via Open Tool -> Filebrowser et sélectionnez Settings->User Management .
Il est possible d'accéder en toute sécurité à n'importe quel port interne d'espace de travail en sélectionnant Open Tool -> Access Port . Avec cette fonctionnalité, vous pouvez accéder à une API REST ou à une application Web exécutée directement dans l'espace de travail avec votre navigateur. La fonction permet aux développeurs de créer, d'exécuter, de tester et de déboguer les API REST ou les applications Web directement à partir de l'espace de travail.
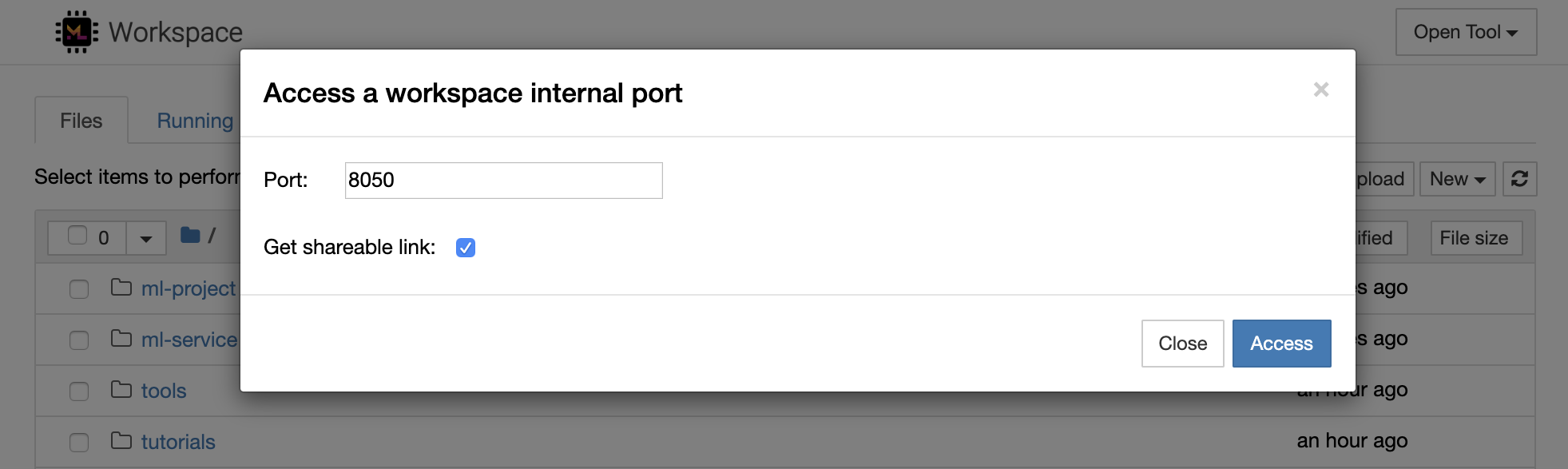
Si vous souhaitez utiliser un client HTTP ou partager un accès à un port donné, vous pouvez sélectionner l'option Get shareable link . Cela génère un lien sécurisé à jeton que toute personne ayant un accès au lien peut utiliser pour accéder au port spécifié.
L'application HTTP doit être résolue à partir d'un chemin d'url relatif ou configurer un chemin de base (
/tools/PORT/). Les outils rendus accessibles de cette façon sont sécurisés par le système d'authentification de l'espace de travail! Si vous décidez de publier vous-même un autre port du conteneur au lieu d'utiliser cette fonctionnalité pour rendre un outil accessible, assurez-vous de le sécuriser via un mécanisme d'authentification!
1234 en exécutant cette commande dans un terminal dans l'espace de travail: python -m http.server 1234Open Tool -> Access Port , le port de saisie 1234 et sélectionnez l'option Get shareable link .Access et vous verrez le contenu fourni par http.server de Python. SSH fournit un ensemble puissant de fonctionnalités qui vous permet d'être plus productif avec vos tâches de développement. Vous pouvez facilement configurer une connexion SSH sécurisée et sans mot de passe à un espace de travail en sélectionnant Open Tool -> SSH . Cela générera une commande de configuration sécurisée qui peut être exécutée sur n'importe quelle machine Linux ou Mac pour configurer une connexion SSH sans mot de passe et sécurisée à l'espace de travail. Alternativement, vous pouvez également télécharger le script de configuration et l'exécuter (au lieu d'utiliser la commande).
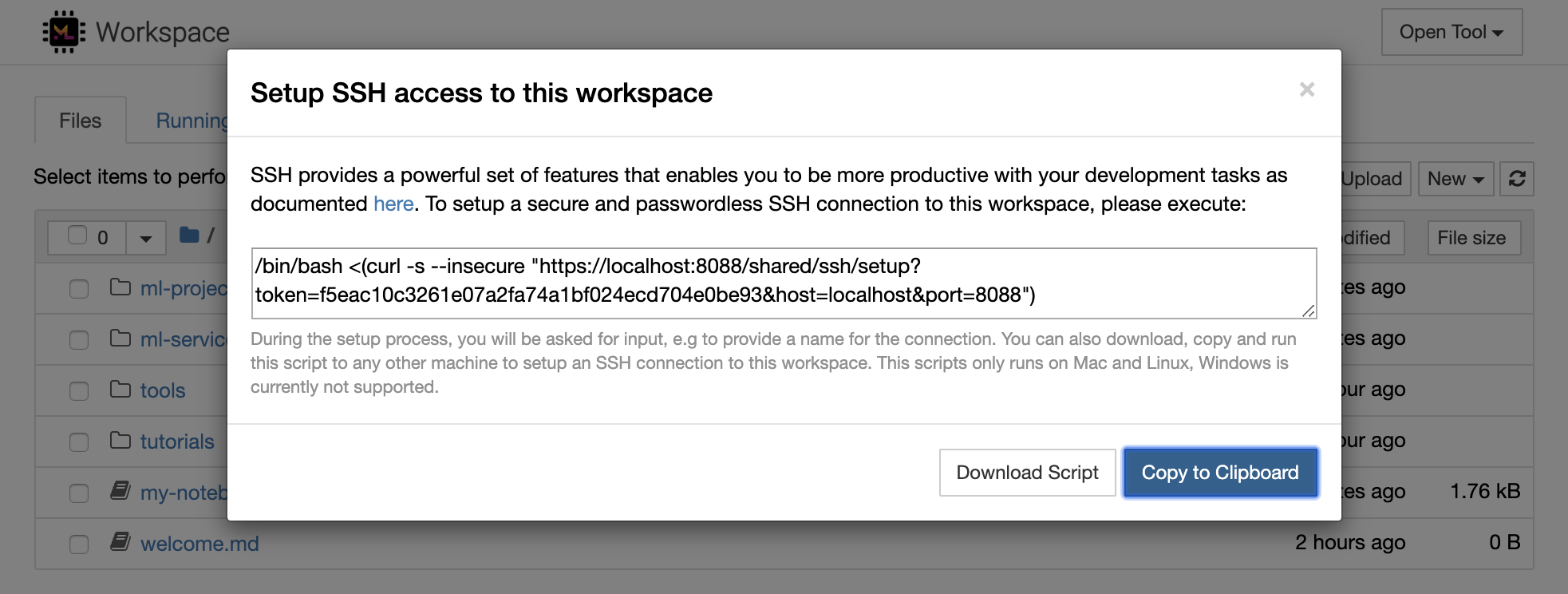
Le script de configuration s'exécute uniquement sur Mac et Linux. Windows n'est actuellement pas pris en charge.
Exécutez simplement la commande de configuration ou le script sur la machine à partir de l'endroit où vous souhaitez configurer une connexion à l'espace de travail et saisir un nom pour la connexion (par exemple, my-workspace ). Vous pourriez également recevoir une entrée supplémentaire pendant le processus, par exemple pour installer un noyau distant si remote_ikernel est installé. Une fois que la connexion SSH sans mot de passe est configurée et testée avec succès, vous pouvez vous connecter en toute sécurité à l'espace de travail en exécutant simplement ssh my-workspace .
Outre la possibilité d'exécuter des commandes sur une machine distante, SSH fournit également une variété d'autres fonctionnalités qui peuvent améliorer votre flux de travail de développement comme décrit dans les sections suivantes.
Une connexion SSH peut être utilisée pour les ports d'application de tunneling de la machine distante vers la machine locale, ou vice versa. Par exemple, vous pouvez exposer le port interne de l'espace de travail 5901 (serveur VNC) à la machine locale sur le port 5000 en exécutant:
ssh -nNT -L 5000:localhost:5901 my-workspacePour exposer un port d'application de votre machine locale à un espace de travail, utilisez l'option
-R(au lieu de-L).
Une fois le tunnel établi, vous pouvez utiliser votre visionneuse VNC préférée sur votre machine locale et vous connecter à vnc://localhost:5000 (mot de passe par défaut: vncpassword ). Pour rendre la connexion du tunnel plus résistante et fiable, nous vous recommandons d'utiliser Autossh pour redémarrer automatiquement les tunnels SSH dans le cas où la connexion meurt:
autossh -M 0 -f -nNT -L 5000:localhost:5901 my-workspaceLe tunneling portuaire est très utile lorsque vous avez démarré un outil basé sur un serveur dans l'espace de travail que vous aimez rendre accessible pour une autre machine. Dans son paramètre par défaut, l'espace de travail propose une variété d'outils qui s'exécutent déjà sur différents ports, tels que:
8080 : port d'espace de travail principal avec accès à tous les outils intégrés.8090 : serveur Jupyter.8054 : VS Code Server.5901 : VNC Server.22 : serveur SSH.Vous pouvez trouver des informations sur le port sur tous les outils de la configuration du superviseur.
Pour plus d'informations sur le tunneling / transfert portuaire, nous recommandons ce guide.
SCP permet aux fichiers et répertoires d'être copiés en toute sécurité sur, à partir ou entre différentes machines via des connexions SSH. Par exemple, pour copier un fichier local ( ./local-file.txt ) dans le dossier /workspace à l'intérieur de l'espace de travail, exécutez:
scp ./local-file.txt my-workspace:/workspace Pour copier le répertoire /workspace de my-workspace au répertoire de travail de la machine locale, exécutez:
scp -r my-workspace:/workspace .Pour plus d'informations sur SCP, nous recommandons ce guide.
RSYNC est un utilitaire pour transférer et synchroniser efficacement les fichiers entre différentes machines (par exemple, via les connexions SSH) en comparant les temps de modification et les tailles des fichiers. La commande RSYNC déterminera quels fichiers doivent être mis à jour à chaque fois qu'il est exécuté, ce qui est beaucoup plus efficace et pratique que d'utiliser quelque chose comme SCP ou SFTP. Par exemple, pour synchroniser tous les contenus d'un dossier local ( ./local-project-folder/ ) dans le /workspace/remote-project-folder/ dossier à l'intérieur de l'espace de travail, exécutez:
rsync -rlptzvP --delete --exclude= " .git " " ./local-project-folder/ " " my-workspace:/workspace/remote-project-folder/ "Si vous avez des modifications à l'intérieur du dossier de l'espace de travail, vous pouvez synchroniser ces modifications dans le dossier local en modifiant les arguments source et de destination:
rsync -rlptzvP --delete --exclude= " .git " " my-workspace:/workspace/remote-project-folder/ " " ./local-project-folder/ "Vous pouvez relancer ces commandes chaque fois que vous souhaitez synchroniser la dernière copie de vos fichiers. RSYNC s'assurera que seules les mises à jour seront transférées.
Vous pouvez trouver plus d'informations sur RSYNC sur cette page Man.
Outre la copie et la synchronisation des données, une connexion SSH peut également être utilisée pour monter les répertoires à partir d'une machine distante dans le système de fichiers local via SSHFS. Par exemple, pour monter le répertoire /workspace de my-workspace dans un chemin local (par exemple /local/folder/path ), exécutez:
sshfs -o reconnect my-workspace:/workspace /local/folder/pathUne fois le répertoire distant monté, vous pouvez interagir avec le système de fichiers distant de la même manière qu'avec n'importe quel répertoire et fichier local.
Pour plus d'informations sur SSHFS, nous recommandons ce guide.
L'espace de travail peut être intégré et utilisé comme exécutif à distance (également connu sous le nom de noyau / machine / interprète distant) pour une variété d'outils de développement et d'ides populaires, tels que le jupyter, le code vs, le pycharm, le colab ou l'hydrogène atome. Ainsi, vous pouvez connecter votre outil de développement préféré en cours d'exécution sur votre machine locale à une machine distante pour l'exécution de code. Cela permet une expérience de développement de qualité locale avec des ressources de calcul hébergées à distance.
Ces intégrations nécessitent généralement une connexion SSH sans mot de passe de la machine locale à l'espace de travail. Pour configurer une connexion SSH, veuillez suivre les étapes expliquées dans la section d'accès SSH.
L'espace de travail peut être ajouté à une instance de jupyter en tant que noyau distant à l'aide de l'outil Remote_ikernel. Si vous avez installé Remote_ikernel ( pip install remote_ikernel ) sur votre machine locale, le script de configuration SSH de l'espace de travail vous offrira automatiquement la possibilité de configurer une connexion du noyau distant.
Lors de l'exécution des noyaux sur des machines distantes, les ordinateurs portables eux-mêmes seront enregistrés sur le système de fichiers local, mais le noyau n'aura accès qu'au système de fichiers de la machine distante exécutant le noyau. Si vous avez besoin de synchroniser les données, vous pouvez utiliser RSYNC, SCP ou SSHFS comme expliqué dans la section d'accès SSH.
Dans le cas où vous souhaitez configurer et gérer manuellement les noyaux distants, utilisez l'outil de ligne de commande Remote_ikernel, comme indiqué ci-dessous:
# Change my-workspace with the name of a workspace SSH connection
remote_ikernel manage --add
--interface=ssh
--kernel_cmd= " ipython kernel -f {connection_file} "
--name= " ml-server (Python) "
--host= " my-workspace " Vous pouvez utiliser les fonctionnalités de ligne de commande Remote_ikernel à liste ( remote_ikernel manage --show ) ou delete ( remote_ikernel manage --delete <REMOTE_KERNEL_NAME> ) Connexions de noyau distantes.
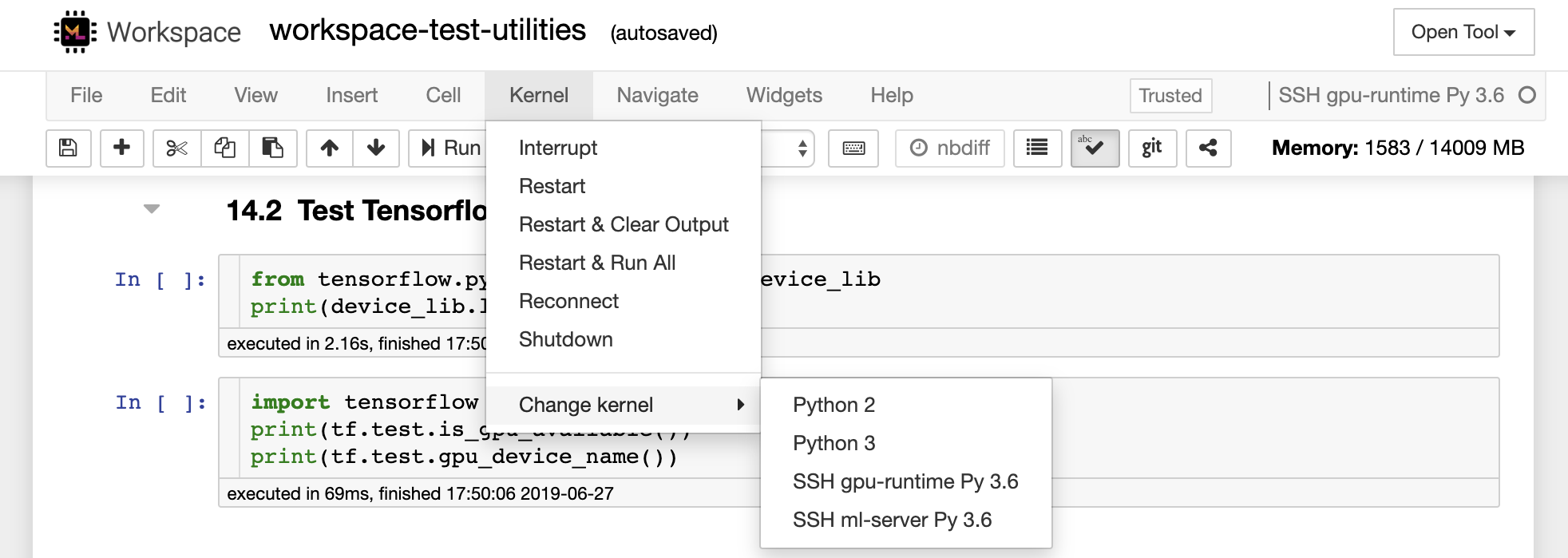
Le code Visual Studio Remote - Extension SSH vous permet d'ouvrir un dossier distant sur n'importe quelle machine distante avec un accès SSH et de travailler avec lui comme vous le feriez si le dossier était sur votre propre machine. Une fois connecté à une machine distante, vous pouvez interagir avec les fichiers et les dossiers n'importe où sur le système de fichiers distant et profiter pleinement de l'ensemble de fonctionnalités de VS Code (Intellisense, débogage et support d'extension). Le découvre et fonctionne prêt à l'emploi avec les connexions SSH sans mot de passe, configurées par le script de configuration SSH de l'espace de travail. Pour permettre à votre application de code locale vs pour se connecter à un espace de travail:
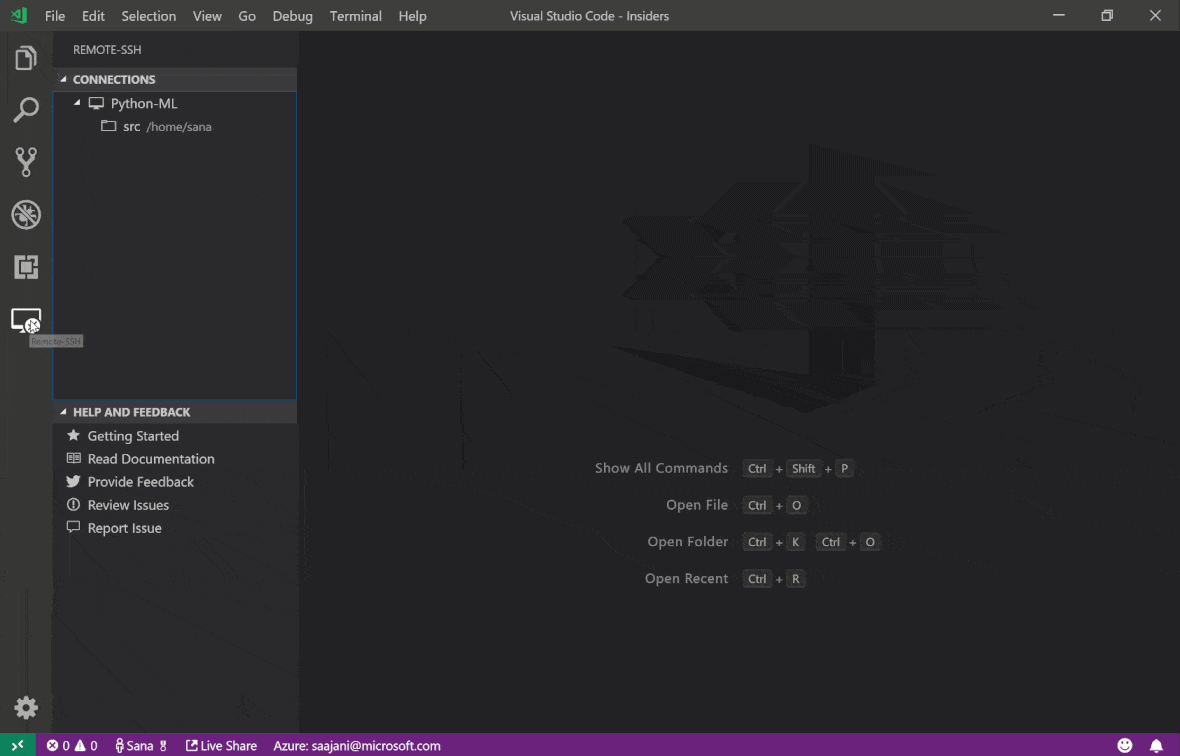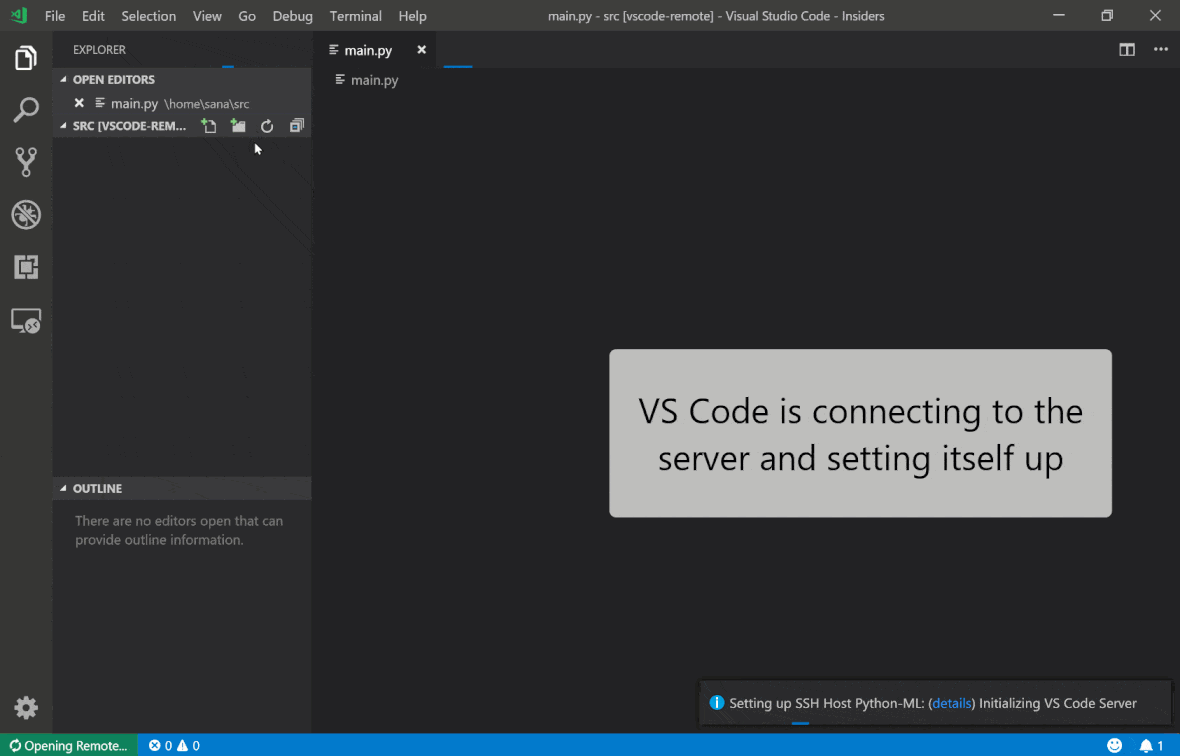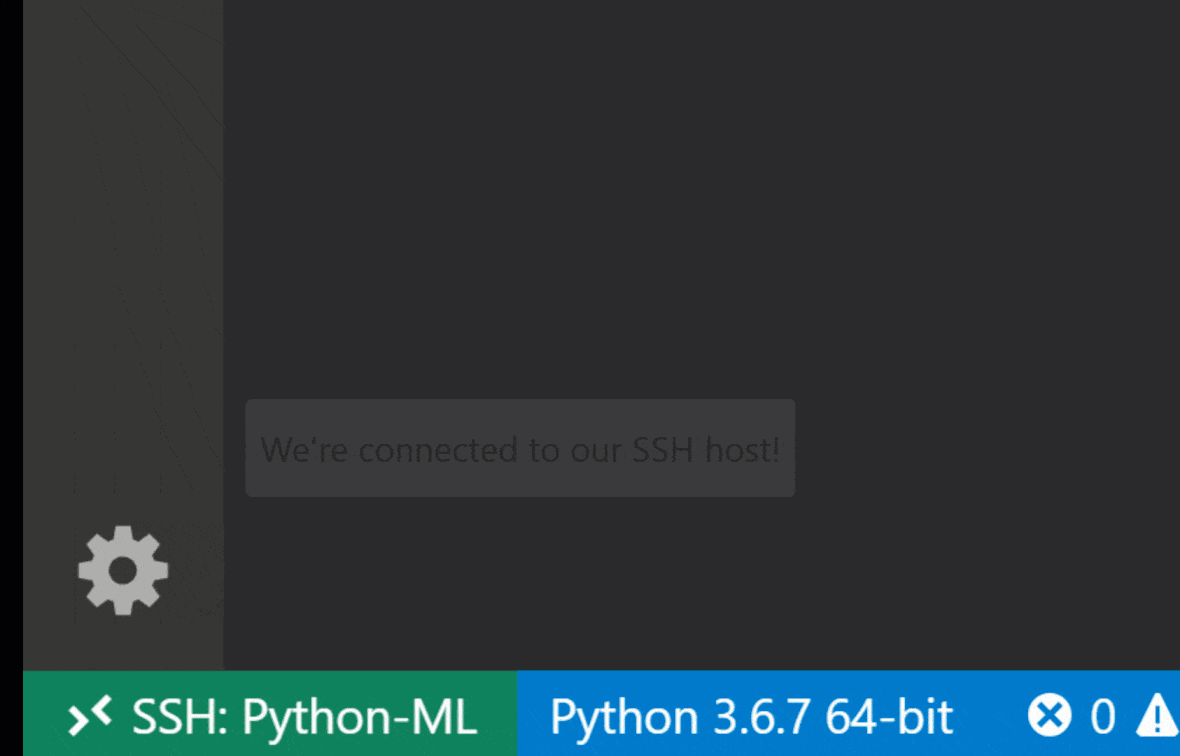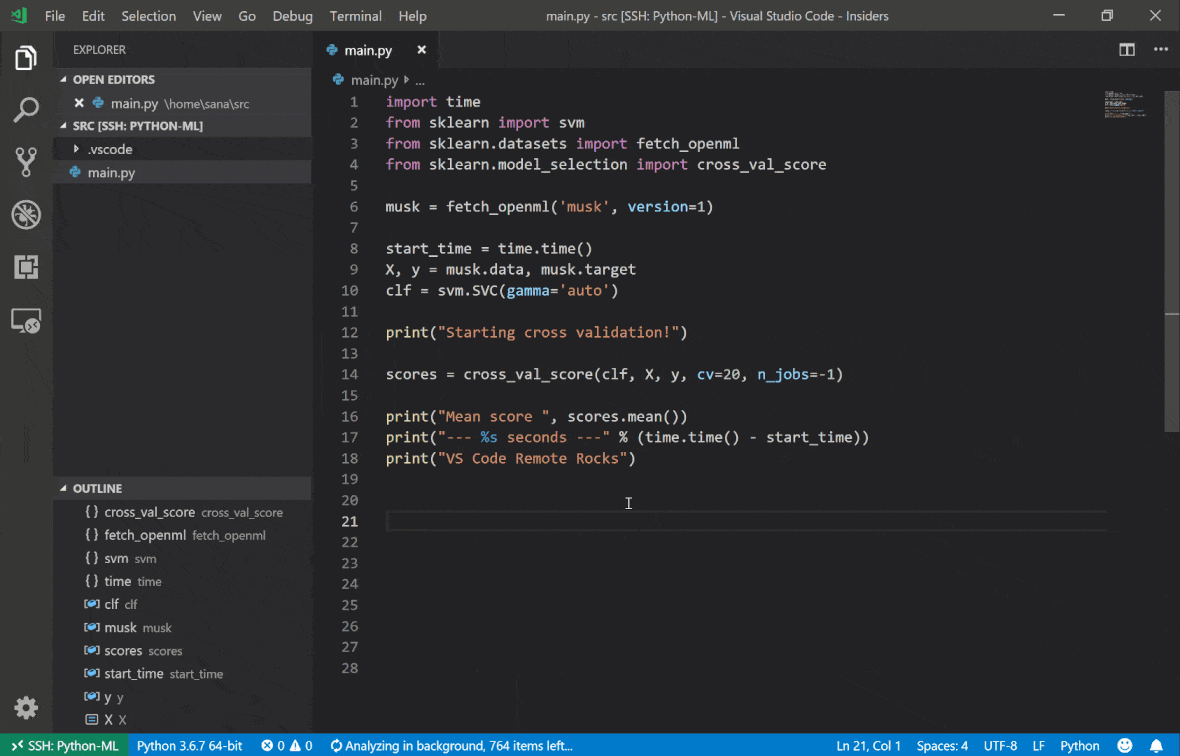
Vous pouvez trouver des fonctionnalités et des informations supplémentaires sur l'extension SSH distante de ce guide.
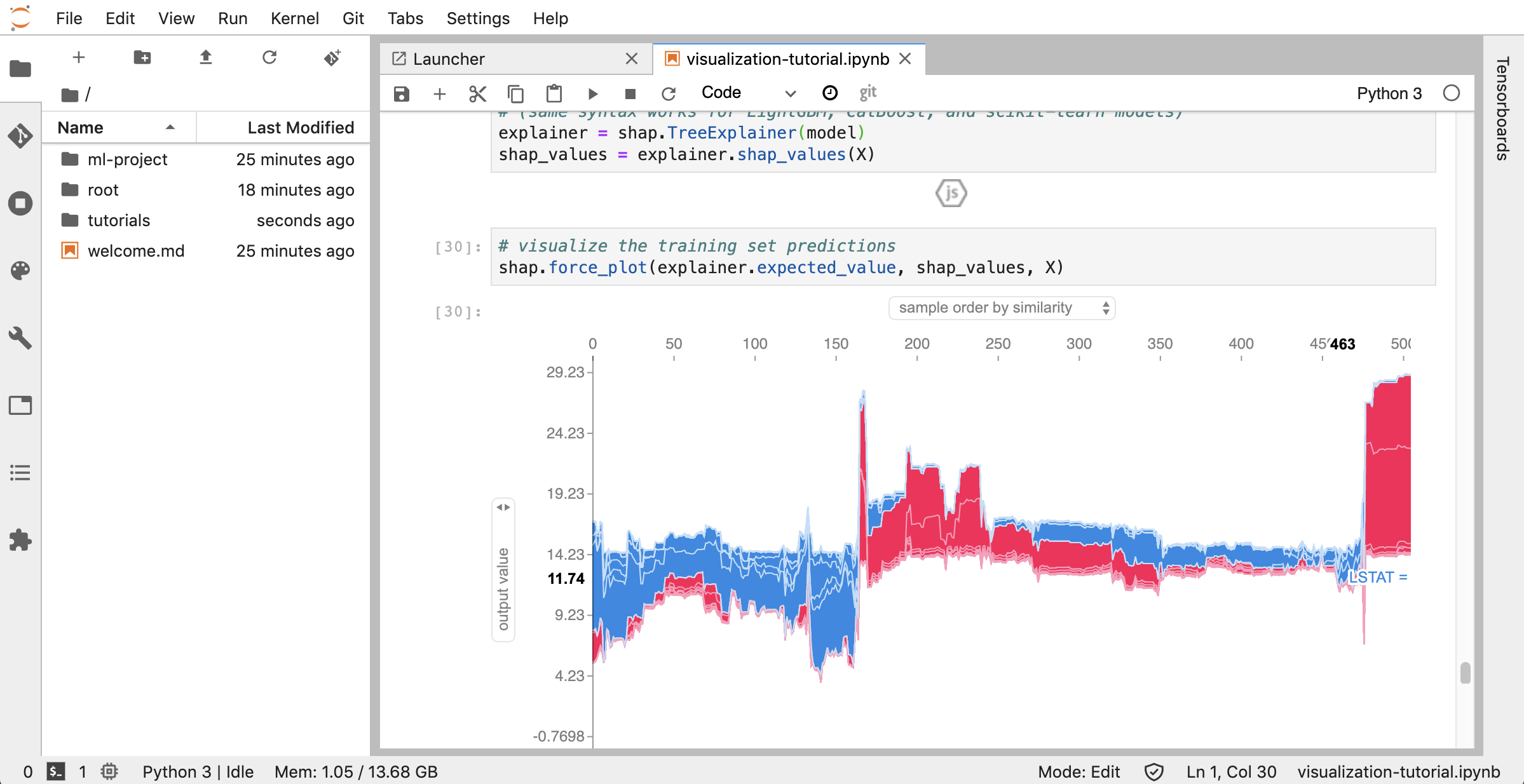
Tensorboard fournit une suite d'outils de visualisation pour faciliter la compréhension, le débogage et l'optimisation de vos courses d'expérience. Il comprend des fonctionnalités de journalisation pour le scalaire, l'histogramme, la structure du modèle, les intérêts et la visualisation du texte et de l'image. L'espace de travail est préinstallé avec une extension Jupyter_tensorboard qui intègre Tensorboard dans l'interface Jupyter avec les fonctionnalités pour démarrer, gérer et arrêter les instances. Vous pouvez ouvrir une nouvelle instance pour un répertoire de journaux valides, comme indiqué ci-dessous:
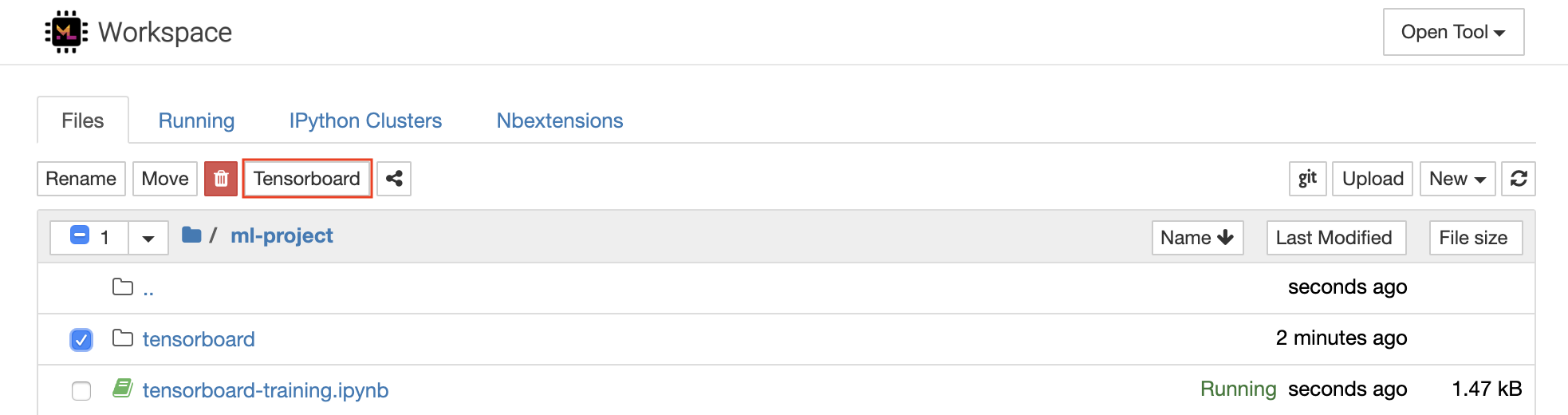
Si vous avez ouvert une instance Tensorboard dans un répertoire de journal valide, vous verrez les visualisations de vos données enregistrées:
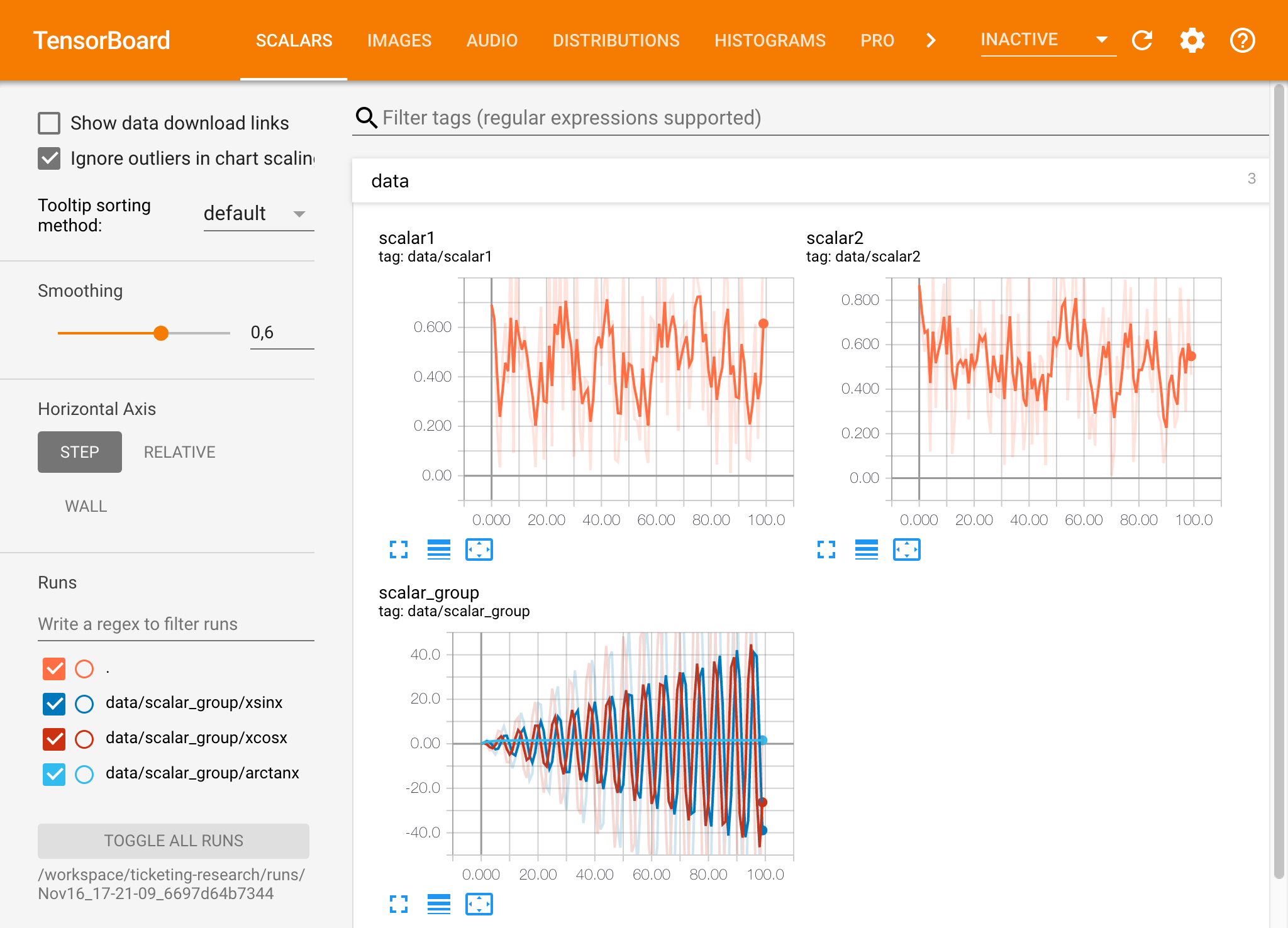
Tensorboard peut être utilisé en combinaison avec de nombreux autres cadres ML en plus de TensorFlow. En utilisant la bibliothèque Tensorboardx, vous pouvez vous connecter essentiellement à partir de n'importe quelle bibliothèque basée sur Python. En outre, Pytorch a une intégration directe du tensorboard comme décrit ici.
Si vous préférez voir le Tensorboard directement dans votre ordinateur portable, vous pouvez utiliser la magie de Jupyter suivante:
%load_ext tensorboard
%tensorboard --logdir /workspace/path/to/logs
L'espace de travail fournit deux outils Web préinstallés pour aider les développeurs pendant la formation des modèles et d'autres tâches d'expérimentation pour obtenir un aperçu de tout ce qui se passe sur le système et de déterminer les goulots d'étranglement des performances.
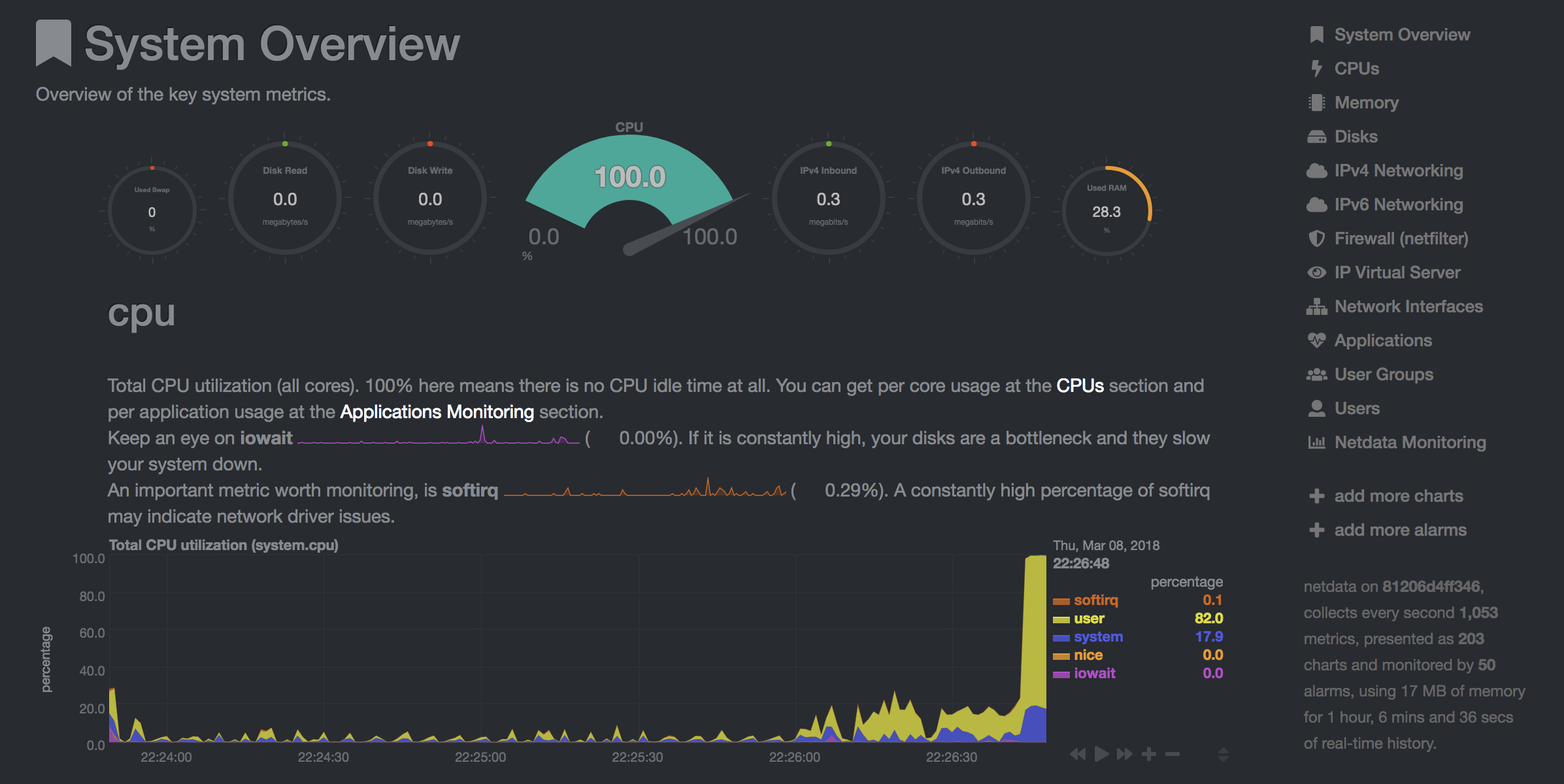
NetData ( Open Tool -> Netdata ) est un tableau de bord matériel et de surveillance des performances en temps réel qui visualise les processus et services sur vos systèmes Linux. Il surveille les mesures sur le CPU, le GPU, la mémoire, les disques, les réseaux, les processus, etc.
Glances ( Open Tool -> Glances ) est également un tableau de bord de surveillance du matériel basé sur le Web et peut être utilisé comme alternative à NetData.
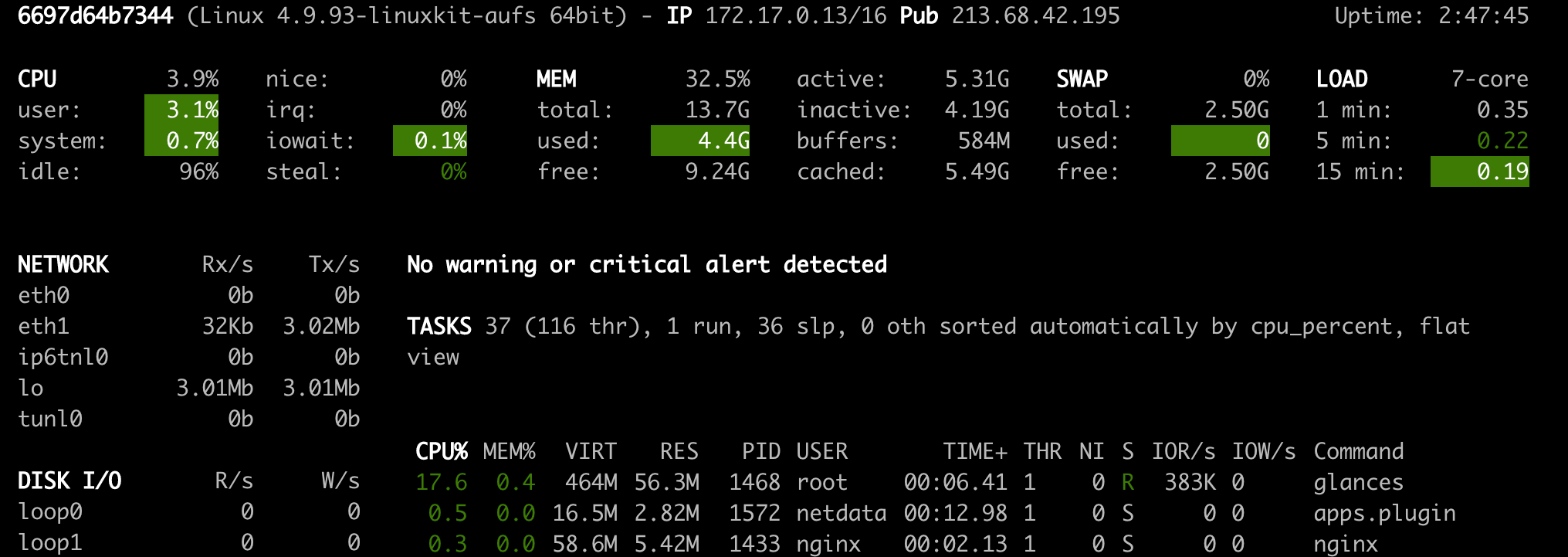
NetData et Glances vous montreront les statistiques matérielles de l'ensemble de la machine sur laquelle le conteneur d'espace de travail est en cours d'exécution.
Un travail est défini comme toute tâche de calcul qui s'exécute pendant un certain temps pour terminer, comme une formation de modèle ou un pipeline de données.
L'image de l'espace de travail peut également être utilisée pour exécuter le code Python arbitraire sans démarrer aucun des outils préinstallés. This provides a seamless way to productize your ML projects since the code that has been developed interactively within the workspace will have the same environment and configuration when run as a job via the same workspace image.
To run Python code as a job, you need to provide a path or URL to a code directory (or script) via EXECUTE_CODE . The code can be either already mounted into the workspace container or downloaded from a version control system (eg, git or svn) as described in the following sections. The selected code path needs to be python executable. In case the selected code is a directory (eg, whenever you download the code from a VCS) you need to put a __main__.py file at the root of this directory. The __main__.py needs to contain the code that starts your job.
You can execute code directly from Git, Mercurial, Subversion, or Bazaar by using the pip-vcs format as described in this guide. For example, to execute code from a subdirectory of a git repository, just run:
docker run --env EXECUTE_CODE= " git+https://github.com/ml-tooling/ml-workspace.git#subdirectory=resources/tests/ml-job " mltooling/ml-workspace:0.13.2For additional information on how to specify branches, commits, or tags please refer to this guide.
In the following example, we mount and execute the current working directory (expected to contain our code) into the /workspace/ml-job/ directory of the workspace:
docker run -v " ${PWD} :/workspace/ml-job/ " --env EXECUTE_CODE= " /workspace/ml-job/ " mltooling/ml-workspace:0.13.2In the case that the pre-installed workspace libraries are not compatible with your code, you can install or change dependencies by just adding one or multiple of the following files to your code directory:
requirements.txt : pip requirements format for pip-installable dependencies.environment.yml : conda environment file to create a separate Python environment.setup.sh : A shell script executed via /bin/bash . The execution order is 1. environment.yml -> 2. setup.sh -> 3. requirements.txt
You can test your job code within the workspace (started normally with interactive tools) by executing the following python script:
python /resources/scripts/execute_code.py /path/to/your/jobIt is also possible to embed your code directly into a custom job image, as shown below:
FROM mltooling/ml-workspace:0.13.2
# Add job code to image
COPY ml-job /workspace/ml-job
ENV EXECUTE_CODE=/workspace/ml-job
# Install requirements only
RUN python /resources/scripts/execute_code.py --requirements-only
# Execute only the code at container startup
CMD [ "python" , "/resources/docker-entrypoint.py" , "--code-only" ]The workspace is pre-installed with many popular interpreters, data science libraries, and ubuntu packages:
conda , pip , apt-get , npm , yarn , sdk , poetry , gdebi ...The full list of installed tools can be found within the Dockerfile.
For every minor version release, we run vulnerability, virus, and security checks within the workspace using safety, clamav, trivy, and snyk via docker scan to make sure that the workspace environment is as secure as possible. We are committed to fix and prevent all high- or critical-severity vulnerabilities. You can find some up-to-date reports here.
The workspace provides a high degree of extensibility. Within the workspace, you have full root & sudo privileges to install any library or tool you need via terminal (eg, pip , apt-get , conda , or npm ). You can open a terminal by one of the following ways:
New -> TerminalApplications -> Terminal EmulatorFile -> New -> TerminalTerminal -> New Terminal Additionally, pre-installed tools such as Jupyter, JupyterLab, and Visual Studio Code each provide their own rich ecosystem of extensions. The workspace also contains a collection of installer scripts for many commonly used development tools or libraries (eg, PyCharm , Zeppelin , RStudio , Starspace ). You can find and execute all tool installers via Open Tool -> Install Tool . Those scripts can be also executed from the Desktop VNC (double-click on the script within the Tools folder on the Desktop VNC).
For example, to install the Apache Zeppelin notebook server, simply execute:
/resources/tools/zeppelin.sh --port=1234 After installation, refresh the Jupyter website and the Zeppelin tool will be available under Open Tool -> Zeppelin . Other tools might only be available within the Desktop VNC (eg, atom or pycharm ) or do not provide any UI (eg, starspace , docker-client ).
As an alternative to extending the workspace at runtime, you can also customize the workspace Docker image to create your own flavor as explained in the FAQ section.
The workspace can be extended in many ways at runtime, as explained here. However, if you like to customize the workspace image with your own software or configuration, you can do that via a Dockerfile as shown below:
# Extend from any of the workspace versions/flavors
FROM mltooling/ml-workspace:0.13.2
# Run you customizations, e.g.
RUN
# Install r-runtime, r-kernel, and r-studio web server from provided install scripts
/bin/bash $RESOURCES_PATH/tools/r-runtime.sh --install &&
/bin/bash $RESOURCES_PATH/tools/r-studio-server.sh --install &&
# Cleanup Layer - removes unneccessary cache files
clean-layer.shFinally, use docker build to build your customized Docker image.
For a more comprehensive Dockerfile example, take a look at the Dockerfile of the R-flavor.
To update a running workspace instance to a more recent version, the running Docker container needs to be replaced with a new container based on the updated workspace image.
All data within the workspace that is not persisted to a mounted volume will be lost during this update process. As mentioned in the persist data section, a volume is expected to be mounted into the /workspace folder. All tools within the workspace are configured to make use of the /workspace folder as the root directory for all source code and data artifacts. During an update, data within other directories will be removed, including installed/updated libraries or certain machine configurations. We have integrated a backup and restore feature ( CONFIG_BACKUP_ENABLED ) for various selected configuration files/folders, such as the user's Jupyter/VS-Code configuration, ~/.gitconfig , and ~/.ssh .
If the workspace is deployed via Docker (Kubernetes will have a different update process), you need to remove the existing container (via docker rm ) and start a new one (via docker run ) with the newer workspace image. Make sure to use the same configuration, volume, name, and port. For example, a workspace (image version 0.8.7 ) was started with this command:
docker run -d
-p 8080:8080
--name "ml-workspace"
-v "/path/on/host:/workspace"
--env AUTHENTICATE_VIA_JUPYTER="mytoken"
--restart always
mltooling/ml-workspace:0.8.7
and needs to be updated to version 0.9.1 , you need to:
docker stop "ml-workspace" && docker rm "ml-workspace"docker run -d -p 8080:8080 --name "ml-workspace" -v "/path/on/host:/workspace" --env AUTHENTICATE_VIA_JUPYTER="mytoken" --restart always mltooling/ml-workspace:0.9.1 If you want to directly connect to the workspace via a VNC client (not using the noVNC webapp), you might be interested in changing certain VNC server configurations. To configure the VNC server, you can provide/overwrite the following environment variables at container start (via docker run option: --env ):
| Variable | Description | Défaut |
|---|---|---|
| VNC_PW | Password of VNC connection. This password only needs to be secure if the VNC server is directly exposed. If it is used via noVNC, it is already protected based on the configured authentication mechanism. | vncpassword |
| VNC_RESOLUTION | Default desktop resolution of VNC connection. When using noVNC, the resolution will be dynamically adapted to the window size. | 1600x900 |
| VNC_COL_DEPTH | Default color depth of VNC connection. | 24 |
Unfortunately, we currently do not support using a non-root user within the workspace. We plan to provide this capability and already started with some refactoring to allow this configuration. However, this still requires a lot more work, refactoring, and testing from our side.
Using root-user (or users with sudo permission) within containers is generally not recommended since, in case of system/kernel vulnerabilities, a user might be able to break out of the container and be able to access the host system. Since it is not very common to have such problematic kernel vulnerabilities, the risk of a severe attack is quite minimal. As explained in the official Docker documentation, containers (even with root users) are generally quite secure in preventing a breakout to the host. And compared to many other container use-cases, we actually want to provide the flexibility to the user to have control and system-level installation permissions within the workspace container.
The workspace comes preinstalled with various common tools to create isolated Python environments (virtual environments). The following sections provide a quick-intro on how to use these tools within the workspace. You can find information on when to use which tool here. Please refer to the documentation of the given tool for additional usage information.
venv (recommended):
To create a virtual environment via venv, execute the following commands:
# Create environment in the working directory
python -m venv my-venv
# Activate environment in shell
source ./my-venv/bin/activate
# Optional: Create Jupyter kernel for this environment
pip install ipykernel
python -m ipykernel install --user --name=my-venv --display-name= " my-venv ( $( python --version ) ) "
# Optional: Close enviornment session
deactivatepipenv (recommended):
To create a virtual environment via pipenv, execute the following commands:
# Create environment in the working directory
pipenv install
# Activate environment session in shell
pipenv shell
# Optional: Create Jupyter kernel for this environment
pipenv install ipykernel
python -m ipykernel install --user --name=my-pipenv --display-name= " my-pipenv ( $( python --version ) ) "
# Optional: Close environment session
exitvirtualenv :
To create a virtual environment via virtualenv, execute the following commands:
# Create environment in the working directory
virtualenv my-virtualenv
# Activate environment session in shell
source ./my-virtualenv/bin/activate
# Optional: Create Jupyter kernel for this environment
pip install ipykernel
python -m ipykernel install --user --name=my-virtualenv --display-name= " my-virtualenv ( $( python --version ) ) "
# Optional: Close environment session
deactivateconda :
To create a virtual environment via conda, execute the following commands:
# Create environment (globally)
conda create -n my-conda-env
# Activate environment session in shell
conda activate my-conda-env
# Optional: Create Jupyter kernel for this environment
python -m ipykernel install --user --name=my-conda-env --display-name= " my-conda-env ( $( python --version ) ) "
# Optional: Close environment session
conda deactivateTip: Shell Commands in Jupyter Notebooks:
If you install and use a virtual environment via a dedicated Jupyter Kernel and use shell commands within Jupyter (eg !pip install matplotlib ), the wrong python/pip version will be used. To use the python/pip version of the selected kernel, do the following instead:
import sys
!{ sys . executable } - m pip install matplotlibThe workspace provides three easy options to install different Python versions alongside the main Python instance: pyenv, pipenv (recommended), conda.
pipenv (recommended):
To install a different python version (eg 3.7.8 ) within the workspace via pipenv, execute the following commands:
# Install python vers
pipenv install --python=3.7.8
# Activate environment session in shell
pipenv shell
# Check python installation
python --version
# Optional: Create Jupyter kernel for this environment
pipenv install ipykernel
python -m ipykernel install --user --name=my-pipenv --display-name= " my-pipenv ( $( python --version ) ) "
# Optional: Close environment session
exitpyenv :
To install a different python version (eg 3.7.8 ) within the workspace via pyenv, execute the following commands:
# Install python version
pyenv install 3.7.8
# Make globally accessible
pyenv global 3.7.8
# Activate python version in shell
pyenv shell 3.7.8
# Check python installation
python3.7 --version
# Optional: Create Jupyter kernel for this python version
python3.7 -m pip install ipykernel
python3.7 -m ipykernel install --user --name=my-pyenv-3.7.8 --display-name= " my-pyenv (Python 3.7.8) "conda :
To install a different python version (eg 3.7.8 ) within the workspace via conda, execute the following commands:
# Create environment with python version
conda create -n my-conda-3.7 python=3.7.8
# Activate environment session in shell
conda activate my-conda-3.7
# Check python installation
python --version
# Optional: Create Jupyter kernel for this python version
pip install ipykernel
python -m ipykernel install --user --name=my-conda-3.7 --display-name= " my-conda ( $( python --version ) ) "
# Optional: Close environment session
conda deactivateTip: Shell Commands in Jupyter Notebooks:
If you install and use another Python version via a dedicated Jupyter Kernel and use shell commands within Jupyter (eg !pip install matplotlib ), the wrong python/pip version will be used. To use the python/pip version of the selected kernel, do the following instead:
import sys
!{ sys . executable } - m pip install matplotlib Certain desktop tools (eg, recent versions of Firefox) or libraries (eg, Pytorch - see Issues: 1, 2) might crash if the shared memory size ( /dev/shm ) is too small. The default shared memory size of Docker is 64MB, which might not be enough for a few tools. You can provide a higher shared memory size via the shm-size docker run option:
docker run --shm-size=2G mltooling/ml-workspace:0.13.2 In general, the performance of running code within Docker is nearly identical compared to running it directly on the machine. However, in case you have limited the container's CPU quota (as explained in this section), the container can still see the full count of CPU cores available on the machine and there is no technical way to prevent this. Many libraries and tools will use the full CPU count (eg, via os.cpu_count() ) to set the number of threads used for multiprocessing/-threading. This might cause the program to start more threads/processes than it can efficiently handle with the available CPU quota, which can tremendously slow down the overall performance. Therefore, it is important to set the available CPU count or the maximum number of threads explicitly to the configured CPU quota. The workspace provides capabilities to detect the number of available CPUs automatically, which are used to configure a variety of common libraries via environment variables such as OMP_NUM_THREADS or MKL_NUM_THREADS . It is also possible to explicitly set the number of available CPUs at container startup via the MAX_NUM_THREADS environment variable (see configuration section). The same environment variable can also be used to get the number of available CPUs at runtime.
Even though the automatic configuration capabilities of the workspace will fix a variety of inefficiencies, we still recommend configuring the number of available CPUs with all libraries explicitly. Par exemple:
import os
MAX_NUM_THREADS = int ( os . getenv ( "MAX_NUM_THREADS" ))
# Set in pytorch
import torch
torch . set_num_threads ( MAX_NUM_THREADS )
# Set in tensorflow
import tensorflow as tf
config = tf . ConfigProto (
device_count = { "CPU" : MAX_NUM_THREADS },
inter_op_parallelism_threads = MAX_NUM_THREADS ,
intra_op_parallelism_threads = MAX_NUM_THREADS ,
)
tf_session = tf . Session ( config = config )
# Set session for keras
import keras . backend as K
K . set_session ( tf_session )
# Set in sklearn estimator
from sklearn . linear_model import LogisticRegression
LogisticRegression ( n_jobs = MAX_NUM_THREADS ). fit ( X , y )
# Set for multiprocessing pool
from multiprocessing import Pool
with Pool ( MAX_NUM_THREADS ) as pool :
results = pool . map ( lst )If you encounter the following error within the container logs when starting the workspace, it will most likely not be possible to run the workspace on your hardware:
exited: nginx (terminated by SIGILL (core dumped); not expected)
The OpenResty/Nginx binary package used within the workspace requires to run on a CPU with SSE4.2 support (see this issue). Unfortunately, some older CPUs do not have support for SSE4.2 and, therefore, will not be able to run the workspace container. On Linux, you can check if your CPU supports SSE4.2 when looking into the cat /proc/cpuinfo flags section. If you encounter this problem, feel free to notify us by commenting on the following issue: #30.
Requirements : Docker and Act are required to be installed on your machine to execute the build process.
To simplify the process of building this project from scratch, we provide build-scripts - based on universal-build - that run all necessary steps (build, test, and release) within a containerized environment. To build and test your changes, execute the following command in the project root folder:
act -b -j buildUnder the hood it uses the build.py files in this repo based on the universal-build library. So, if you want to build it locally, you can also execute this command in the project root folder to build the docker container:
python build.py --makeFor additional script options:
python build.py --helpRefer to our contribution guides for more detailed information on our build scripts and development process.
Licensed Apache 2.0 . Created and maintained with ❤️ by developers from Berlin.