Esta es una aplicación interactiva basada en la web que envuelve el popular paquete ClusterProfiler que implementa métodos para analizar y visualizar perfiles funcionales de coordenadas genómicas, clústeres de genes y genes.
Los usuarios pueden cargar sus propios datos de expresión génica diferencial (DGE) de DESEQ2 o importar datos de la aplicación DESEQ2SHINY aguas arriba.
Esta aplicación permite un análisis de enriquecimiento de genes de genes rápidos y fácil (GSEA) de las vías GO-Terms y Kegg.
Está destinado a proporcionar una interfaz intuitiva para que los investigadores carguen fácilmente y realicen GSEA en los datos de RNA-seq de manera interactiva sin conocimiento previo de programación en R.
Las imágenes producidas incluyen gráficos de puntos, gráficos de red de categoría, gráficos de mapas de enriquecimiento, gráficos inducidos por GO, gráficos de GSEA y gráficos de vía Kegg enriquecidos utilizando el paquete PathView .
La aplicación sigue este tutorial
Consulte a continuación, por ejemplo, los gráficos de salida.
Esta aplicación acepta los siguientes tipos de datos de entrada:
Para fines de demostración, puede seleccionar "datos de ejemplo"
Puede seguir los pasos después para ejecutar el análisis que refleja el tutorial para familiarizarse con la aplicación.
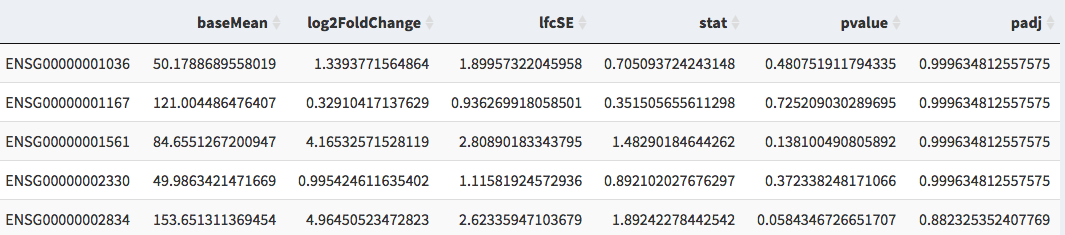
Un archivo .csv/.txt que contiene una tabla de datos de expresión génica diferencial (DGE)
P.ej. la salida de DESEQ2
El archivo puede ser delimitado por coma o pestaña
Las columnas requeridas son el nombre del gen/ID, el cambio de log2 plegable, los valores ajustados por P
Tendrá que seleccionar los nombres de la columna que coincidan con las columnas requeridas anteriormente
Para un archivo de muestra, haga clic aquí
Figura 2: Eg. Archivo de datos DGE
Los resultados de la salida se mostrarán y/o se puede descargar
Se incluyen varias formas de visualizaciones para GO/KEGG:
Guangchuangyu/clusterprofiler
Mohammed Khalfan - Tutorial de enriquecimiento del conjunto de genes