Это веб-интерактивное приложение, которое завершает популярный пакет кластернопрофилеров , который реализует методы для анализа и визуализации функциональных профилей геномных координат, генных и генных кластеров.
Пользователи могут загружать свои собственные данные дифференциальной экспрессии генов (DGE) из DESEQ2 или импортировать данные из приложения DESEQ2Shiny .
Это приложение обеспечивает быстрый и простой анализ обогащения наборов генов (GSEA) путей GO-Terms и KEGG.
Он предназначен для предоставления интуитивно понятного интерфейса для исследователей, чтобы легко загружать и выполнять GSEA на данных RNA-seq , без предварительных знаний по программированию в R.
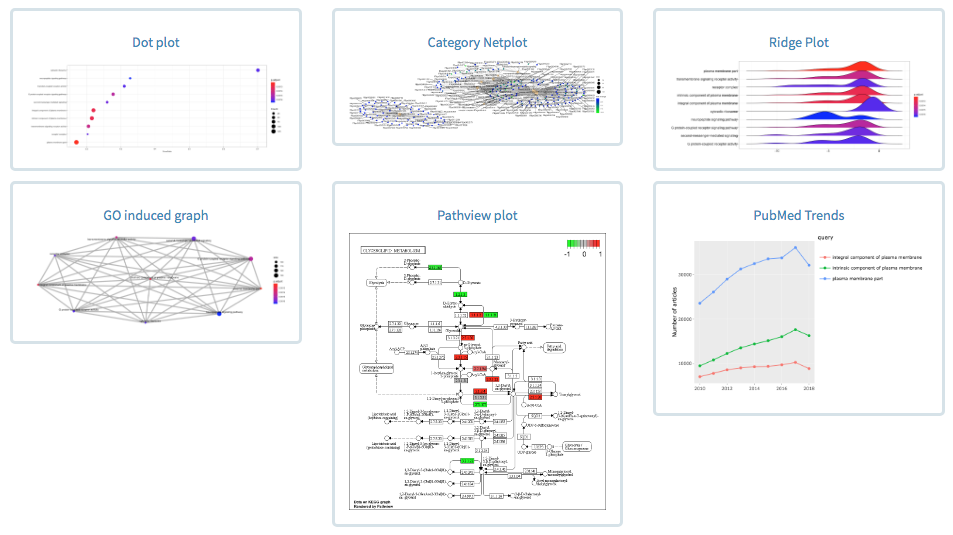
Изготовленные визуальные эффекты включают в себя точечные графики, участки категории, графики, графики карты обогащения, GO, графики, графики GSEA и обогащенные графики пути KEGG с использованием пакета PathView .
Приложение следует за этим учебником
См. Ниже, например, выводные графики.
Это приложение принимает следующие типы входных данных:
Для демонстрационных целей вы можете выбрать «Примерные данные»
После этого вы можете выполнить шаги, чтобы запустить анализ, отражая учебник, чтобы познакомиться с приложением
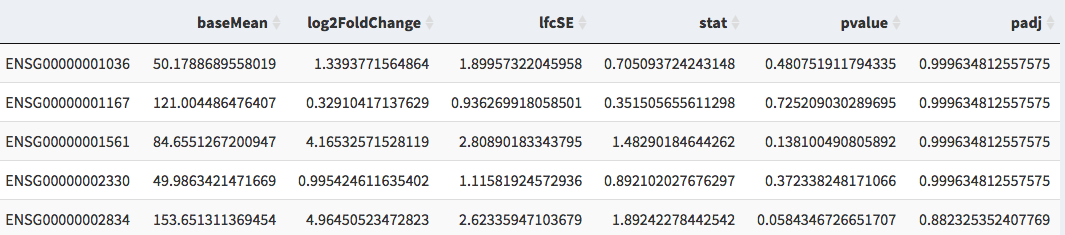
Файл .csv/.txt, который содержит данные о дифференциальной экспрессии генов (DGE)
Например. вывод DESEQ2
Файл может быть либо запятой, либо вкладкой.
Требуемые столбцы-это имя гена/ID, изменение сгиба Log2, P-поправки значения
Вам придется выбрать имена столбцов, которые соответствуют вышеуказанным столбцам
Для примера файла нажмите здесь
Рисунок 2: Например. DGE файл данных
Результаты выходных данных будут отображаться и/или можно загрузить
Различные формы визуализаций включены для GO/KEGG:
Guangchuangyu/ClusterProfiler
Мухаммед Халфан - Учебник по обогащению генов