Periplus está atualmente em Alpha e não está pronto para produção. O projeto está sob desenvolvimento ativo e ainda não é recomendado para uso em sistemas de produção.
O Periplus é um cache de banco de dados de vetor em memória de código aberto, criado na biblioteca de busca de similaridade vetorial da Meta FAISS. O projeto pode ser melhor considerado como "Redis for Vector Bathabases". Ele foi projetado para armazenar um subconjunto dinamicamente atualizado de uma grande coleção de vetores inteiramente na memória, cumprindo consultas sem interagir com outros nós no horário da consulta. Quando o Periplus recebe uma consulta, primeiro avalia se tem a parte relevante do índice em residência. Se isso acontecer, resolve a consulta com a resposta apropriada. Caso contrário, ele retorna uma falta de cache e deixa o consultor buscar os dados do banco de dados. Periplus não foi projetado para funcionar isoladamente. Em vez disso, destina -se a formar uma camada de cache modular e flexível para um banco de dados vetorial separado que forma a camada de persistência. O objetivo disso é ativar a menor latência e a escala horizontal fácil para aumentar a taxa de transferência. Para uma descrição mais detalhada da inspiração por trás do Periplus e como ele funciona, você pode ler o blog de anúncios: Apresentando Periplus: Uma nova abordagem para o cache do banco de dados vetorial.
O Periplus usa um índice de arquivo invertido (fertilização in vitro) como base para o gerenciamento de cache. Os índices de arquivo invertido particionam o espaço vetorial em células contíguas definidas por um conjunto de vetores centróides, onde cada célula é definida como a região que está mais próxima de seu centróide do que de qualquer outro centróide. As consultas são então resolvidas pela primeira vez calculando as distâncias do vetor de consulta ao conjunto de centróides e depois pesquisando apenas as células definidas pelos centróides mais próximos do N_Probe (Pesquise Hyperparameter). A Periplus aproveita isso mantendo um subconjunto dessas células em residência a qualquer momento e apenas resolvendo consultas relevantes para esse subconjunto enquanto rejeitam as que não são as erros de cache. O periplus carrega e despeja células inteiras de fertilização in vitro de cada vez para manter a integridade do índice e garantir a recuperação equivalente (em hits de cache) a um índice de fertilização in vitro padrão. As células de fertilização in vitro são carregadas consultando o banco de dados vetorial por meio de um proxy com uma lista de IDs de vetores que o periplus mantém para rastrear quais vetores ocupam quais células. Essas operações podem ser invocadas pelo usuário usando comandos de carga , pesquisa e despejo . Para detalhes, consulte a seção Comandos Periplus abaixo.
O periplus pode ser executado como um contêiner do docker ou pode ser construído a partir da fonte e executado como executável. No momento, nenhum binário oficial está disponível. Executar periplus como contêiner é a abordagem recomendada, mas ambos são opções viáveis.
Atualmente, a imagem do Docker suporta apenas arquiteturas AMD64. Essa restrição decorre da imagem base, mas mais arquiteturas serão apoiadas em um futuro próximo. Existem 2 maneiras de executar o Periplus como contêiner: Baixe a imagem oficial do Docker do DockerHub (recomendado) ou construa a imagem você mesmo. O primeiro passo em ambos os casos é instalar o Docker, se você ainda não o fez. As instruções para fazer isso podem ser encontradas aqui.
docker image pull qdl123/periplus:latest .docker run -p 3000:3000 qdl123/periplus:latest git clone https://github.com/QDL123/Periplus.gitcd <path-to-periplus-repo>/Periplusdocker build -t periplus-image .docker run -p 3000:3000 periplus-image .O Periplus usa o CMake para o sistema de construção. Ele espera que todas as dependências tenham binários pré-compilados instalados via Homebrew. O Homebrew é suportado por MacOS, Ubuntu e WSL se você estiver no Windows. O periplus foi construído no macOS/ARM64 e Ubuntu/AMD64. Todas as outras combinações de sistema operacional e arquitetura não são testadas. Para construir periplus a partir da fonte, siga as seguintes etapas:
brew install faiss curl cpr rapidjson libomp catch2 cmakegit clone https://github.com/QDL123/Periplus.gitcd <path-to-periplus-repo>/Peripluscmake -S . -B buildcmake --build build./build/periplus -p 3000 Qualquer sistema que use periplus consistirá em 4 componentes: o banco de dados vetorial, um proxy de banco de dados que permite que o periplus carregue dados do banco de dados, uma instância do Periplus e um aplicativo cliente.
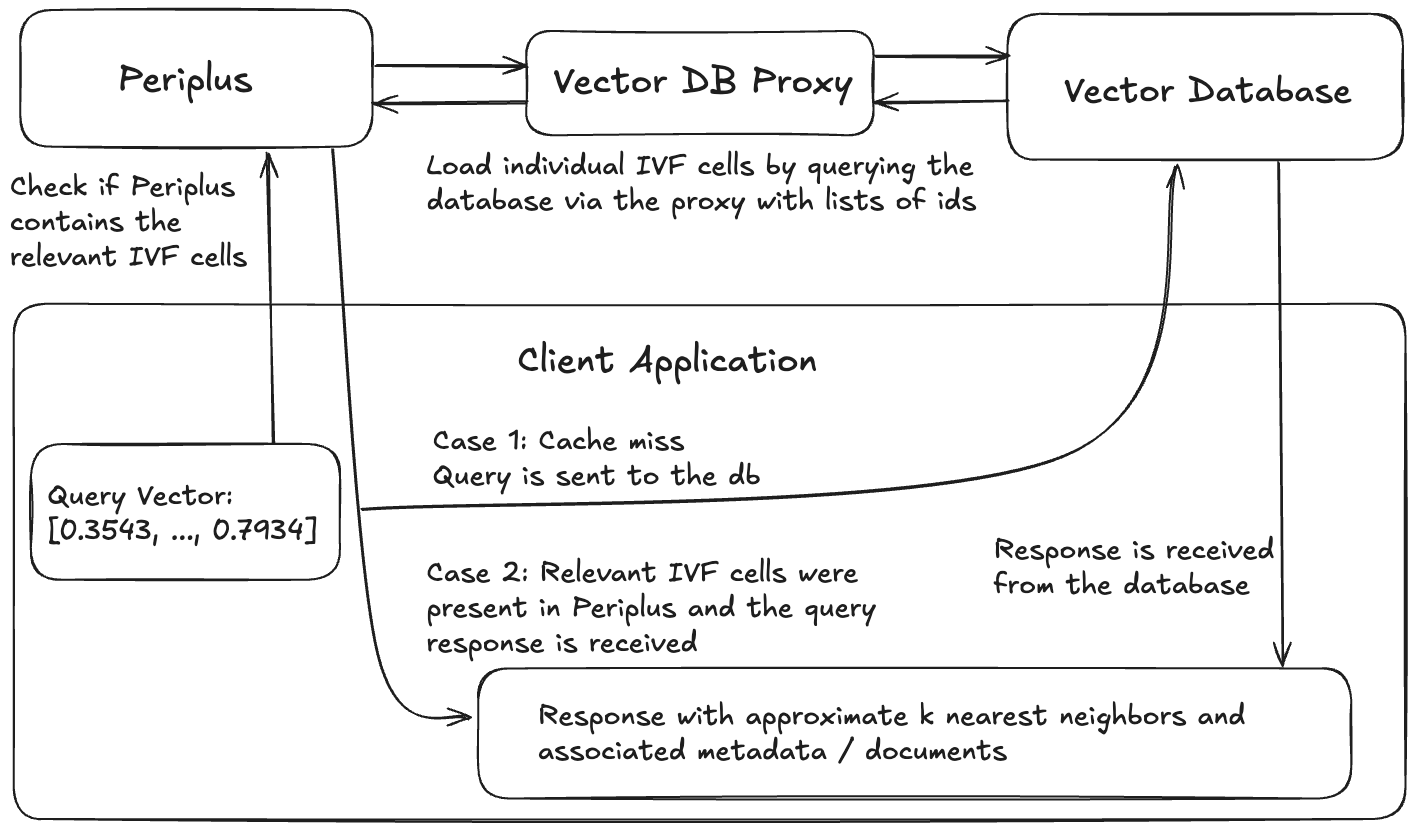
Exemplo de arquitetura de um aplicativo usando periplus.
Qualquer banco de dados vetorial que permita procurar dados por um identificador exclusivo (praticamente todos eles) funcionará. O Periplus foi projetado para ser mais benéfico ao trabalhar com colecos de vetor realmente grandes (escala bilhão), onde o índice deve viver no sistema de arquivos em oposição à RAM, embora isso não seja um requisito.
O objetivo do proxy do banco de dados Vector é fornecer uma interface consistente para o periplus interagir com o banco de dados do vetor. O proxy deve implementar uma interface REST que aceite solicitações de postagens do seguinte formulário:
URL: Isso é flexível e pode ser especificado pelo cliente Periplus.
Cabeçalhos: "Content-Type": "application/json
Corpo:
{
"ids" : [ " id-1 " , " id-2 " , " id-3 " ]
}Resposta:
{
"results" : [
{
"id" : " String " ,
"embedding" : [ 0.1 , 0.2 , 0.3 ],
"document" : " String " ,
"metdata" : " String "
}
]
}Para facilitar a implementação desse terminal, você pode usar o pacote Python Periplus-Proxy, que usa o FASTAPI para configurar tudo. Tudo o que o usuário precisa fazer é implementar a seguinte função e passá -la como um argumento:
async def fetch_ids(request: Query) -> QueryResult
Para detalhes sobre como fazer isso, você pode conferir o pacote periplus-proxy readme.md.
Siga as instruções acima para iniciar uma instância do Periplus.
Para interagir com sua instância do Periplus, use a biblioteca do cliente Periplus. Atualmente, apenas o Python é suportado. Para detalhes sobre a biblioteca do cliente, você pode ver que é readme.md.
from periplus_client import Periplus
# host, port
client = Periplus ( "localhost" , 13 )
# vector dimensionality, database proxy url, options: (nTotal)
await client . initialize ( d = d , db_url = url , options = { "nTotal" : 50000 })
training_data = [[ 0.43456 , ..., 0.38759 ], ...]
await client . train ( training_data )
ids = [ "0" , ..., "n" ]
embeddings = [[ 0.43456 , ..., 0.38759 ], ...]
await client . add ( ids = ids , embeddings = embeddings )
load_options = { "n_load" : 2 }
# query_vector, optional: options object
await client . load ([ embeddings [ 0 ]] load_options )
# k, query_vector
response = await client . search ( 5 , [ embeddings [ 0 ]])
print ( response )
'''
[ // Results for each of the n query vectors in xq
[ // K nearest neighbors to this corresponding index in the xq list
(
id="n",
embedding=[0.43456, ..., 0.38759],
document="",
metadata="{}"
),
...
],
...
]
'''
# query_vector
await client . evict ( embeddings [ 0 ])Congratulamo -nos com contribuições para o Periplus! Para aprender a começar, dê uma olhada no guia de contribuição.
Este projeto está licenciado sob a licença do MIT - consulte o arquivo de licença para obter detalhes.