repo นี้มีรูปแบบรหัสและข้อมูลของบทความของเรา: Linkbert: รูปแบบภาษาที่เตรียมไว้พร้อมลิงก์เอกสาร (ACL 2022) [PDF] [HuggingFace Models]
Linkbert เป็นรูปแบบภาษาที่ผ่านการฝึกอบรมใหม่ (การปรับปรุง Bert) ที่จับ ลิงก์เอกสาร เช่นการเชื่อมโยงหลายมิติและลิงก์การอ้างอิงเพื่อรวมความรู้ที่ครอบคลุมผ่านเอกสารหลายฉบับ โดยเฉพาะอย่างยิ่งมันถูกปรับสภาพโดยการให้อาหารเอกสารที่เชื่อมโยงในบริบทของโมเดลภาษาเดียวกันนอกเหนือจากการใช้เอกสารเดียวเช่นเดียวกับในเบิร์ต
Linkbert สามารถใช้แทน Drop-in สำหรับ Bert มันบรรลุประสิทธิภาพที่ดีขึ้นสำหรับงานการทำความเข้าใจภาษาทั่วไป (เช่นการจำแนกประเภทข้อความ) และยังมีประสิทธิภาพโดยเฉพาะอย่างยิ่งสำหรับงาน ที่ต้องใช้ความรู้มาก (เช่นการตอบคำถาม) และงาน ข้ามเอกสาร (เช่นความเข้าใจในการอ่านการดึงเอกสาร)
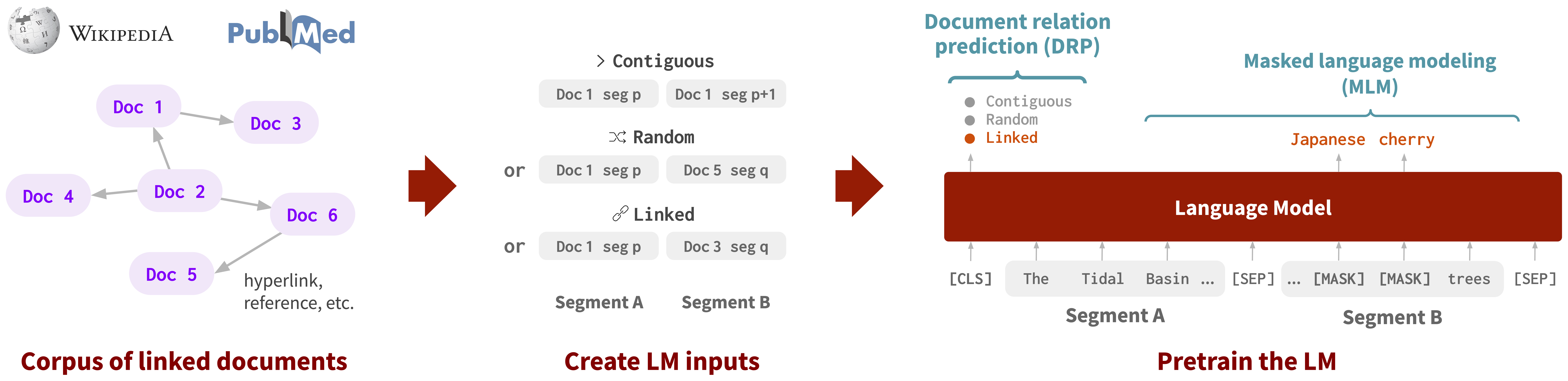
เราเปิดตัว Linkbert ที่ผ่านการฝึกฝน (-Base และ -ขนาดใหญ่) สำหรับทั้งโดเมนทั่วไปและโดเมนชีวการแพทย์ รุ่นเหล่านี้มีรูปแบบเดียวกับรุ่น HuggingFace Bert และคุณสามารถสลับกับรุ่น Linkbert ได้อย่างง่ายดาย
| แบบอย่าง | ขนาด | โดเมน | คลังแสง | ลิงค์ดาวน์โหลด (? HuggingFace) |
|---|---|---|---|---|
| Linkbert-base | พารามิเตอร์ 110m | ทั่วไป | Wikipedia กับไฮเปอร์ลิงก์ | Michiyasunaga/Linkbert-Base |
| ตัวใหญ่ Linkbert | พารามิเตอร์ 340m | ทั่วไป | Wikipedia กับไฮเปอร์ลิงก์ | Michiyasunaga/Linkbert-large |
| Biolinkbert-base | พารามิเตอร์ 110m | ชีวการแพทย์ | PubMed พร้อมลิงค์การอ้างอิง | Michiyasunaga/Biolinkbert-base |
| Biolinkbert-large | พารามิเตอร์ 340m | ชีวการแพทย์ | PubMed พร้อมลิงค์การอ้างอิง | Michiyasunaga/Biolinkbert-large |
เพื่อใช้โมเดลเหล่านี้ใน? Transformers:
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
model = AutoModel . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
inputs = tokenizer ( "Hello, my dog is cute" , return_tensors = "pt" )
outputs = model ( ** inputs ) ในการปรับแต่งโมเดลดูหัวข้อ 2 และ 3 ด้านล่าง เมื่อได้รับการปรับแต่งในงานดาวน์สตรีม Linkbert จะได้ผลลัพธ์ต่อไปนี้
เกณฑ์มาตรฐานทั่วไป (MRQA และกาว):
| Hotpotqa | เรื่องไม่สำคัญ | searchqa | NaturalQ | newsqa | ทีม | กาว | |
|---|---|---|---|---|---|---|---|
| F1 | F1 | F1 | F1 | F1 | F1 | คะแนน AVG | |
| เบิร์ตเบส | 76.0 | 70.3 | 74.2 | 76.5 | 65.7 | 88.7 | 79.2 |
| Linkbert-base | 78.2 | 73.9 | 76.8 | 78.3 | 69.3 | 90.1 | 79.6 |
| เบิร์ตขนาดใหญ่ | 78.1 | 73.7 | 78.3 | 79.0 | 70.9 | 91.1 | 80.7 |
| ตัวใหญ่ Linkbert | 80.8 | 78.2 | 80.5 | 81.0 | 72.6 | 92.7 | 81.1 |
เกณฑ์มาตรฐานด้านชีวการแพทย์ (Blurb, Medqa, MMLU, ฯลฯ ): Biolinkbert บรรลุถึงสถานะใหม่หรือไม่?
| คะแนนแจ้งเตือน | PubMedqa | BioAsq | medqa-usmle | |
|---|---|---|---|---|
| PubMedbert-base | 81.10 | 55.8 | 87.5 | 38.1 |
| Biolinkbert-base | 83.39 | 70.2 | 91.4 | 40.0 |
| Biolinkbert-large | 84.30 | 72.2 | 94.8 | 44.6 |
| ยา MMLU-Professional | |
|---|---|
| GPT-3 (175 params) | 38.7 |
| UnifiedQa (11b params) | 43.2 |
| Biolinkbert-large (340m params) | 50.7 |
เรียกใช้คำสั่งต่อไปนี้เพื่อสร้างสภาพแวดล้อม conda:
conda create -n linkbert python=3.8
source activate linkbert
pip install torch==1.10.1+cu113 -f https://download.pytorch.org/whl/cu113/torch_stable.html
pip install transformers==4.9.1 datasets==1.11.0 fairscale==0.4.0 wandb sklearn seqevalคุณสามารถดาวน์โหลดชุดข้อมูลที่ประมวลผลล่วงหน้าซึ่งเราประเมิน Linkbert จาก [ที่นี่] เพียงดาวน์โหลดไฟล์ zip นี้แล้วคลายซิป ซึ่งรวมถึง:
พวกเขาทั้งหมดถูกประมวลผลล่วงหน้าในรูปแบบชุดข้อมูล HuggingFace
หากคุณต้องการประมวลผลข้อมูลดิบล่วงหน้าคุณสามารถทำตามขั้นตอนต่อไปนี้:
scripts/download_raw_data.shscripts/preprocess_{mrqa,blurb,medqa,mmlu}.py เปลี่ยนไดเรกทอรีการทำงานเป็น src/ และทำตามคำแนะนำด้านล่างสำหรับแต่ละชุดข้อมูล
เพื่อปรับแต่งชุดข้อมูล MRQA (HOTPOTQA, Triviaqa, NaturalQuestions, SearchQa, NewsQa, Squad), Run Commands ที่ระบุไว้ใน run_examples_mrqa_linkbert-{base,large}.sh
เพื่อปรับแต่งชุดข้อมูลชีวการแพทย์ที่ประกาศแจ้ง (PubMedqa, BioAsq, HOC, Chemprot, Pico, ฯลฯ ), คำสั่งเรียกใช้ที่ระบุไว้ใน run_examples_blurb_biolinkbert-{base,large}.sh
หากต้องการปรับแต่งชุดข้อมูล Medqa-Usmle ให้เรียกใช้คำสั่งเรียกใช้ที่แสดงใน run_examples_medqa_biolinkbert-{base,large}.sh
ในการประเมินโมเดลที่ปรับแต่งเพิ่มเติมเกี่ยวกับยา MMLU-Professional ให้เรียกใช้คำสั่งที่แสดงที่ด้านล่างของ run_examples_medqa_biolinkbert-large.sh
นอกจากนี้เรายังจัดหาแผ่นงาน Codalab ซึ่งเราบันทึกการทดลองของเรา คุณอาจพบว่ามีประโยชน์สำหรับการจำลองการทดลองโดยใช้โมเดลรหัสข้อมูลและสภาพแวดล้อมเดียวกัน
หากคุณพบว่างานของเรามีประโยชน์โปรดอ้างอิงสิ่งต่อไปนี้:
@InProceedings { yasunaga2022linkbert ,
author = { Michihiro Yasunaga and Jure Leskovec and Percy Liang } ,
title = { LinkBERT: Pretraining Language Models with Document Links } ,
year = { 2022 } ,
booktitle = { Association for Computational Linguistics (ACL) } ,
}