В этом репошету предоставляет модель, код и данные нашей статьи: Linkbert: модели предварительного подготовки языков со ссылками на документы (ACL 2022). [PDF] [модели HuggingFace]]
Linkbert - это новая модель с предварительным языком (улучшение BERT), которая фиксирует ссылки на документы , такие как гиперссылки и ссылки на цитирование, включающие знания, которые охватывают несколько документов. В частности, он был предварительно проведен путем подачи связанных документов в тот же контекст модели языка, помимо использования одного документа, как в BERT.
Linkbert может использоваться в качестве замены BRET. Он достигает лучшей производительности для общих языковых задач (например, классификация текста), а также особенно эффективен для задач интенсивных знаний (например, ответа на вопрос) и задач по перекрестному документу (например, понимание прочитанного, поиск документа).
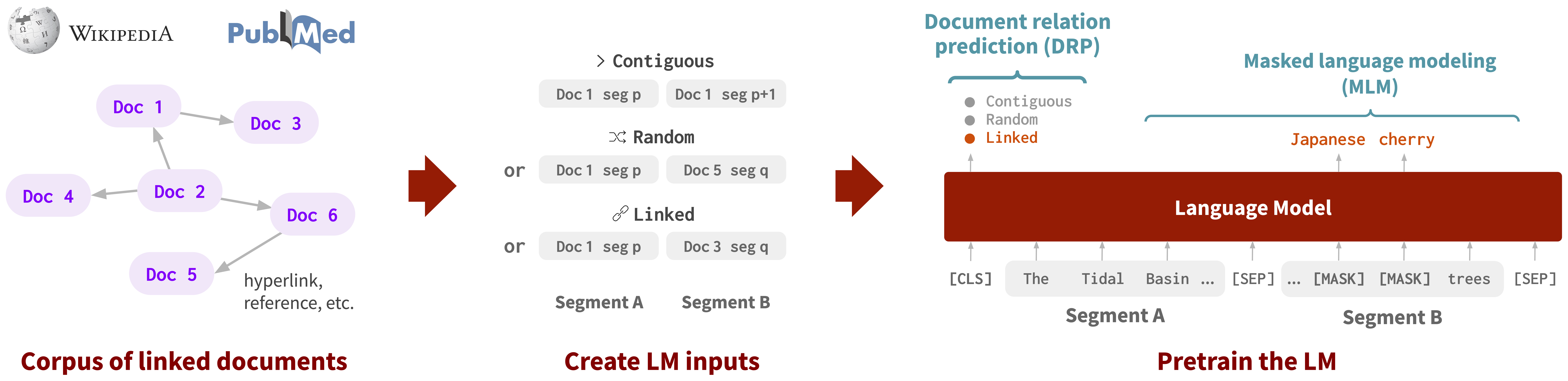
Мы выпускаем предварительно проведенный Linkbert (-base и -large размеры) как для общего домена, так и для биомедицинского домена. Эти модели имеют тот же формат, что и модели BERT Huggingface, и вы можете легко переключить их с помощью моделей Linkbert.
| Модель | Размер | Домен | Предварительный корпус | Ссылка загрузки (? HuggingFace) |
|---|---|---|---|---|
| Linkbert-Base | Параметры 110 м | Общий | Википедия с гиперссылками | Michiyasunaga/Linkbert-Base |
| Линкберт-Ларж | Параметры 340 м | Общий | Википедия с гиперссылками | Мичиясунага/Линкберт-Ларж |
| Biolinkbert-Base | Параметры 110 м | Биомедицина | Pubmed со ссылками на цитирование | Мичиясунага/Биолинкберт-База |
| Биолинкберт-широкий | Параметры 340 м | Биомедицина | Pubmed со ссылками на цитирование | Мичиясунага/Биолинкберт-Ларж |
Использовать эти модели в? Трансформеры:
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
model = AutoModel . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
inputs = tokenizer ( "Hello, my dog is cute" , return_tensors = "pt" )
outputs = model ( ** inputs ) Чтобы точно настроить модели, см. Раздел 2 и 3 ниже. Когда Линкберт достигает следующих результатов, настраивается на последующих задачах.
Общие тесты (MRQA и клей):
| Hotpotqa | Витривиака | Searchqa | Натуральный | Newsqa | Отряд | КЛЕЙ | |
|---|---|---|---|---|---|---|---|
| F1 | F1 | F1 | F1 | F1 | F1 | AVG SCORE | |
| Берт-баз | 76.0 | 70.3 | 74.2 | 76.5 | 65,7 | 88.7 | 79,2 |
| Linkbert-Base | 78.2 | 73,9 | 76.8 | 78.3 | 69,3 | 90.1 | 79,6 |
| Берт-широкий | 78.1 | 73,7 | 78.3 | 79,0 | 70,9 | 91.1 | 80.7 |
| Линкберт-Ларж | 80.8 | 78.2 | 80.5 | 81.0 | 72,6 | 92.7 | 81.1 |
Биомедицинские тесты (Blurb, Medqa, MMLU и т. Д.): Биолинкберт достигает нового современного?
| Оценка рекламы | PubMedqa | BioAsq | Medqa-Usmle | |
|---|---|---|---|---|
| PubMedbert-Base | 81.10 | 55,8 | 87.5 | 38.1 |
| Biolinkbert-Base | 83,39 | 70.2 | 91.4 | 40.0 |
| Биолинкберт-широкий | 84.30 | 72,2 | 94,8 | 44,6 |
| MMLU-Профессиональная медицина | |
|---|---|
| GPT-3 (175 параметров) | 38.7 |
| Unifiedqa (11b Params) | 43.2 |
| Биолинкберт-широкий (340 м) параметры) | 50,7 |
Запустите следующие команды для создания среды Conda:
conda create -n linkbert python=3.8
source activate linkbert
pip install torch==1.10.1+cu113 -f https://download.pytorch.org/whl/cu113/torch_stable.html
pip install transformers==4.9.1 datasets==1.11.0 fairscale==0.4.0 wandb sklearn seqevalВы можете скачать предварительные наборы данных, на которых мы оценили Linkbert из [здесь] . Просто загрузите этот zip -файл и раскрипируйте его. Это включает в себя:
Все они предварительно обработаны в формате набора данных HuggingFaceface.
Если вы хотите предварительно обработать необработанные данные с нуля, вы можете предпринять следующие шаги:
scripts/download_raw_data.shscripts/preprocess_{mrqa,blurb,medqa,mmlu}.py . Измените рабочий каталог на src/ и следуйте инструкциям ниже для каждого набора данных.
Для точной настройки для наборов данных MRQA (HotPotqa, Triviaqa, NaturalQuestions, SearchQA, Newsqa, Squad), Run Commands, перечисленные в run_examples_mrqa_linkbert-{base,large}.sh .
Для тонкой настройки наборов данных биомедиальных данных Blurb (Pubmedqa, BioASQ, HOC, ChemProt, PICO и т. Д.), Команды RUN, перечисленные в run_examples_blurb_biolinkbert-{base,large}.sh .
Чтобы точно настроить набор данных MedQA-USMLE, запустите команды, перечисленные в run_examples_medqa_biolinkbert-{base,large}.sh .
Чтобы оценить тонкую модель дополнительно на MMLU-профессиональную медицину, запустите команды, перечисленные внизу run_examples_medqa_biolinkbert-large.sh .
Мы также предоставляем лист Codalab, на котором мы записываем наши эксперименты. Вы можете найти его полезным для копирования экспериментов с использованием той же модели, кода, данных и среды.
Если вы найдете нашу работу полезной, пожалуйста, укажите следующее:
@InProceedings { yasunaga2022linkbert ,
author = { Michihiro Yasunaga and Jure Leskovec and Percy Liang } ,
title = { LinkBERT: Pretraining Language Models with Document Links } ,
year = { 2022 } ,
booktitle = { Association for Computational Linguistics (ACL) } ,
}