このレポは、私たちの論文のモデル、コード、およびデータを提供します:Linkbert:Document Links(ACL 2022)を使用して言語モデルを事前に削除します。 [PDF] [ハギングフェイスモデル]
Linkbertは、ハイパーリンクや引用リンクなどのドキュメントリンクをキャプチャして、複数のドキュメントにまたがる知識を含める新しい前提条件モデル(BERTの改善)です。具体的には、BERTのように単一のドキュメントを使用することに加えて、リンクされたドキュメントを同じ言語モデルのコンテキストにフィードすることで事前に処理されました。
Linkbertは、Bertのドロップイン代替品として使用できます。一般的な言語理解タスク(テキスト分類など)のパフォーマンスが向上し、知識集約型タスク(質問応答など)およびクロスドキュメントタスク(読解力、ドキュメントの検索など)にも特に効果的です。
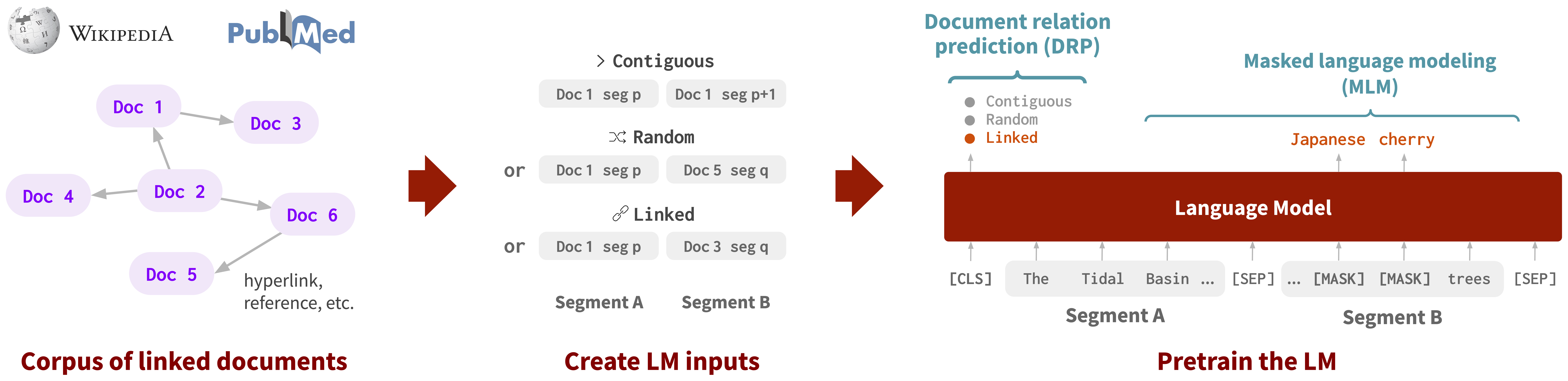
一般的なドメインと生物医学の両方のドメインの両方に対して、前処理されたLinkbert(-baseおよび-largeサイズ)をリリースします。これらのモデルは、Huggingface Bertモデルと同じ形式であり、Linkbertモデルで簡単に切り替えることができます。
| モデル | サイズ | ドメイン | 前脱子コーパス | ダウンロードリンク(?huggingface) |
|---|---|---|---|---|
| Linkbert-base | 110mパラメーター | 一般的な | ハイパーリンク付きウィキペディア | ミシヤスナガ/リンクベースベース |
| Linkbert-Large | 340mパラメーター | 一般的な | ハイパーリンク付きウィキペディア | ミシヤスナガ/リンクバートラージ |
| Biolinkbert-base | 110mパラメーター | バイオメディシン | 引用リンクを使用してPubMed | ミシヤスナガ/Biolinkbert-base |
| Biolinkbert-Large | 340mパラメーター | バイオメディシン | 引用リンクを使用してPubMed | ミシヤスナガ/Biolinkbert-Large |
これらのモデルを使用するには?トランス:
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
model = AutoModel . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
inputs = tokenizer ( "Hello, my dog is cute" , return_tensors = "pt" )
outputs = model ( ** inputs )モデルを微調整するには、以下のセクション2と3を参照してください。ダウンストリームタスクで微調整されると、Linkbertは次の結果を達成します。
一般的なベンチマーク(MRQAと接着剤):
| hotpotqa | Triviaqa | searchqa | NaturalQ | Newsqa | 分隊 | のり | |
|---|---|---|---|---|---|---|---|
| F1 | F1 | F1 | F1 | F1 | F1 | AVGスコア | |
| バートベース | 76.0 | 70.3 | 74.2 | 76.5 | 65.7 | 88.7 | 79.2 |
| Linkbert-base | 78.2 | 73.9 | 76.8 | 78.3 | 69.3 | 90.1 | 79.6 |
| バート・ラージ | 78.1 | 73.7 | 78.3 | 79.0 | 70.9 | 91.1 | 80.7 |
| Linkbert-Large | 80.8 | 78.2 | 80.5 | 81.0 | 72.6 | 92.7 | 81.1 |
生物医学のベンチマーク(宣伝文句、MEDQA、MMLUなど): Biolinkbertは新しい最先端を達成していますか?
| ぼやけたスコア | PubMedqa | Bioasq | Medqa-usmle | |
|---|---|---|---|---|
| PubMedbert-base | 81.10 | 55.8 | 87.5 | 38.1 |
| Biolinkbert-base | 83.39 | 70.2 | 91.4 | 40.0 |
| Biolinkbert-Large | 84.30 | 72.2 | 94.8 | 44.6 |
| MMLUプロフェッショナル医学 | |
|---|---|
| GPT-3(175 PARAMS) | 38.7 |
| unifiedqa(11b params) | 43.2 |
| Biolinkbert-Large(340m Params) | 50.7 |
次のコマンドを実行して、コンドラ環境を作成します。
conda create -n linkbert python=3.8
source activate linkbert
pip install torch==1.10.1+cu113 -f https://download.pytorch.org/whl/cu113/torch_stable.html
pip install transformers==4.9.1 datasets==1.11.0 fairscale==0.4.0 wandb sklearn seqeval[こちら]からLinkbertを評価した前処理されたデータセットをダウンロードできます。このzipファイルをダウンロードして、解凍します。これには次のものが含まれます。
それらはすべて、Huggingfaceデータセット形式で前処理されます。
生データをゼロから前処理したい場合は、次の手順を実行できます。
scripts/download_raw_data.shの指示に従って、元のソースから生データセットを最初にダウンロードしてくださいscripts/preprocess_{mrqa,blurb,medqa,mmlu}.pyを実行します。 作業ディレクトリをsrc/に変更し、各データセットの以下の指示に従ってください。
MRQAデータセット(Hotpotqa、triviaqa、naturalquestions、searchqa、newsqa、squad)を微調整するには、 run_examples_mrqa_linkbert-{base,large}.shにリストされているRunコマンド。
宣伝文学データセット(PubMedQA、BioASQ、HOC、ChemProt、PICOなど)を微調整するには、 run_examples_blurb_biolinkbert-{base,large}.shにリストされている実行コマンドを実行します。
medqa-usmleデータセットを微調整するには、 run_examples_medqa_biolinkbert-{base,large}.shにリストされているrunコマンドを実行します。
MMLUプロフェッショナル医学の微調整モデルをさらに評価するには、 run_examples_medqa_biolinkbert-large.shの下部にリストされているコマンドを実行します。
また、Codalabワークシートも提供しており、実験を記録します。同じモデル、コード、データ、環境を使用して実験を複製するのに役立つ場合があります。
私たちの作品が役立つ場合は、以下を引用してください。
@InProceedings { yasunaga2022linkbert ,
author = { Michihiro Yasunaga and Jure Leskovec and Percy Liang } ,
title = { LinkBERT: Pretraining Language Models with Document Links } ,
year = { 2022 } ,
booktitle = { Association for Computational Linguistics (ACL) } ,
}