Este repositório fornece o modelo, código e dados do nosso artigo: Linkbert: modelos de idioma pré -treinamento com links de documentos (ACL 2022). [PDF] [Modelos Huggingface]
Linkbert é um novo modelo de linguagem pré -teria (melhoria do BERT) que captura links de documentos, como hiperlinks e links de citação, para incluir conhecimento que abrange vários documentos. Especificamente, foi pré -levado pela alimentação de documentos vinculados no mesmo contexto de modelo de idioma, além de usar um único documento como em Bert.
O Linkbert pode ser usado como uma substituição para Bert. Ele atinge um melhor desempenho para tarefas gerais de compreensão de idiomas (por exemplo, classificação de texto) e também é particularmente eficaz para tarefas intensivas em conhecimento (por exemplo, resposta a perguntas) e tarefas entre documentos (por exemplo, compreensão de leitura, recuperação de documentos).
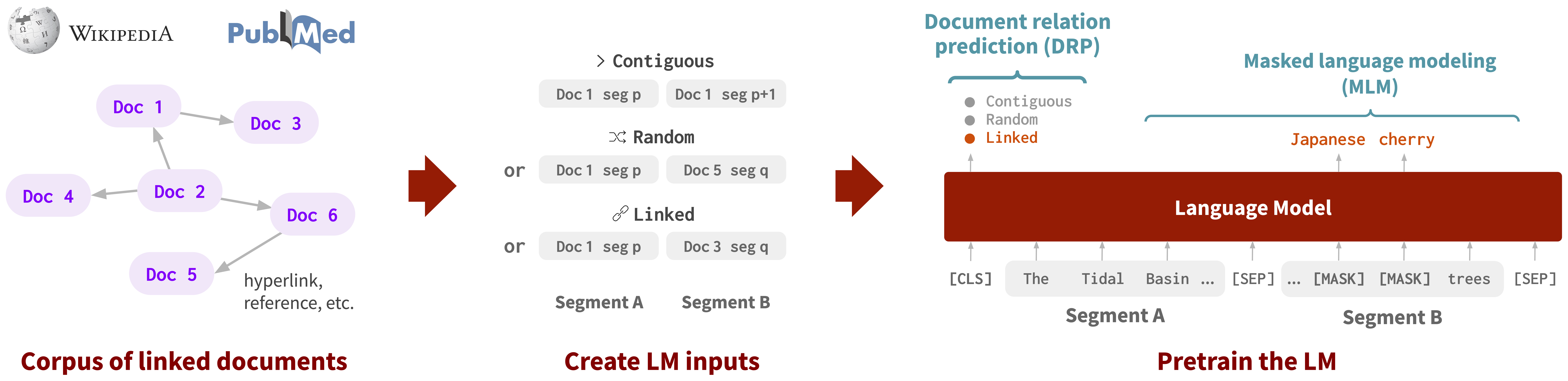
Lançamos os tamanhos de Linkbert (-Base e -Large pré -terenciosos) para o domínio geral e o domínio biomédico. Esses modelos têm o mesmo formato que os modelos Bert Huggingface e você pode alterá -los facilmente com os modelos Linkbert.
| Modelo | Tamanho | Domínio | Corpus pré -treinamento | Baixe o link (? Huggingface) |
|---|---|---|---|---|
| Linkbert-Base | Parâmetros de 110m | Em geral | Wikipedia com hiperlinks | Michiyasunaga/Linkbert-Base |
| Linkbert-Large | 340M parâmetros | Em geral | Wikipedia com hiperlinks | Michiyasunaga/Linkbert-Large |
| Biolinkbert-Base | Parâmetros de 110m | Biomedicina | PubMed com links de citação | Michiyasunaga/Biolinkbert-Base |
| Biolinkbert-Large | 340M parâmetros | Biomedicina | PubMed com links de citação | Michiyasunaga/Biolinkbert-Large |
Para usar esses modelos? Transformadores:
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
model = AutoModel . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
inputs = tokenizer ( "Hello, my dog is cute" , return_tensors = "pt" )
outputs = model ( ** inputs ) Para ajustar os modelos, consulte a Seção 2 e 3 abaixo. Quando ajustado em tarefas a jusante, o Linkbert alcança os seguintes resultados.
Benchmarks gerais (MRQA e cola):
| Hotpotqa | Triviaqa | SearchQa | Naturalq | Newsqa | Esquadrão | COLA | |
|---|---|---|---|---|---|---|---|
| F1 | F1 | F1 | F1 | F1 | F1 | Pontuação AVG | |
| Bert-base | 76.0 | 70.3 | 74.2 | 76.5 | 65.7 | 88.7 | 79.2 |
| Linkbert-Base | 78.2 | 73.9 | 76.8 | 78.3 | 69.3 | 90.1 | 79.6 |
| Bert-Large | 78.1 | 73.7 | 78.3 | 79.0 | 70.9 | 91.1 | 80.7 |
| Linkbert-Large | 80.8 | 78.2 | 80.5 | 81.0 | 72.6 | 92.7 | 81.1 |
Benchmarks biomédicos (Blurb, MedQA, MMLU, etc): Biolinkbert atinge o novo estado da arte?
| Pontuação da sinopse | PubMedqa | Bioasq | MedQA-USMLE | |
|---|---|---|---|---|
| PubMedbert-Base | 81.10 | 55.8 | 87.5 | 38.1 |
| Biolinkbert-Base | 83.39 | 70.2 | 91.4 | 40.0 |
| Biolinkbert-Large | 84.30 | 72.2 | 94.8 | 44.6 |
| Medicina MMLU-Profissional | |
|---|---|
| GPT-3 (175 params) | 38.7 |
| UnifiedQa (11b params) | 43.2 |
| Biolinkbert-Large (340m Params) | 50.7 |
Execute os seguintes comandos para criar um ambiente de conda:
conda create -n linkbert python=3.8
source activate linkbert
pip install torch==1.10.1+cu113 -f https://download.pytorch.org/whl/cu113/torch_stable.html
pip install transformers==4.9.1 datasets==1.11.0 fairscale==0.4.0 wandb sklearn seqevalVocê pode baixar os conjuntos de dados pré -processados nos quais avaliamos o Linkbert de [aqui] . Basta baixar este arquivo zip e descompactá -lo. Isso inclui:
Todos eles são pré -processados no formato do conjunto de dados HuggingFace.
Se você deseja pré -processar os dados brutos do zero, pode tomar as seguintes etapas:
scripts/download_raw_data.shscripts/preprocess_{mrqa,blurb,medqa,mmlu}.py . Altere o diretório de trabalho para src/ e siga as instruções abaixo para cada conjunto de dados.
Para ajustar os conjuntos de dados MRQA (Hotpotqa, Triviaqa, NaturalQuestions, SearchQa, NewsQA, Squad), execute comandos listados em run_examples_mrqa_linkbert-{base,large}.sh .
Para ajustar os conjuntos de dados biomediais da sinopse (PubMedqa, Bioasq, Hoc, ChemProt, Pico, etc.), execute comandos listados em run_examples_blurb_biolinkbert-{base,large}.sh .
Para ajustar o conjunto de dados MedQA-USMLE, execute os comandos listados em run_examples_medqa_biolinkbert-{base,large}.sh .
Para avaliar o modelo de ajuste fino adicionalmente na medicina mmlu-profissional, execute os comandos listados na parte inferior de run_examples_medqa_biolinkbert-large.sh .
Também fornecemos planilha do Codalab, na qual registramos nossos experimentos. Você pode achar útil para replicar os experimentos usando o mesmo modelo, código, dados e ambiente.
Se você achar útil nosso trabalho, cite o seguinte:
@InProceedings { yasunaga2022linkbert ,
author = { Michihiro Yasunaga and Jure Leskovec and Percy Liang } ,
title = { LinkBERT: Pretraining Language Models with Document Links } ,
year = { 2022 } ,
booktitle = { Association for Computational Linguistics (ACL) } ,
}