Este repositorio proporciona el modelo, el código y los datos de nuestro documento: Linkbert: modelos de lenguaje previo al ejercicio con enlaces de documentos (ACL 2022). [PDF] [Modelos Huggingface]
Linkbert es un nuevo modelo de lenguaje previo a la aparición (mejora de Bert) que captura enlaces de documentos como hipervínculos y enlaces de citas para incluir conocimiento que abarca múltiples documentos. Específicamente, fue provocado alimentando documentos vinculados en el mismo contexto del modelo de lenguaje, además de usar un solo documento que en Bert.
Linkbert se puede utilizar como reemplazo de berts. Logra un mejor rendimiento para las tareas generales de comprensión del idioma (por ejemplo, la clasificación de texto), y también es particularmente efectivo para las tareas intensivas en conocimiento (por ejemplo, respondiendo a preguntas) y tareas de documentos cruzados (por ejemplo, comprensión de lectura, recuperación de documentos).
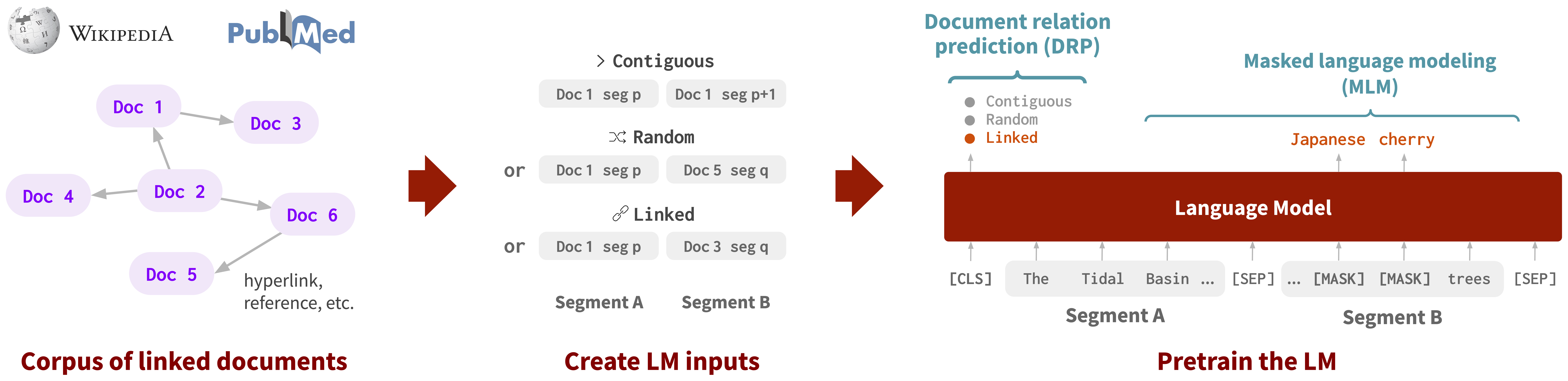
Liberamos el Linkbert previsto (tamaños de base y de base) tanto para el dominio general como para el dominio biomédico. Estos modelos tienen el mismo formato que los modelos Huggingface Bert, y puede cambiarlos fácilmente con modelos Linkbert.
| Modelo | Tamaño | Dominio | Corpus preventivo | Descargar enlace (? Huggingface) |
|---|---|---|---|---|
| Base de enlace | Parámetros de 110m | General | Wikipedia con hipervínculos | Michiyasunaga/Linkbert-Base |
| Linkbert-Large | 340m parámetros | General | Wikipedia con hipervínculos | Michiyasunaga/Linkbert-Large |
| Base de biolinkbert | Parámetros de 110m | Biomedicina | PubMed con enlaces de citas | Michiyasunaga/Biolinkbert-Base |
| Biolinkbert-large | 340m parámetros | Biomedicina | PubMed con enlaces de citas | Michiyasunaga/Biolinkbert-Large |
Para usar estos modelos en? Transformadores:
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
model = AutoModel . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
inputs = tokenizer ( "Hello, my dog is cute" , return_tensors = "pt" )
outputs = model ( ** inputs ) Para ajustar los modelos, consulte la Sección 2 y 3 a continuación. Cuando se ajusta las tareas aguas abajo, LinkBert logra los siguientes resultados.
Puntos de referencia generales (MRQA y pegamento):
| Hotpotqa | Triviaqa | Búsqueda | Naturalq | Newsqa | Equipo | PEGAMENTO | |
|---|---|---|---|---|---|---|---|
| F1 | F1 | F1 | F1 | F1 | F1 | Puntaje AVG | |
| Base | 76.0 | 70.3 | 74.2 | 76.5 | 65.7 | 88.7 | 79.2 |
| Base de enlace | 78.2 | 73.9 | 76.8 | 78.3 | 69.3 | 90.1 | 79.6 |
| Bernemacia | 78.1 | 73.7 | 78.3 | 79.0 | 70.9 | 91.1 | 80.7 |
| Linkbert-Large | 80.8 | 78.2 | 80.5 | 81.0 | 72.6 | 92.7 | 81.1 |
Bienchets biomedicales (Brurb, Medqa, MMLU, etc.): ¿ Biolinkbert alcanza un nuevo estado del arte?
| Puntaje | PubMedqa | Bioasq | Medqa-Usmle | |
|---|---|---|---|---|
| Base de pubmedbert | 81.10 | 55.8 | 87.5 | 38.1 |
| Base de biolinkbert | 83.39 | 70.2 | 91.4 | 40.0 |
| Biolinkbert-large | 84.30 | 72.2 | 94.8 | 44.6 |
| Medicina MMLU-Profesional | |
|---|---|
| GPT-3 (175 parámetros) | 38.7 |
| Unifiedqa (parámetros 11b) | 43.2 |
| Biolinkbert-Large (parámetros de 340m) | 50.7 |
Ejecute los siguientes comandos para crear un entorno de conda:
conda create -n linkbert python=3.8
source activate linkbert
pip install torch==1.10.1+cu113 -f https://download.pytorch.org/whl/cu113/torch_stable.html
pip install transformers==4.9.1 datasets==1.11.0 fairscale==0.4.0 wandb sklearn seqevalPuede descargar los conjuntos de datos preprocesados en los que evaluamos LinkBert de [aquí] . Simplemente descargue este archivo zip y descompite. Esto incluye:
Todos están preprocesados en el formato de conjunto de datos Huggingface.
Si desea preprocesar los datos sin procesar desde cero, puede tomar los siguientes pasos:
scripts/download_raw_data.shscripts/preprocess_{mrqa,blurb,medqa,mmlu}.py . Cambie el directorio de trabajo a src/ y siga las instrucciones a continuación para cada conjunto de datos.
Para ajustar los conjuntos de datos MRQA (hotpotqa, triviaqa, naturalQuestions, searchqa, newsqa, squad), ejecutar comandos enumerados en run_examples_mrqa_linkbert-{base,large}.sh .
Para ajustar para los conjuntos de datos biomediales de blurbs (PubMedqa, BioAsq, Hoc, ChemProt, Pico, etc.), ejecute comandos enumerados en run_examples_blurb_biolinkbert-{base,large}.sh .
Para ajustar para el conjunto de datos MEDQA-USMLE, ejecute los comandos enumerados en run_examples_medqa_biolinkbert-{base,large}.sh .
Para evaluar el modelo ajustado adicionalmente en MMLU-Professional Medicine, ejecute los comandos enumerados en la parte inferior de run_examples_medqa_biolinkbert-large.sh .
También proporcionamos hoja de trabajo Codalab, en la que registramos nuestros experimentos. Puede encontrar útil para replicar los experimentos utilizando el mismo modelo, código, datos y entorno.
Si encuentra útil nuestro trabajo, cite lo siguiente:
@InProceedings { yasunaga2022linkbert ,
author = { Michihiro Yasunaga and Jure Leskovec and Percy Liang } ,
title = { LinkBERT: Pretraining Language Models with Document Links } ,
year = { 2022 } ,
booktitle = { Association for Computational Linguistics (ACL) } ,
}