Dieses Repo bietet das Modell, Code und Daten unseres Papiers: Linkbert: Vorabsprachmodelle mit Dokumentlinks (ACL 2022). [PDF] [Huggingface -Modelle]
Linkbert ist ein neues vorgezogenes Sprachmodell (Verbesserung von Bert), das Dokumentlinks wie Hyperlinks und Zitierlinks erfasst, die Wissen über mehrere Dokumente hinweg umfassen. Insbesondere wurde es durch die Fütterung verknüpfter Dokumente in denselben Sprachmodellkontext vorgebracht, neben der Verwendung eines einzelnen Dokuments wie in Bert.
Linkbert kann als Drop-In-Ersatz für Bert verwendet werden. Es erzielt eine bessere Leistung für allgemeine Sprachverständnisaufgaben (z. B. Textklassifizierung) und ist auch besonders effektiv für wissensintensive Aufgaben (z. B. Beantwortung von Fragen) und Kreuzdokumentaufgaben (z. B. Leseverständnis, Dokumentenabruf).
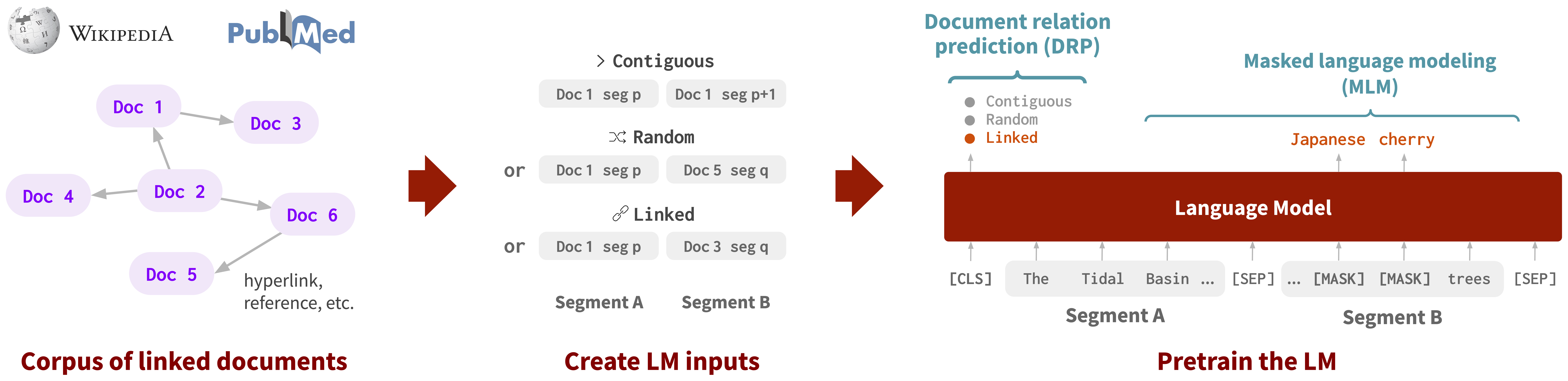
Wir geben den vorgefertigten Linkbert (-Base- und -letzungsgrößen) sowohl für die allgemeine Domäne als auch für die biomedizinische Domäne frei. Diese Modelle verfügen über das gleiche Format wie die Suggingface -Bert -Modelle und Sie können sie problemlos mit Linkbert -Modellen wechseln.
| Modell | Größe | Domain | Vorab -Korpus | Link herunterladen (? Umarmung) |
|---|---|---|---|---|
| Linkbert-Base | 110m Parameter | Allgemein | Wikipedia mit Hyperlinks | Michiyasunaga/Linkbert-Base |
| Linkbert-large | 340m Parameter | Allgemein | Wikipedia mit Hyperlinks | Michiyasunaga/Linkbert-Large |
| Biolinkbert-Base | 110m Parameter | Biomedizin | PubMed mit Zitierlinks | Michiyasunaga/Biolinkbert-Base |
| Biolinkbert-Large | 340m Parameter | Biomedizin | PubMed mit Zitierlinks | Michiyasunaga/Biolinkbert-Large |
Um diese Modelle zu verwenden? Transformatoren:
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
model = AutoModel . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
inputs = tokenizer ( "Hello, my dog is cute" , return_tensors = "pt" )
outputs = model ( ** inputs ) Um die Modelle zu optimieren, siehe Abschnitt 2 und 3 unten. Wenn Linkbert bei nachgeschalteten Aufgaben fein abgestimmt ist, erzielt er die folgenden Ergebnisse.
Allgemeine Benchmarks (MRQA und Kleber):
| Hotpotqa | Triviaqa | Searchqa | Naturalq | Newsqa | Kader | KLEBER | |
|---|---|---|---|---|---|---|---|
| F1 | F1 | F1 | F1 | F1 | F1 | AVG Score | |
| Bert-Base | 76.0 | 70.3 | 74.2 | 76,5 | 65.7 | 88.7 | 79,2 |
| Linkbert-Base | 78,2 | 73,9 | 76,8 | 78,3 | 69.3 | 90.1 | 79,6 |
| Bert-Large | 78.1 | 73.7 | 78,3 | 79,0 | 70,9 | 91.1 | 80.7 |
| Linkbert-large | 80.8 | 78,2 | 80.5 | 81.0 | 72.6 | 92.7 | 81.1 |
Biomedizinische Benchmarks (Klappentext, Medqa, MMLU usw.): Biolinkbert erreicht neue hochmoderne Art?
| Klappentext | PubMedqa | Bioasq | Medqa-usmle | |
|---|---|---|---|---|
| PubMedbert-Base | 81.10 | 55.8 | 87,5 | 38.1 |
| Biolinkbert-Base | 83.39 | 70,2 | 91.4 | 40.0 |
| Biolinkbert-Large | 84.30 | 72.2 | 94.8 | 44,6 |
| MMLU-professionelle Medizin | |
|---|---|
| GPT-3 (175 Parameter) | 38,7 |
| UnifiedQa (11B -Parameter) | 43.2 |
| Biolinkbert-Large (340 m Parames) | 50.7 |
Führen Sie die folgenden Befehle aus, um eine Conda -Umgebung zu erstellen:
conda create -n linkbert python=3.8
source activate linkbert
pip install torch==1.10.1+cu113 -f https://download.pytorch.org/whl/cu113/torch_stable.html
pip install transformers==4.9.1 datasets==1.11.0 fairscale==0.4.0 wandb sklearn seqevalSie können die vorverarbeiteten Datensätze herunterladen, auf denen wir Linkbert von [hier] bewertet haben. Laden Sie einfach diese ZIP -Datei herunter und entpacken Sie sie. Dies beinhaltet:
Sie sind alle im Dataset -Format von Huggingface -Dataset vorverarbeitet.
Wenn Sie die Rohdaten von Grund auf neu vorbereiten möchten, können Sie die folgenden Schritte ausführen:
scripts/download_raw_data.sh folgen.scripts/preprocess_{mrqa,blurb,medqa,mmlu}.py aus. Ändern Sie das Arbeitsverzeichnis in src/ und befolgen Sie die folgenden Anweisungen für jeden Datensatz.
Um die MRQA-Datensätze (Hotpotqa, Triviaqa, NaturalQuestions, Searchqa, Newsqa, Squad) zu optimieren, rennen Sie Befehle, die in run_examples_mrqa_linkbert-{base,large}.sh aufgeführt sind.
Um die biomedialen Datensätze der Blurby (PubMedqa, Bioasq, HOC, ChemProt, Pico usw.) zu optimieren, rennen Sie Befehle, die in run_examples_blurb_biolinkbert-{base,large}.sh aufgeführt sind.
Um für den Medqa-Usmle-Datensatz zu optimieren, führen Sie Befehle aus, die in run_examples_medqa_biolinkbert-{base,large}.sh aufgeführt sind.
Um das fein abgestimmte Modell zusätzlich auf der MMLU-professionellen Medizin zu bewerten, führen Sie die Befehle aus, die am Ende von run_examples_medqa_biolinkbert-large.sh aufgeführt sind.
Wir bieten auch Codalab -Arbeitsblatt an, auf dem wir unsere Experimente aufzeichnen. Möglicherweise finden Sie es nützlich, um die Experimente mit demselben Modell, demselben Code, der Daten und derselben Umgebung zu replizieren.
Wenn Sie unsere Arbeit hilfreich finden, geben Sie Folgendes an:
@InProceedings { yasunaga2022linkbert ,
author = { Michihiro Yasunaga and Jure Leskovec and Percy Liang } ,
title = { LinkBERT: Pretraining Language Models with Document Links } ,
year = { 2022 } ,
booktitle = { Association for Computational Linguistics (ACL) } ,
}