Ce repo fournit le modèle, le code et les données de notre article: Linkbert: Modèles de langue pré-formation avec des liens de document (ACL 2022). [PDF] [Modèles de câlins]
Linkbert est un nouveau modèle de langage pré-entraîné (amélioration de Bert) qui capture des liens de documents tels que les hyperliens et les liens de citation pour inclure des connaissances qui s'étend sur plusieurs documents. Plus précisément, il a été pré-entraîné en alimentant des documents liés dans le même contexte de modèle de langue, en plus d'utiliser un seul document comme dans Bert.
Linkbert peut être utilisé en remplacement de débarquement pour Bert. Il réalise de meilleures performances pour les tâches générales de compréhension des langues (par exemple, la classification du texte) et est également particulièrement efficace pour les tâches à forte intensité de connaissances (par exemple, répondant aux tâches de la question de la lecture (par exemple, compréhension de la lecture, récupération de documents).
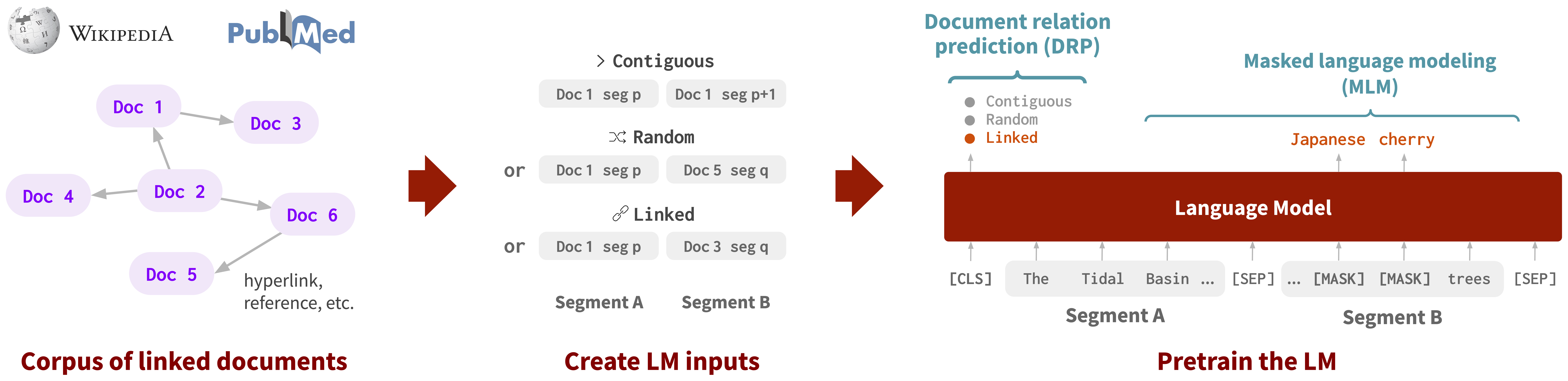
Nous libérons les tailles de linkbert pré-entraîneuses (-base et -llare) pour le domaine général et le domaine biomédical. Ces modèles ont le même format que les modèles BERT HUGGINGFACT, et vous pouvez facilement les commuter avec des modèles Linkbert.
| Modèle | Taille | Domaine | Corpus pré-formation | Télécharger le lien (? Huggingface) |
|---|---|---|---|---|
| Base de Linkbert | Paramètres de 110 m | Général | Wikipedia avec hyperliennes | michiyasunaga / linkbert-base |
| Linkbert-Large | Paramètres de 340 m | Général | Wikipedia avec hyperliennes | Michiyasunaga / Linkbert-Garg |
| Biolinkbert-base | Paramètres de 110 m | Biomédecine | PubMed avec des liens de citation | Michiyasunaga / Biolinkbert-base |
| Biolinkbert-Large | Paramètres de 340 m | Biomédecine | PubMed avec des liens de citation | Michiyasunaga / Biolinkbert-Large |
Pour utiliser ces modèles? Transformers:
from transformers import AutoTokenizer , AutoModel
tokenizer = AutoTokenizer . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
model = AutoModel . from_pretrained ( 'michiyasunaga/LinkBERT-large' )
inputs = tokenizer ( "Hello, my dog is cute" , return_tensors = "pt" )
outputs = model ( ** inputs ) Pour affiner les modèles, voir les sections 2 et 3 ci-dessous. Lorsqu'il est affiné sur les tâches en aval, Linkbert obtient les résultats suivants.
Benchmarks généraux (MRQA et Glue):
| Hotpotqa | Triviaqa | SearchQA | Naturelq | Newsqa | Équipe | COLLE | |
|---|---|---|---|---|---|---|---|
| F1 | F1 | F1 | F1 | F1 | F1 | Score AVG | |
| Bascule | 76.0 | 70.3 | 74.2 | 76.5 | 65.7 | 88.7 | 79.2 |
| Base de Linkbert | 78.2 | 73.9 | 76.8 | 78.3 | 69.3 | 90.1 | 79.6 |
| Bert-grand | 78.1 | 73.7 | 78.3 | 79.0 | 70.9 | 91.1 | 80.7 |
| Linkbert-Large | 80.8 | 78.2 | 80.5 | 81.0 | 72.6 | 92.7 | 81.1 |
Benchmarks biomédicaux (Blurb, Medqa, MMLU, etc.): Biolinkbert atteint une nouvelle pointe?
| Score de texte | PubMedqa | Bioasq | MEDQA-USMLE | |
|---|---|---|---|---|
| PubMedbert-base | 81.10 | 55.8 | 87.5 | 38.1 |
| Biolinkbert-base | 83.39 | 70.2 | 91.4 | 40.0 |
| Biolinkbert-Large | 84.30 | 72.2 | 94.8 | 44.6 |
| Médecine MMLU-professionnelle | |
|---|---|
| GPT-3 (175 params) | 38.7 |
| UnifiedQA (11b params) | 43.2 |
| Biolinkbert-Large (paramètres de 340 m) | 50.7 |
Exécutez les commandes suivantes pour créer un environnement conda:
conda create -n linkbert python=3.8
source activate linkbert
pip install torch==1.10.1+cu113 -f https://download.pytorch.org/whl/cu113/torch_stable.html
pip install transformers==4.9.1 datasets==1.11.0 fairscale==0.4.0 wandb sklearn seqevalVous pouvez télécharger les ensembles de données prétraités sur lesquels nous avons évalué Linkbert à partir de [ici] . Téléchargez simplement ce fichier zip et dézippez-le. Cela comprend:
Ils sont tous prétraités dans le format de jeu de données HuggingFace.
Si vous souhaitez prétraiter les données brutes à partir de zéro, vous pouvez prendre les étapes suivantes:
scripts/download_raw_data.shscripts/preprocess_{mrqa,blurb,medqa,mmlu}.py . Modifiez le répertoire de travail en src/ et suivez les instructions ci-dessous pour chaque ensemble de données.
Pour affiner pour les ensembles de données MRQA (Hotpotqa, Triviaqa, NaturalQuertions, SearchQA, Newsqa, Squad), les commandes d'exécution répertoriées dans run_examples_mrqa_linkbert-{base,large}.sh .
Pour affiner les ensembles de données biomédiaux Blurb (PubMedqa, Bioasq, Hoc, ChemProt, Pico, etc.), exécutez les commandes répertoriées dans run_examples_blurb_biolinkbert-{base,large}.sh .
Pour affiner pour l'ensemble de données Medqa-Usmle, exécutez les commandes répertoriées dans run_examples_medqa_biolinkbert-{base,large}.sh .
Pour évaluer le modèle affiné en plus sur MMLU-Professional Medicine, exécutez les commandes énumérées en bas de run_examples_medqa_biolinkbert-large.sh .
Nous fournissons également une feuille de calcul Codalab, sur laquelle nous enregistrons nos expériences. Vous pouvez le trouver utile pour reproduire les expériences en utilisant le même modèle, code, données et environnement.
Si vous trouvez notre travail utile, veuillez citer ce qui suit:
@InProceedings { yasunaga2022linkbert ,
author = { Michihiro Yasunaga and Jure Leskovec and Percy Liang } ,
title = { LinkBERT: Pretraining Language Models with Document Links } ,
year = { 2022 } ,
booktitle = { Association for Computational Linguistics (ACL) } ,
}