CBGBENCH: Complex Binding Graph Clachmark является эталоном для конструкции молекулы с генеративной целью.
Бумага [diffbp] | Бумага [D3FG] | Эталон
Примечание
Это официальный репозиторий кода статьи «Cbgbench: заполните пробел графика связывания белковой молекулы», который направлен на объединение конструкции молекулы с учетом целей с помощью отдельной реализации. До сих пор мы включили 7 методов, как показано ниже:
| Модель | Бумажная ссылка | GitHub |
|---|---|---|
| Pocket2mol | https://arxiv.org/abs/2205.07249 | https://github.com/pengxingang/pocket2mol |
| Graphbp | https://arxiv.org/abs/2204.09410 | https://github.com/divelab/graphbp |
| Diffsbdd | https://arxiv.org/abs/2210.13695 | https://github.com/arneschneuing/diffsbdd |
| Diffbp | https://arxiv.org/abs/2211.11214 | Вот официальная реализация. |
| TargetDiff | https://arxiv.org/abs/2303.03543 | https://github.com/guanjq/targetdiff |
| ФЛАГ | https://openreview.net/forum?id=rq13idf0f73 | https://github.com/zaixizhang/flag |
| D3FG | https://arxiv.org/abs/2306.13769 | Вот официальная реализация. |
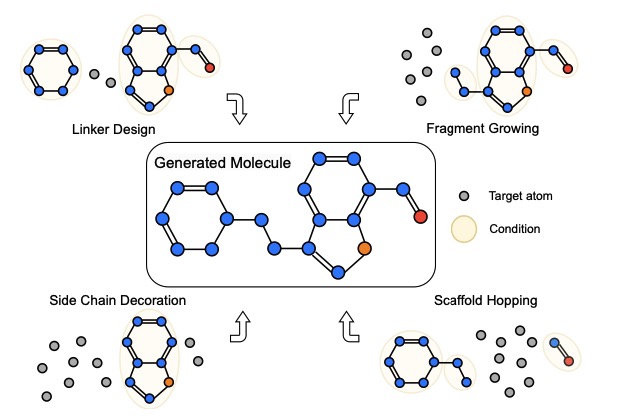
Эти модели изначально созданы для генерации молекул de novo , и мы расширяем больше задач, включая linker design , fragment growing , scaffold hopping и side chain decoration .
conda env create -f environment.yml
conda activate cbgbench
conda install pytorch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2 pytorch-cuda=11.8 -c pytorch -c nvidia
conda install pyg pytorch-scatter pytorch-cluster -c pyg
# install rdkit, efgs, obabel, etc.
pip install --use-pep517 EFGs
pip install biopython
pip install lxml
conda install rdkit openbabel tensorboard tqdm pyyaml easydict python-lmdb -c conda-forge
# install plip
mkdir tools
cd tools
git clone https://github.com/pharmai/plip.git
cd plip
python setup.py install
alias plip='python plip/plip/plipcmd.py'
cd ..
### Note that if there is an error in setup.py, it can be ignored as long as openbabel is installed.
# install docking tools
conda install -c conda-forge numpy swig boost-cpp sphinx sphinx_rtd_theme
python -m pip install git+https://github.com/Valdes-Tresanco-MS/AutoDockTools_py3
pip install meeko==0.1.dev3 scipy pdb2pqr vina
# If you are unable to install vina, you can try: conda install vina
# If you encounter the following error:
# ImportError: libtiff.so.5: cannot open shared object file: No such file or directory
# Please try the following steps to resolve it:
pip uninstall pillow
pip install pillow
(i) Загрузите обработанный набор данных с Google Drive. Для каждого набора данных это соответствует различным методам и задачам. Пожалуйста, обратитесь к таблице ниже, чтобы найти обработанные данные, которые вам нужно использовать. Обратите внимание, что для обучения D3FG это двухэтапная модель, которая сначала генерирует функциональные группы, а затем генерирует атомы линкера, поэтому мы назвали двухэтапную модель «d3fg_fg» и «d3fg_linker», и данные обучения отличаются для них.
Таблица: Имя файла данных, соответствующее различным методам и задачам.
| модель | задача | имя файла |
|---|---|---|
| Pocket2mol | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| Graphbp | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| Diffsbdd | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| Diffbp | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| TargetDiff | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| ФЛАГ | de novo | data/pl_arfg/crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb |
| D3fg_fg | de novo | data/pl_fg/crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb |
| D3fg_linker | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Pocket2mol | фрагмент | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Graphbp | фрагмент | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Diffsbdd | фрагмент | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Diffbp | фрагмент | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| TargetDiff | фрагмент | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Pocket2mol | линкер | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Graphbp | линкер | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Diffsbdd | линкер | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Diffbp | линкер | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| TargetDiff | линкер | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Pocket2mol | строительные леса | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| Graphbp | строительные леса | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| Diffsbdd | строительные леса | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| Diffbp | строительные леса | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| TargetDiff | строительные леса | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| Pocket2mol | боковая цепь | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Graphbp | боковая цепь | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Diffsbdd | боковая цепь | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Diffbp | боковая цепь | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| TargetDiff | боковая цепь | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
(ii) Скопировать наборы данных в ./data dir, в котором будет выглядеть полный каталог файлов данных
- CGBBench
- data
- pl
- crossdocked_name2id.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- pl_arfg
- crossdocked_name2id_arfuncgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb
- pl_decomp
- crossdocked_name2id_frag.pt
- crossdocked_name2id_linker.pt
- crossdocked_name2id_scaffold.pt
- crossdocked_name2id_sidechain.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb
- pl_fg
- crossdocked_name2id_funcgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb
(i) Скачать crossdocked_v1.1_rmsd1.0.tar.gz с TargetDiff Drive и скопируйте его на ./raw_data/ с
mkdir raw_data
tar -xzvf crossdocked_v1.1_rmsd1.0.tar.gz ./raw_data
(ii) Запустите следующее:
python ./scripts/extract_pockets.py --source raw_data/crossdocked_v1.1_rmsd1.0 --dest raw_data/crossdocked_v1.1_rmsd1.0_pocket10
(iii) При обучении набор данных будет подготовлен для каждой задачи. Пожалуйста, обратитесь к Training .
Кроме того, вы можете загрузить процессы Targets File case_study с Google Drive и скопировать его в каталог ./data , который приведет к указанию к
- CGBBench
- data
- case_study
- processed
- case_study_processed_fullatom.lmdb
- case_study_processed_funcgroup.lmdb
- case_study_name2id.pt
...
python train.py --config ./configs/{task}/train/{method}.yml --logdir ./logs/{task}/{method}
{Task} может быть заменен на denovo , linker , frag , scaffold и sidechain , и {Method} может быть заменен с помощью имени модели. В следующей таблице приведены подробные пары задач метода и замену.
Таблица: Пары методов задач, используемые для обучения с нуля.
| Метод | Задача | {method} + {task} |
|---|---|---|
| Pocket2mol | de novo | Pocket2mol + denovo |
| Graphbp | de novo | graphbp + denovo |
| Diffsbdd | de novo | diffsbdd + denovo |
| Diffbp | de novo | diffbp + denovo |
| TargetDiff | de novo | TargetDiff + denovo |
| ФЛАГ | de novo | Флаг + Деново |
| D3FG | de novo | d3fg_fg + denovo; d3fg_linker + denovo |
| Pocket2mol | Дизайн линкера | Pocket2mol + Linker |
| Graphbp | Дизайн линкера | graphbp + linker |
| Diffsbdd | Дизайн линкера | DiffsBdd + Линкер |
| Diffbp | Дизайн линкера | Diffbp + Linker |
| TargetDiff | Дизайн линкера | TargetDiff + Linker |
| Pocket2mol | Фрагмент растут | Pocket2mol + Frag |
| Graphbp | Фрагмент растут | graphbp + frag |
| Diffsbdd | Фрагмент растут | diffsbdd + frag |
| Diffbp | Фрагмент растут | diffbp + frag |
| TargetDiff | Фрагмент растут | TargetDiff + frag |
| Pocket2mol | Спрыгивание каркасов | Pocket2mol + каркас |
| Graphbp | Спрыгивание каркасов | GraphBP + каркас |
| Diffsbdd | Спрыгивание каркасов | Diffsbdd + Скасто |
| Diffbp | Спрыгивание каркасов | DIFFBP + СПАСК |
| TargetDiff | Спрыгивание каркасов | TargetDiff + Скафолд |
| Pocket2mol | Украшение боковой цепи | Pocket2mol + SideChain |
| Graphbp | Украшение боковой цепи | GraphBP + SideChain |
| Diffsbdd | Украшение боковой цепи | DiffsBdd + SideChain |
| Diffbp | Украшение боковой цепи | Diffbp + SideChain |
| TargetDiff | Украшение боковой цепи | TargetDiff + SideChain |
Обратите внимание, что D3FG и FLAG не совместимы с расширенными задачами, а D3FG использует стратегии 2-ступенчатых тренировок, поэтому, если вы хотите тренировать D3FG, вам нужно запустить:
python train.py --config ./configs/denovo/train/d3fg_fg.yml --logdir ./logs/denovo/d3fg_fg
python train.py --config ./configs/denovo/train/d3fg_linker.yml --logdir ./logs/denovo/d3fg_linker
TODO: таблица с предварительно подготовленными моделями
Загрузите предварительные контрольные точки .pt , которые вам нужны, и скопируйте их в ./logs/ , что приведет к следующей структуре каталога:
# TODO
- CBGBench
- logs
- denovo
- d3fg_fg
- pretrain
- checkpoints
- 951000.pt
- d3fg_linker
- pretrain
- checkpoints
- 4840000.pt
- diffbp
- pretrain
- checkpoints
- 4848000.pt
...
- frag
- diffbp
- pretrain
- checkpoints
- 476000.pt
...
...
После обучения модели вы можете нарисовать образцы из них на тестовых карманах, со следующим:
bash generate.sh --method {method} --task {task} --tag {tag} --checkpoint {ckpt_number}
В команде {method} и {task} пара может быть найдена в бывшей таблице на парах методов. {TAG} должен быть заменен selftrain или pretrain , в соответствии с используемыми вами контрольными точками. Если параметр - -checkpoint предоставляется без числа, он автоматически найдет последний файл .pt . Если число предоставлено, он будет использовать указанный файл контрольной точки.
Например, если вы хотите генерировать образцы в тестовом наборе для дизайна de novo с самостоятельной моделью TargetDiff , вы можете запустить:
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint
Или если вы хотите проверить контрольную точку 100000-й.
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint 100000
Обратите внимание, что D3FG использует стратегию генерации с двумя шагами, поэтому команда должна быть
bash generate.sh --method d3fg_fg --task denovo --tag {tag} --checkpoint # generate the functional groups
bash generate.sh --method d3fg_linker --task denovo --tag {tag} --checkpoint # generate the linkers
Запустите следующее:
cd evaluate_scripts
bash evaluate.sh --method {method} --task {task} --tag {tag}
Например, если вы сами обучили TargetDiff, бегите
bash evaluate.sh --method targetdiff --task denovo --tag selftrain
bash evaluate.sh --method targetdiff --task frag --tag selftrain
bash evaluate.sh --method targetdiff --task linker --tag selftrain
bash evaluate.sh --method targetdiff --task scaffold --tag selftrain
bash evaluate.sh --method targetdiff --task sidechain --tag selftrain
Или, если вы загрузили предварительные модели, пожалуйста, запустите
cd evaluate_scripts
bash bash evaluate.sh --method {method} --task {task} --tag pretrained
Используйте предварительные модели для гибкого генерации молекул на реальных целях.
Включите больше моделей, таких как 3DSBDD, DeCompdiff, Voxmol и Ligan.
Освободите предварительно проведенные модели, проверьте выполнимость и воспроизводительность после публикации бумаги.
Кодовая база инициализируется Хайтао Лин, а CBGBench V1 - как наша мотивация; Последующий запуск репозитория кода будет управляться Годзин Чжао и его команда технологий DP.
Если вы обнаружите, что репозиторий полезен для ваших исследований или проектов, пожалуйста, укажите на нашу эталонную бумагу в качестве ссылки:
@misc{lin2024cbgbench,
title={CBGBench: Fill in the Blank of Protein-Molecule Complex Binding Graph},
author={Haitao Lin and Guojiang Zhao and Odin Zhang and Yufei Huang and Lirong Wu and Zicheng Liu and Siyuan Li and Cheng Tan and Zhifeng Gao and Stan Z. Li},
year={2024},
eprint={2406.10840},
archivePrefix={arXiv},
}
И наши прежние работы DIFFBP и D3FG:
@misc{lin2024diffbpgenerativediffusion3d,
title={DiffBP: Generative Diffusion of 3D Molecules for Target Protein Binding},
author={Haitao Lin and Yufei Huang and Odin Zhang and Siqi Ma and Meng Liu and Xuanjing Li and Lirong Wu and Jishui Wang and Tingjun Hou and Stan Z. Li},
year={2024},
eprint={2211.11214},
archivePrefix={arXiv},
primaryClass={q-bio.BM},
url={https://arxiv.org/abs/2211.11214},
}
@inproceedings{
lin2023functionalgroupbased,
title={Functional-Group-Based Diffusion for Pocket-Specific Molecule Generation and Elaboration},
author={Haitao Lin and Yufei Huang and Odin Zhang and Yunfan Liu and Lirong Wu and Siyuan Li and Zhiyuan Chen and Stan Z. Li},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=lRG11M91dx}
}
Этот проект лицензирован в соответствии с условиями лицензии GPL-3.0.