CBGBEnch: A referência de gráficos de ligação complexa é uma referência para o design generativo da molécula com reconhecimento de destino.
Papel [diffbp] | Papel [d3fg] | Benchmark
Observação
Este é o repositório oficial de código do artigo 'CBGBEnch: preencha o espaço em branco do gráfico de ligação da molécula de proteína', que visa unificar o design da molécula com reconhecimento de destino com implementação de código único. Até agora, incluímos 7 métodos como mostrado abaixo:
| Modelo | Link em papel | Github |
|---|---|---|
| Pocket2mol | https://arxiv.org/abs/2205.07249 | https://github.com/pengxingang/pocket2mol |
| Graphbp | https://arxiv.org/abs/2204.09410 | https://github.com/divelab/graphbp |
| DIFFSBDD | https://arxiv.org/abs/2210.13695 | https://github.com/arneschneuing/diffsbdd |
| Diffbp | https://arxiv.org/abs/2211.11214 | Aqui está a implementação oficial. |
| TargetDiff | https://arxiv.org/abs/2303.03543 | https://github.com/guanjq/targetdiff |
| BANDEIRA | https://openreview.net/forum?id=rq13idf0f73 | https://github.com/zaixizhang/flag |
| D3fg | https://arxiv.org/abs/2306.13769 | Aqui está a implementação oficial. |
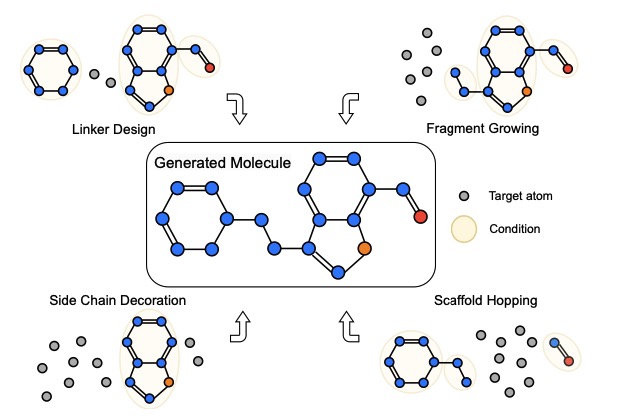
Esses modelos são inicialmente estabelecidos para a geração de moléculas de novo e estendemos mais tarefas, incluindo linker design , fragment growing , scaffold hopping e side chain decoration .
conda env create -f environment.yml
conda activate cbgbench
conda install pytorch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2 pytorch-cuda=11.8 -c pytorch -c nvidia
conda install pyg pytorch-scatter pytorch-cluster -c pyg
# install rdkit, efgs, obabel, etc.
pip install --use-pep517 EFGs
pip install biopython
pip install lxml
conda install rdkit openbabel tensorboard tqdm pyyaml easydict python-lmdb -c conda-forge
# install plip
mkdir tools
cd tools
git clone https://github.com/pharmai/plip.git
cd plip
python setup.py install
alias plip='python plip/plip/plipcmd.py'
cd ..
### Note that if there is an error in setup.py, it can be ignored as long as openbabel is installed.
# install docking tools
conda install -c conda-forge numpy swig boost-cpp sphinx sphinx_rtd_theme
python -m pip install git+https://github.com/Valdes-Tresanco-MS/AutoDockTools_py3
pip install meeko==0.1.dev3 scipy pdb2pqr vina
# If you are unable to install vina, you can try: conda install vina
# If you encounter the following error:
# ImportError: libtiff.so.5: cannot open shared object file: No such file or directory
# Please try the following steps to resolve it:
pip uninstall pillow
pip install pillow
(i) Faça o download do conjunto de dados processado do Google Drive. Para cada conjunto de dados, corresponde a diferentes métodos e tarefas. Consulte a tabela abaixo para encontrar os dados processados que você precisa usar. Observe que, para treinar D3FG, é um modelo de dois estágios que gera primeiro grupos funcionais e gera átomos de ligação, por isso nomeamos o modelo de dois estágios 'D3FG_FG' e 'D3FG_Linker' e os dados de treinamento são diferentes para eles.
Tabela: o nome do arquivo de dados correspondente a diferentes métodos e tarefas.
| modelo | tarefa | Nome do arquivo |
|---|---|---|
| Pocket2mol | de Novo | Data/PL/Crossdocked_V1.1_RMSD1.0_Pocket10_Processed_fullatom.lmdb |
| Graphbp | de Novo | Data/PL/Crossdocked_V1.1_RMSD1.0_Pocket10_Processed_fullatom.lmdb |
| DIFFSBDD | de Novo | Data/PL/Crossdocked_V1.1_RMSD1.0_Pocket10_Processed_fullatom.lmdb |
| Diffbp | de Novo | Data/PL/Crossdocked_V1.1_RMSD1.0_Pocket10_Processed_fullatom.lmdb |
| TargetDiff | de Novo | Data/PL/Crossdocked_V1.1_RMSD1.0_Pocket10_Processed_fullatom.lmdb |
| BANDEIRA | de Novo | data/pl_arfg/crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb |
| D3fg_fg | de Novo | data/pl_fg/crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb |
| D3fg_linker | de Novo | Data/PL/Crossdocked_V1.1_RMSD1.0_Pocket10_Processed_linker.lmdb |
| Pocket2mol | fragmento | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Graphbp | fragmento | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| DIFFSBDD | fragmento | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Diffbp | fragmento | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| TargetDiff | fragmento | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Pocket2mol | Linker | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Graphbp | Linker | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| DIFFSBDD | Linker | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Diffbp | Linker | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| TargetDiff | Linker | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Pocket2mol | andaime | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| Graphbp | andaime | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| DIFFSBDD | andaime | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| Diffbp | andaime | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| TargetDiff | andaime | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| Pocket2mol | Corrente lateral | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Graphbp | Corrente lateral | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| DIFFSBDD | Corrente lateral | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Diffbp | Corrente lateral | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| TargetDiff | Corrente lateral | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
(ii) copiar os conjuntos de dados para ./data dir, em que o diretório completo de arquivos de dados será como
- CGBBench
- data
- pl
- crossdocked_name2id.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- pl_arfg
- crossdocked_name2id_arfuncgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb
- pl_decomp
- crossdocked_name2id_frag.pt
- crossdocked_name2id_linker.pt
- crossdocked_name2id_scaffold.pt
- crossdocked_name2id_sidechain.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb
- pl_fg
- crossdocked_name2id_funcgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb
(i) Faça o download crossdocked_v1.1_rmsd1.0.tar.gz da TargetDiff Drive e copie -o para ./raw_data/ com
mkdir raw_data
tar -xzvf crossdocked_v1.1_rmsd1.0.tar.gz ./raw_data
(ii) execute o seguinte:
python ./scripts/extract_pockets.py --source raw_data/crossdocked_v1.1_rmsd1.0 --dest raw_data/crossdocked_v1.1_rmsd1.0_pocket10
(iii) No treinamento, o conjunto de dados será preparado para cada tarefa. Por favor, consulte o Training .
Além disso, você pode baixar os processos alvos de arquivo case_study do google drive e copiá -lo para o diretório ./data , que liderará o diretor para
- CGBBench
- data
- case_study
- processed
- case_study_processed_fullatom.lmdb
- case_study_processed_funcgroup.lmdb
- case_study_name2id.pt
...
python train.py --config ./configs/{task}/train/{method}.yml --logdir ./logs/{task}/{method}
{Task} pode ser substituído por denovo , linker , frag , scaffold e sidechain , e {Method} pode ser substituído pelo nome do modelo. A tabela a seguir fornece os pares detalhados do método-tarefa e a substituição.
Tabela: pares de tarefas de método usados para treinar do zero.
| Método | Tarefa | {método} + {tarefa} |
|---|---|---|
| Pocket2mol | de Novo | Pocket2mol + Denovo |
| Graphbp | de Novo | Graphbp + Denovo |
| DIFFSBDD | de Novo | DIFFSBDD + DENOVO |
| Diffbp | de Novo | diffbp + denovo |
| TargetDiff | de Novo | TargetDiff + Denovo |
| BANDEIRA | de Novo | Flag + Denovo |
| D3fg | de Novo | d3fg_fg + denovo; D3FG_LINKER + DENOVO |
| Pocket2mol | Design de vinculadores | Pocket2mol + ligante |
| Graphbp | Design de vinculadores | GraphBP + Linker |
| DIFFSBDD | Design de vinculadores | DIFFSBDD + LINKER |
| Diffbp | Design de vinculadores | Linker diffbp + |
| TargetDiff | Design de vinculadores | TargetDiff + Linker |
| Pocket2mol | Fragmento crescendo | Pocket2mol + frag |
| Graphbp | Fragmento crescendo | Graphbp + frag |
| DIFFSBDD | Fragmento crescendo | DIFFSBDD + frag |
| Diffbp | Fragmento crescendo | diffbp + frag |
| TargetDiff | Fragmento crescendo | TargetDiff + frag |
| Pocket2mol | pulando de andaime | Pocket2mol + andaime |
| Graphbp | pulando de andaime | Graphbp + andaime |
| DIFFSBDD | pulando de andaime | DIFFSBDD + Scaffold |
| Diffbp | pulando de andaime | DIFFBP + Scaffold |
| TargetDiff | pulando de andaime | TargetDiff + andaime |
| Pocket2mol | decoração da cadeia lateral | Pocket2mol + Sidechain |
| Graphbp | decoração da cadeia lateral | GraphBP + Sidechain |
| DIFFSBDD | decoração da cadeia lateral | DIFFSBDD + Sidechain |
| Diffbp | decoração da cadeia lateral | Diffbp + Sidechain |
| TargetDiff | decoração da cadeia lateral | TargetDiff + Sidechain |
Observe que o D3FG e o Flag não são compatíveis com as tarefas estendidas, e o D3FG utiliza estratégias de treinamento em 2 estágios; portanto, se você deseja treinar o D3FG, precisa executar:
python train.py --config ./configs/denovo/train/d3fg_fg.yml --logdir ./logs/denovo/d3fg_fg
python train.py --config ./configs/denovo/train/d3fg_linker.yml --logdir ./logs/denovo/d3fg_linker
TODO: Tabela com modelos pré -teriados
Faça o download dos pontos de verificação .pt pré -teremam que você precisa e copie -os para ./logs/ , o que levará à seguinte estrutura de diretório:
# TODO
- CBGBench
- logs
- denovo
- d3fg_fg
- pretrain
- checkpoints
- 951000.pt
- d3fg_linker
- pretrain
- checkpoints
- 4840000.pt
- diffbp
- pretrain
- checkpoints
- 4848000.pt
...
- frag
- diffbp
- pretrain
- checkpoints
- 476000.pt
...
...
Depois que o modelo é treinado, você pode desenhar amostras deles nos bolsos de teste, com o seguinte:
bash generate.sh --method {method} --task {task} --tag {tag} --checkpoint {ckpt_number}
No comando, o par {Method} e {Task} podem ser encontrados na tabela anterior em pares de tarefas de método. {tag} deve ser substituído por selftrain ou pretrain , de acordo com os pontos de verificação que você usa. Se o parâmetro -Checkpoint for fornecido sem um número, ele encontrará automaticamente o arquivo .pt mais recente. Se um número for fornecido, ele usará o arquivo de ponto de verificação especificado.
Por exemplo, se você deseja gerar amostras no conjunto de testes para design de novo com modelo de TargetDiff auto-treinado , você pode executar:
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint
Ou se você deseja testar o ponto de verificação dos 100000 tés.
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint 100000
Observe que D3FG usa uma estratégia de geração de duas etapas, então o comando deve ser
bash generate.sh --method d3fg_fg --task denovo --tag {tag} --checkpoint # generate the functional groups
bash generate.sh --method d3fg_linker --task denovo --tag {tag} --checkpoint # generate the linkers
Execute o seguinte:
cd evaluate_scripts
bash evaluate.sh --method {method} --task {task} --tag {tag}
Por exemplo, se você treinou o TargetDiff sozinho, corra
bash evaluate.sh --method targetdiff --task denovo --tag selftrain
bash evaluate.sh --method targetdiff --task frag --tag selftrain
bash evaluate.sh --method targetdiff --task linker --tag selftrain
bash evaluate.sh --method targetdiff --task scaffold --tag selftrain
bash evaluate.sh --method targetdiff --task sidechain --tag selftrain
Ou se você baixou modelos pré -tenhados, corra
cd evaluate_scripts
bash bash evaluate.sh --method {method} --task {task} --tag pretrained
Use os modelos pré-treinados para gerar moléculas nos alvos do mundo real com flexibilidade.
Inclua mais modelos, como 3DSBDD, Decompdiff, Voxmol e Ligan.
Libere os modelos pré -treinados, teste a viabilidade e a reproducividade depois que o artigo for publicado.
A base de código é inicializada por Haitao Lin, com o CBGBEnch V1 como nossa motivação; A execução subsequente do repositório de código será gerenciada por Guojiang Zhao e sua equipe de tecnologia DP.
Se você achar que o repositório é útil para sua pesquisa ou projetos, cite em nosso artigo de referência como referência:
@misc{lin2024cbgbench,
title={CBGBench: Fill in the Blank of Protein-Molecule Complex Binding Graph},
author={Haitao Lin and Guojiang Zhao and Odin Zhang and Yufei Huang and Lirong Wu and Zicheng Liu and Siyuan Li and Cheng Tan and Zhifeng Gao and Stan Z. Li},
year={2024},
eprint={2406.10840},
archivePrefix={arXiv},
}
E nossas antigas obras de diffbp e d3fg:
@misc{lin2024diffbpgenerativediffusion3d,
title={DiffBP: Generative Diffusion of 3D Molecules for Target Protein Binding},
author={Haitao Lin and Yufei Huang and Odin Zhang and Siqi Ma and Meng Liu and Xuanjing Li and Lirong Wu and Jishui Wang and Tingjun Hou and Stan Z. Li},
year={2024},
eprint={2211.11214},
archivePrefix={arXiv},
primaryClass={q-bio.BM},
url={https://arxiv.org/abs/2211.11214},
}
@inproceedings{
lin2023functionalgroupbased,
title={Functional-Group-Based Diffusion for Pocket-Specific Molecule Generation and Elaboration},
author={Haitao Lin and Yufei Huang and Odin Zhang and Yunfan Liu and Lirong Wu and Siyuan Li and Zhiyuan Chen and Stan Z. Li},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=lRG11M91dx}
}
Este projeto está licenciado nos termos da licença GPL-3.0.