CBGBench:複雑なバインディンググラフベンチマークは、生成ターゲット認識分子設計のベンチマークです。
論文[diffbp] |紙[d3fg] |ベンチマーク
注記
これは、論文「CBGBench:タンパク質分子結合グラフの空白を入力する」の公式コードリポジトリです。これは、単一のコード実装でターゲット認識分子設計を統一することを目的としています。これまで、以下に示すように7つの方法を含めています。
| モデル | 紙のリンク | github |
|---|---|---|
| pocket2mol | https://arxiv.org/abs/2205.07249 | https://github.com/pengxingang/pocket2mol |
| graphbp | https://arxiv.org/abs/2204.09410 | https://github.com/divelab/graphbp |
| diffsbdd | https://arxiv.org/abs/2210.13695 | https://github.com/arneschneuing/diffsbdd |
| diffbp | https://arxiv.org/abs/2211.11214 | 公式の実装は次のとおりです。 |
| TargetDiff | https://arxiv.org/abs/2303.03543 | https://github.com/guanjq/targetdiff |
| フラグ | https://openreview.net/forum?id=rq13Idf0f73 | https://github.com/zaixizhang/flag |
| D3FG | https://arxiv.org/abs/2306.13769 | 公式の実装は次のとおりです。 |
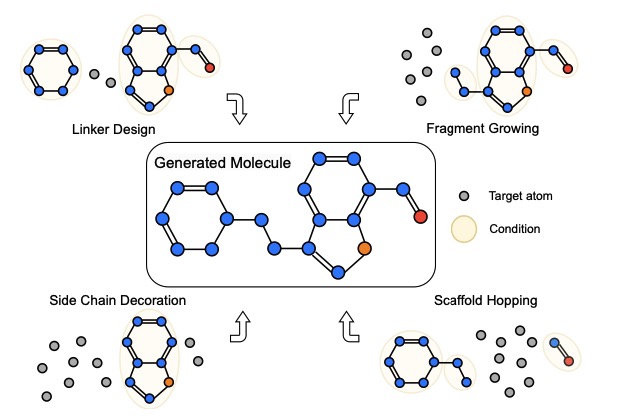
これらのモデルは、最初にde novo分子の生成のために確立されており、 linker design 、 fragment growing 、 scaffold hopping 、 side chain decorationなど、より多くのタスクを拡張します。
conda env create -f environment.yml
conda activate cbgbench
conda install pytorch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2 pytorch-cuda=11.8 -c pytorch -c nvidia
conda install pyg pytorch-scatter pytorch-cluster -c pyg
# install rdkit, efgs, obabel, etc.
pip install --use-pep517 EFGs
pip install biopython
pip install lxml
conda install rdkit openbabel tensorboard tqdm pyyaml easydict python-lmdb -c conda-forge
# install plip
mkdir tools
cd tools
git clone https://github.com/pharmai/plip.git
cd plip
python setup.py install
alias plip='python plip/plip/plipcmd.py'
cd ..
### Note that if there is an error in setup.py, it can be ignored as long as openbabel is installed.
# install docking tools
conda install -c conda-forge numpy swig boost-cpp sphinx sphinx_rtd_theme
python -m pip install git+https://github.com/Valdes-Tresanco-MS/AutoDockTools_py3
pip install meeko==0.1.dev3 scipy pdb2pqr vina
# If you are unable to install vina, you can try: conda install vina
# If you encounter the following error:
# ImportError: libtiff.so.5: cannot open shared object file: No such file or directory
# Please try the following steps to resolve it:
pip uninstall pillow
pip install pillow
(i)Googleドライブから処理されたデータセットをダウンロードします。各データセットについて、それは異なる方法とタスクに対応します。以下の表を参照して、使用する必要がある処理されたデータを見つけてください。 D3FGをトレーニングするために、最初に機能グループを生成してからリンカー原子を生成する2段階モデルであるため、2段モデル「D3FG_FG」と「D3FG_Linker」と命名し、トレーニングデータは異なります。
表:異なるメソッドとタスクに対応するデータファイル名。
| モデル | タスク | ファイル名 |
|---|---|---|
| pocket2mol | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| graphbp | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| diffsbdd | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| diffbp | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| TargetDiff | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| フラグ | de novo | data/pl_arfg/crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb |
| D3FG_FG | de novo | data/pl_fg/crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb |
| d3fg_linker | de novo | data/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| pocket2mol | 断片 | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| graphbp | 断片 | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| diffsbdd | 断片 | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| diffbp | 断片 | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| TargetDiff | 断片 | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| pocket2mol | リンカ | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| graphbp | リンカ | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| diffsbdd | リンカ | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| diffbp | リンカ | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| TargetDiff | リンカ | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| pocket2mol | 足場 | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| graphbp | 足場 | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| diffsbdd | 足場 | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| diffbp | 足場 | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| TargetDiff | 足場 | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| pocket2mol | サイドチェーン | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| graphbp | サイドチェーン | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| diffsbdd | サイドチェーン | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| diffbp | サイドチェーン | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| TargetDiff | サイドチェーン | data/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
(ii)データセットを./data dirにコピーします。完全なデータファイルディレクトリは次のようになります
- CGBBench
- data
- pl
- crossdocked_name2id.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- pl_arfg
- crossdocked_name2id_arfuncgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb
- pl_decomp
- crossdocked_name2id_frag.pt
- crossdocked_name2id_linker.pt
- crossdocked_name2id_scaffold.pt
- crossdocked_name2id_sidechain.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb
- pl_fg
- crossdocked_name2id_funcgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb
(i)TargetDiff Driveからcrossdocked_v1.1_rmsd1.0.tar.gzをダウンロードし、 ./raw_data/ raw_data/でそれをコピーします
mkdir raw_data
tar -xzvf crossdocked_v1.1_rmsd1.0.tar.gz ./raw_data
(ii)以下を実行します。
python ./scripts/extract_pockets.py --source raw_data/crossdocked_v1.1_rmsd1.0 --dest raw_data/crossdocked_v1.1_rmsd1.0_pocket10
(iii)トレーニングでは、各タスクのデータセットが準備されます。 Trainingを参照してください。
また、Googleドライブからプロセスターゲットファイルcase_studyをダウンロードして、それを./dataディレクトリにコピーすることもできます。
- CGBBench
- data
- case_study
- processed
- case_study_processed_fullatom.lmdb
- case_study_processed_funcgroup.lmdb
- case_study_name2id.pt
...
python train.py --config ./configs/{task}/train/{method}.yml --logdir ./logs/{task}/{method}
{タスク}は、 denovo 、 linker 、 frag 、 scaffold 、 sidechainに置き換えられ、{メソッド}はモデル名に置き換えることができます。次の表には、詳細なメソッドタスクペアと交換が示されています。
表:ゼロからトレーニングするために使用されるメソッドタスクペア。
| 方法 | タスク | {method} + {task} |
|---|---|---|
| pocket2mol | de novo | pocket2mol + denovo |
| graphbp | de novo | graphbp + denovo |
| diffsbdd | de novo | diffsbdd + denovo |
| diffbp | de novo | diffbp + denovo |
| TargetDiff | de novo | TargetDiff + Denovo |
| フラグ | de novo | フラグ +デノボ |
| D3FG | de novo | D3FG_FG + DENOVO; d3fg_linker + denovo |
| pocket2mol | リンカー設計 | pocket2mol +リンカー |
| graphbp | リンカー設計 | GraphBP +リンカー |
| diffsbdd | リンカー設計 | diffsbdd +リンカー |
| diffbp | リンカー設計 | diffbp +リンカー |
| TargetDiff | リンカー設計 | TargetDiff +リンカー |
| pocket2mol | 断片の成長 | pocket2mol + frag |
| graphbp | 断片の成長 | GraphBP +フラグ |
| diffsbdd | 断片の成長 | diffsbdd + frag |
| diffbp | 断片の成長 | diffbp + frag |
| TargetDiff | 断片の成長 | TargetDiff +フラグ |
| pocket2mol | 足場ホッピング | pocket2mol +足場 |
| graphbp | 足場ホッピング | GraphBP +足場 |
| diffsbdd | 足場ホッピング | diffsbdd +足場 |
| diffbp | 足場ホッピング | diffbp +足場 |
| TargetDiff | 足場ホッピング | TargetDiff +足場 |
| pocket2mol | サイドチェーンの装飾 | pocket2mol + sidechain |
| graphbp | サイドチェーンの装飾 | graphbp + sidechain |
| diffsbdd | サイドチェーンの装飾 | diffsbdd + sidechain |
| diffbp | サイドチェーンの装飾 | diffbp + sidechain |
| TargetDiff | サイドチェーンの装飾 | TargetDiff + Sidechain |
D3FGとフラグは拡張タスクと互換性がなく、D3FGは2段階トレーニング戦略を利用しているため、D3FGを自分でトレーニングしたい場合は、実行する必要があります。
python train.py --config ./configs/denovo/train/d3fg_fg.yml --logdir ./logs/denovo/d3fg_fg
python train.py --config ./configs/denovo/train/d3fg_linker.yml --logdir ./logs/denovo/d3fg_linker
TODO:前処理されたモデルを備えたテーブル
必要な前提条件の.ptチェックポイントをダウンロードし、 ./logs/にコピーして、次のディレクトリ構造につながります。
# TODO
- CBGBench
- logs
- denovo
- d3fg_fg
- pretrain
- checkpoints
- 951000.pt
- d3fg_linker
- pretrain
- checkpoints
- 4840000.pt
- diffbp
- pretrain
- checkpoints
- 4848000.pt
...
- frag
- diffbp
- pretrain
- checkpoints
- 476000.pt
...
...
モデルがトレーニングされたら、テストポケットにサンプルからサンプルを描画できます。
bash generate.sh --method {method} --task {task} --tag {tag} --checkpoint {ckpt_number}
コマンドでは、{method}および{task}ペアは、Method-taskペアの前のテーブルにあります。 {タグ}は、使用するチェックポイントに従って、 selftrainまたはpretrainに置き換える必要があります。 -checkpointパラメーターが番号なしで提供されている場合、最新の.ptファイルが自動的に見つかります。番号が提供されている場合、指定されたチェックポイントファイルを使用します。
たとえば、自己訓練されたTargetDiffモデルを使用してDe Novoデザインのテストセットでサンプルを生成する場合は、次を実行できます。
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint
または、100000番目のチェックポイントをテストする場合。
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint 100000
D3FG 2段階の生成戦略を使用しているため、コマンドは
bash generate.sh --method d3fg_fg --task denovo --tag {tag} --checkpoint # generate the functional groups
bash generate.sh --method d3fg_linker --task denovo --tag {tag} --checkpoint # generate the linkers
以下を実行します:
cd evaluate_scripts
bash evaluate.sh --method {method} --task {task} --tag {tag}
たとえば、自分でTargetDiffをトレーニングした場合は、実行してください
bash evaluate.sh --method targetdiff --task denovo --tag selftrain
bash evaluate.sh --method targetdiff --task frag --tag selftrain
bash evaluate.sh --method targetdiff --task linker --tag selftrain
bash evaluate.sh --method targetdiff --task scaffold --tag selftrain
bash evaluate.sh --method targetdiff --task sidechain --tag selftrain
または、冒険モデルをダウンロードした場合は、実行してください
cd evaluate_scripts
bash bash evaluate.sh --method {method} --task {task} --tag pretrained
前提条件のモデルを使用して、実際のターゲットで分子を柔軟に生成します。
3DSBDD、Decompdiff、Voxmol、Liganなどのモデルを追加します。
前提条件のモデルをリリースし、論文が公開されたら、実現可能性と再現性をテストします。
コードベースはハイタオリンによって初期化され、CBGBench V1は私たちの動機となっています。コードリポジトリのその後の実行は、Guojiang Zhaoと彼のDPテクノロジーチームによって管理されます。
リポジトリが調査やプロジェクトに役立つ場合は、参照としてベンチマークペーパーに引用してください。
@misc{lin2024cbgbench,
title={CBGBench: Fill in the Blank of Protein-Molecule Complex Binding Graph},
author={Haitao Lin and Guojiang Zhao and Odin Zhang and Yufei Huang and Lirong Wu and Zicheng Liu and Siyuan Li and Cheng Tan and Zhifeng Gao and Stan Z. Li},
year={2024},
eprint={2406.10840},
archivePrefix={arXiv},
}
diffbpとd3fgの以前の作品:
@misc{lin2024diffbpgenerativediffusion3d,
title={DiffBP: Generative Diffusion of 3D Molecules for Target Protein Binding},
author={Haitao Lin and Yufei Huang and Odin Zhang and Siqi Ma and Meng Liu and Xuanjing Li and Lirong Wu and Jishui Wang and Tingjun Hou and Stan Z. Li},
year={2024},
eprint={2211.11214},
archivePrefix={arXiv},
primaryClass={q-bio.BM},
url={https://arxiv.org/abs/2211.11214},
}
@inproceedings{
lin2023functionalgroupbased,
title={Functional-Group-Based Diffusion for Pocket-Specific Molecule Generation and Elaboration},
author={Haitao Lin and Yufei Huang and Odin Zhang and Yunfan Liu and Lirong Wu and Siyuan Li and Zhiyuan Chen and Stan Z. Li},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=lRG11M91dx}
}
このプロジェクトは、GPL-3.0ライセンスの条件に基づいてライセンスされています。