CBGBENCH: Le graphique de liaison complexe Benchmark est une référence pour la conception générative des molécules de la cible.
Papier [Diffbp] | Papier [d3fg] | Référence
Note
Il s'agit du référentiel de code officiel de l'article «CBGBench: remplissez le blanc du graphique de liaison des protéines molécules», qui vise à unifier la conception de molécules consacrées à la cible avec une implémentation de code unique. Jusqu'à présent, nous avons inclus 7 méthodes comme indiqué ci-dessous:
| Modèle | Lien papier | Github |
|---|---|---|
| Poche2mol | https://arxiv.org/abs/2205.07249 | https://github.com/pengxingang/pocket2mol |
| Graphbp | https://arxiv.org/abs/2204.09410 | https://github.com/divelab/graphbp |
| Diffsbdd | https://arxiv.org/abs/2210.13695 | https://github.com/arneschneuing/diffsbdd |
| Diffbp | https://arxiv.org/abs/2211.11214 | Voici la mise en œuvre officielle. |
| Targetdiff | https://arxiv.org/abs/2303.03543 | https://github.com/guanjq/targetdiff |
| DRAPEAU | https://openreview.net/forum?id=rq13idf0f73 | https://github.com/zaixizhang/flag |
| D3fg | https://arxiv.org/abs/2306.13769 | Voici la mise en œuvre officielle. |
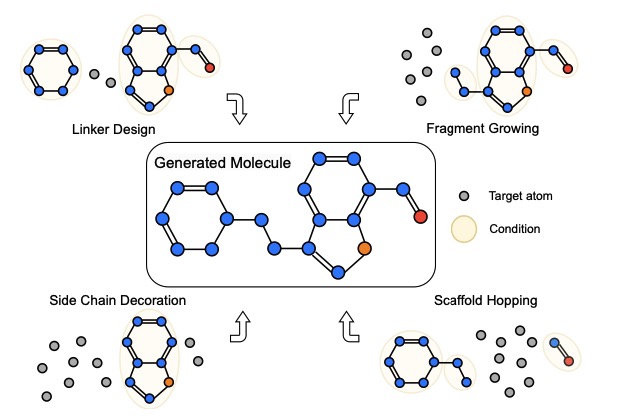
Ces modèles sont initialement établis pour la génération de molécules de novo , et nous étendons plus de tâches, notamment linker design , fragment growing , scaffold hopping et side chain decoration .
conda env create -f environment.yml
conda activate cbgbench
conda install pytorch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2 pytorch-cuda=11.8 -c pytorch -c nvidia
conda install pyg pytorch-scatter pytorch-cluster -c pyg
# install rdkit, efgs, obabel, etc.
pip install --use-pep517 EFGs
pip install biopython
pip install lxml
conda install rdkit openbabel tensorboard tqdm pyyaml easydict python-lmdb -c conda-forge
# install plip
mkdir tools
cd tools
git clone https://github.com/pharmai/plip.git
cd plip
python setup.py install
alias plip='python plip/plip/plipcmd.py'
cd ..
### Note that if there is an error in setup.py, it can be ignored as long as openbabel is installed.
# install docking tools
conda install -c conda-forge numpy swig boost-cpp sphinx sphinx_rtd_theme
python -m pip install git+https://github.com/Valdes-Tresanco-MS/AutoDockTools_py3
pip install meeko==0.1.dev3 scipy pdb2pqr vina
# If you are unable to install vina, you can try: conda install vina
# If you encounter the following error:
# ImportError: libtiff.so.5: cannot open shared object file: No such file or directory
# Please try the following steps to resolve it:
pip uninstall pillow
pip install pillow
(i) Téléchargez l'ensemble de données traité depuis Google Drive. Pour chaque ensemble de données, il correspond à différentes méthodes et tâches. Veuillez vous référer au tableau ci-dessous pour trouver les données traitées que vous devez utiliser. Notez que pour former D3FG, il s'agit d'un modèle en deux étapes qui génère d'abord des groupes fonctionnels, puis génère des atomes de liaison, nous avons donc nommé le modèle en deux étapes 'D3FG_FG' et 'D3FG_Linker', et les données de formation sont différentes pour eux.
Tableau: le nom du fichier de données correspondant à différentes méthodes et tâches.
| modèle | tâche | nom de fichier |
|---|---|---|
| Poche2mol | de novo | data / pl / crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| Graphbp | de novo | data / pl / crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| Diffsbdd | de novo | data / pl / crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| Diffbp | de novo | data / pl / crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| Targetdiff | de novo | data / pl / crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| DRAPEAU | de novo | DATA / PL_ARFG / CROSSDOCKED_V1.1_RMSD1.0_POCKING10_PROCEDED_ARFUNCGROUP.lmdb |
| D3fg_fg | de novo | DATA / PL_FG / CROSSDOCKED_V1.1_RMSD1.0_POCKING10_PROCEDED_FUNCGROUP.lmdb |
| D3fg_linker | de novo | DATA / PL / CROSSDOCKED_V1.1_RMSD1.0_POCKING10_PROCEDED_LINKER.lmdb |
| Poche2mol | fragment | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Graphbp | fragment | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Diffsbdd | fragment | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Diffbp | fragment | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Targetdiff | fragment | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| Poche2mol | linker | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Graphbp | linker | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Diffsbdd | linker | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Diffbp | linker | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Targetdiff | linker | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| Poche2mol | échafaud | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_sffold.lmdb |
| Graphbp | échafaud | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_sffold.lmdb |
| Diffsbdd | échafaud | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_sffold.lmdb |
| Diffbp | échafaud | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_sffold.lmdb |
| Targetdiff | échafaud | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_sffold.lmdb |
| Poche2mol | chaîne latérale | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Graphbp | chaîne latérale | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Diffsbdd | chaîne latérale | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Diffbp | chaîne latérale | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| Targetdiff | chaîne latérale | data / pl_decomp / crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
(ii) Copiez les ensembles de données sur ./data Dir, dans lesquels le répertoire de fichiers de données complet ressemblera à
- CGBBench
- data
- pl
- crossdocked_name2id.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- pl_arfg
- crossdocked_name2id_arfuncgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb
- pl_decomp
- crossdocked_name2id_frag.pt
- crossdocked_name2id_linker.pt
- crossdocked_name2id_scaffold.pt
- crossdocked_name2id_sidechain.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb
- pl_fg
- crossdocked_name2id_funcgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb
(i) Téléchargez crossdocked_v1.1_rmsd1.0.tar.gz de TargetDiff Drive, et copiez-le sur ./raw_data/ avec
mkdir raw_data
tar -xzvf crossdocked_v1.1_rmsd1.0.tar.gz ./raw_data
(ii) Exécutez ce qui suit:
python ./scripts/extract_pockets.py --source raw_data/crossdocked_v1.1_rmsd1.0 --dest raw_data/crossdocked_v1.1_rmsd1.0_pocket10
(iii) Dans la formation, l'ensemble de données sera préparé pour chaque tâche. Veuillez vous référer à Training .
En outre, vous pouvez télécharger les processus Targets File case_study à partir de Google Drive et le copier dans le répertoire ./data , qui mènera le DIR à
- CGBBench
- data
- case_study
- processed
- case_study_processed_fullatom.lmdb
- case_study_processed_funcgroup.lmdb
- case_study_name2id.pt
...
python train.py --config ./configs/{task}/train/{method}.yml --logdir ./logs/{task}/{method}
{Task} peut être remplacé par denovo , linker , frag , scaffold et sidechain , et {méthode} peut être remplacé par le nom du modèle. Le tableau suivant donne les paires de tâches méthode détaillées et le remplacement.
Tableau: Paires de tâches méthode utilisées pour s'entraîner à partir de zéro.
| Méthode | Tâche | {méthode} + {tâche} |
|---|---|---|
| Poche2mol | de novo | Pocket2Mol + Denovo |
| Graphbp | de novo | GraphBP + Denovo |
| Diffsbdd | de novo | diffsbdd + denovo |
| Diffbp | de novo | Diffbp + Denovo |
| Targetdiff | de novo | TargetDiff + Denovo |
| DRAPEAU | de novo | Flag + Denovo |
| D3fg | de novo | d3fg_fg + denovo; d3fg_linker + denovo |
| Poche2mol | conception de liens | Pocket2Mol + linker |
| Graphbp | conception de liens | graphbp + linker |
| Diffsbdd | conception de liens | diffsbdd + linker |
| Diffbp | conception de liens | diffbp + linker |
| Targetdiff | conception de liens | TargetDiff + Linker |
| Poche2mol | fragment croissant | Pocket2Mol + frag |
| Graphbp | fragment croissant | graphbp + frag |
| Diffsbdd | fragment croissant | diffsbdd + frag |
| Diffbp | fragment croissant | diffbp + frag |
| Targetdiff | fragment croissant | TargetDiff + Frag |
| Poche2mol | saut d'échafaudage | Pocket2mol + échafaudage |
| Graphbp | saut d'échafaudage | GraphBP + échafaudage |
| Diffsbdd | saut d'échafaudage | diffsbdd + échafaudage |
| Diffbp | saut d'échafaudage | Diffbp + échafaudage |
| Targetdiff | saut d'échafaudage | TargetDiff + échafaudage |
| Poche2mol | décoration de la chaîne latérale | Pocket2Mol + Sidechain |
| Graphbp | décoration de la chaîne latérale | GraphBP + Sidechain |
| Diffsbdd | décoration de la chaîne latérale | diffsbdd + sidechain |
| Diffbp | décoration de la chaîne latérale | Diffbp + Sidechain |
| Targetdiff | décoration de la chaîne latérale | TargetDiff + Sidechain |
Notez que D3FG et Flag ne sont pas compatibles avec les tâches étendues, et D3FG utilise des stratégies de formation en 2 étapes, donc si vous souhaitez former D3FG vous-même, vous devez courir:
python train.py --config ./configs/denovo/train/d3fg_fg.yml --logdir ./logs/denovo/d3fg_fg
python train.py --config ./configs/denovo/train/d3fg_linker.yml --logdir ./logs/denovo/d3fg_linker
TODO: table avec des modèles pré-entraînés
Téléchargez les points de contrôle .pt pré-entraînés dont vous avez besoin et copiez-les dans ./logs/ , ce qui mènera à la structure du répertoire suivant:
# TODO
- CBGBench
- logs
- denovo
- d3fg_fg
- pretrain
- checkpoints
- 951000.pt
- d3fg_linker
- pretrain
- checkpoints
- 4840000.pt
- diffbp
- pretrain
- checkpoints
- 4848000.pt
...
- frag
- diffbp
- pretrain
- checkpoints
- 476000.pt
...
...
Une fois le modèle formé, vous pouvez les tirer des échantillons sur les poches de test, avec les suivantes:
bash generate.sh --method {method} --task {task} --tag {tag} --checkpoint {ckpt_number}
Dans la commande, la paire {méthode} et {tâche} se trouve dans l'ancienne table sur des paires de méthode. {TAG} doit être remplacé par selftrain ou pretrain , selon les points de contrôle que vous utilisez. Si le paramètre --Checkpoint est fourni sans un nombre, il trouvera automatiquement le dernier fichier .pt . Si un nombre est fourni, il utilisera le fichier de point de contrôle spécifié.
Par exemple, si vous souhaitez générer des échantillons sur l'ensemble de tests pour la conception de novo avec le modèle TargetDiff auto-formé , vous pouvez exécuter:
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint
Ou si vous souhaitez tester le point de contrôle du 100000 h.
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint 100000
Notez que D3FG utilise une stratégie de génération en deux étapes, donc la commande doit être
bash generate.sh --method d3fg_fg --task denovo --tag {tag} --checkpoint # generate the functional groups
bash generate.sh --method d3fg_linker --task denovo --tag {tag} --checkpoint # generate the linkers
Exécutez ce qui suit:
cd evaluate_scripts
bash evaluate.sh --method {method} --task {task} --tag {tag}
Par exemple, si vous avez formé TargetDiff par vous-même, courez
bash evaluate.sh --method targetdiff --task denovo --tag selftrain
bash evaluate.sh --method targetdiff --task frag --tag selftrain
bash evaluate.sh --method targetdiff --task linker --tag selftrain
bash evaluate.sh --method targetdiff --task scaffold --tag selftrain
bash evaluate.sh --method targetdiff --task sidechain --tag selftrain
Ou si vous avez téléchargé des modèles pré-entraînés, veuillez courir
cd evaluate_scripts
bash bash evaluate.sh --method {method} --task {task} --tag pretrained
Utilisez les modèles pré-entraînés pour générer des molécules sur des cibles du monde réel de manière flexible.
Incluez plus de modèles, tels que 3DSBDD, Decompdiff, Voxmol et Ligan.
Libérez les modèles pré-entraînés, testez la faisabilité et la reproductivité une fois le document publié.
La base de code est initialisée par Haitao Lin, avec le CBGBench V1 comme motivation; Le fonctionnement ultérieur du référentiel de code sera géré par Guojiang Zhao et son équipe de technologie DP.
Si vous trouvez que le référentiel est utile à vos recherches ou projets, veuillez citer notre article de référence comme référence:
@misc{lin2024cbgbench,
title={CBGBench: Fill in the Blank of Protein-Molecule Complex Binding Graph},
author={Haitao Lin and Guojiang Zhao and Odin Zhang and Yufei Huang and Lirong Wu and Zicheng Liu and Siyuan Li and Cheng Tan and Zhifeng Gao and Stan Z. Li},
year={2024},
eprint={2406.10840},
archivePrefix={arXiv},
}
Et nos anciennes œuvres de Diffbp et D3FG:
@misc{lin2024diffbpgenerativediffusion3d,
title={DiffBP: Generative Diffusion of 3D Molecules for Target Protein Binding},
author={Haitao Lin and Yufei Huang and Odin Zhang and Siqi Ma and Meng Liu and Xuanjing Li and Lirong Wu and Jishui Wang and Tingjun Hou and Stan Z. Li},
year={2024},
eprint={2211.11214},
archivePrefix={arXiv},
primaryClass={q-bio.BM},
url={https://arxiv.org/abs/2211.11214},
}
@inproceedings{
lin2023functionalgroupbased,
title={Functional-Group-Based Diffusion for Pocket-Specific Molecule Generation and Elaboration},
author={Haitao Lin and Yufei Huang and Odin Zhang and Yunfan Liu and Lirong Wu and Siyuan Li and Zhiyuan Chen and Stan Z. Li},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=lRG11M91dx}
}
Ce projet est concédé sous licence de la licence GPL-3.0.