CBGBENCH : 복잡한 바인딩 그래프 벤치 마크는 생성 목표 인식 분자 설계의 벤치 마크입니다.
종이 [diffbp] | 논문 [d3fg] | 기준
메모
이것은 단일 코드 구현으로 목표 인식 분자 설계를 통합하는 것을 목표로하는 논문 'CBGBENCH : 단백질-분자 결합 그래프 블랭크'의 공식 코드 저장소입니다. 지금까지는 다음과 같이 7 가지 방법을 포함했습니다.
| 모델 | 종이 링크 | github |
|---|---|---|
| pocket2mol | https://arxiv.org/abs/2205.07249 | https://github.com/pengxingang/pocket2mol |
| 그래프 BP | https://arxiv.org/abs/2204.09410 | https://github.com/divelab/graphbp |
| diffsbdd | https://arxiv.org/abs/2210.13695 | https://github.com/arneschneuing/diffsbdd |
| diffbp | https://arxiv.org/abs/2211.11214 | 공식 구현은 다음과 같습니다. |
| TargetDiff | https://arxiv.org/abs/2303.03543 | https://github.com/guanjq/targetdiff |
| 깃발 | https://openreview.net/forum?id=rq13idf0f73 | https://github.com/zaixizhang/flag |
| D3FG | https://arxiv.org/abs/2306.13769 | 공식 구현은 다음과 같습니다. |
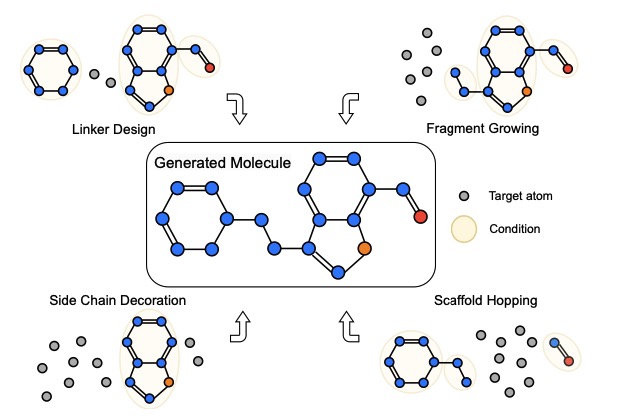
이 모델은 처음에 de novo 분자 생성을 위해 확립되었으며 linker design , fragment growing , scaffold hopping 및 side chain decoration 포함한 더 많은 작업을 확장합니다.
conda env create -f environment.yml
conda activate cbgbench
conda install pytorch==2.0.1 torchvision==0.15.2 torchaudio==2.0.2 pytorch-cuda=11.8 -c pytorch -c nvidia
conda install pyg pytorch-scatter pytorch-cluster -c pyg
# install rdkit, efgs, obabel, etc.
pip install --use-pep517 EFGs
pip install biopython
pip install lxml
conda install rdkit openbabel tensorboard tqdm pyyaml easydict python-lmdb -c conda-forge
# install plip
mkdir tools
cd tools
git clone https://github.com/pharmai/plip.git
cd plip
python setup.py install
alias plip='python plip/plip/plipcmd.py'
cd ..
### Note that if there is an error in setup.py, it can be ignored as long as openbabel is installed.
# install docking tools
conda install -c conda-forge numpy swig boost-cpp sphinx sphinx_rtd_theme
python -m pip install git+https://github.com/Valdes-Tresanco-MS/AutoDockTools_py3
pip install meeko==0.1.dev3 scipy pdb2pqr vina
# If you are unable to install vina, you can try: conda install vina
# If you encounter the following error:
# ImportError: libtiff.so.5: cannot open shared object file: No such file or directory
# Please try the following steps to resolve it:
pip uninstall pillow
pip install pillow
(i) Google 드라이브에서 처리 된 데이터 세트를 다운로드하십시오. 각 데이터 세트에 대해 다른 방법과 작업에 해당합니다. 사용해야 할 처리 된 데이터를 찾으려면 아래 표를 참조하십시오. D3FG를 훈련시키기 위해서는 먼저 기능 그룹을 생성 한 다음 링커 원자를 생성하는 2 단계 모델이므로 2 단계 모델 'D3FG_FG'및 'D3FG_LINKER'를 명명했으며 교육 데이터가 다릅니다.
표 : 다른 방법 및 작업에 해당하는 데이터 파일 이름.
| 모델 | 일 | 파일 이름 |
|---|---|---|
| pocket2mol | 드 노보 | 데이터/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| 그래프 BP | 드 노보 | 데이터/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| diffsbdd | 드 노보 | 데이터/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| diffbp | 드 노보 | 데이터/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| TargetDiff | 드 노보 | 데이터/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb |
| 깃발 | 드 노보 | 데이터/pl_arfg/crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb |
| D3FG_FG | 드 노보 | 데이터/pl_fg/crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb |
| D3FG_LINKER | 드 노보 | 데이터/pl/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| pocket2mol | 파편 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| 그래프 BP | 파편 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| diffsbdd | 파편 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| diffbp | 파편 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| TargetDiff | 파편 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb |
| pocket2mol | 링커 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| 그래프 BP | 링커 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| diffsbdd | 링커 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| diffbp | 링커 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| TargetDiff | 링커 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb |
| pocket2mol | 발판 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| 그래프 BP | 발판 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| diffsbdd | 발판 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| diffbp | 발판 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| TargetDiff | 발판 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb |
| pocket2mol | 사이드 체인 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| 그래프 BP | 사이드 체인 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| diffsbdd | 사이드 체인 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| diffbp | 사이드 체인 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
| TargetDiff | 사이드 체인 | 데이터/pl_decomp/crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb |
(ii) 데이터 세트를 ./data dir로 복사하여 전체 데이터 파일 디렉토리가
- CGBBench
- data
- pl
- crossdocked_name2id.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_fullatom.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- pl_arfg
- crossdocked_name2id_arfuncgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_arfuncgroup.lmdb
- pl_decomp
- crossdocked_name2id_frag.pt
- crossdocked_name2id_linker.pt
- crossdocked_name2id_scaffold.pt
- crossdocked_name2id_sidechain.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_frag.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_linker.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_scaffold.lmdb
- crossdocked_v1.1_rmsd1.0_pocket10_processed_sidechain.lmdb
- pl_fg
- crossdocked_name2id_funcgroup.pt
- crossdocked_v1.1_rmsd1.0_pocket10_processed_funcgroup.lmdb
(i) TargetDiff Drive에서 crossdocked_v1.1_rmsd1.0.tar.gz 다운로드하고 ./raw_data/ 로 복사하십시오.
mkdir raw_data
tar -xzvf crossdocked_v1.1_rmsd1.0.tar.gz ./raw_data
(ii) 다음을 실행하십시오.
python ./scripts/extract_pockets.py --source raw_data/crossdocked_v1.1_rmsd1.0 --dest raw_data/crossdocked_v1.1_rmsd1.0_pocket10
(iii) 훈련시 데이터 세트는 각 작업에 대해 준비됩니다. Training 을 참조하십시오.
또한 Google 드라이브에서 프로세스 대상 파일 case_study 다운로드하여 ./data 디렉토리로 복사하여 DIR을 이끌어 낼 수 있습니다.
- CGBBench
- data
- case_study
- processed
- case_study_processed_fullatom.lmdb
- case_study_processed_funcgroup.lmdb
- case_study_name2id.pt
...
python train.py --config ./configs/{task}/train/{method}.yml --logdir ./logs/{task}/{method}
{task}는 denovo , linker , frag , scaffold 및 sidechain 으로 대체 될 수 있으며 {Method}는 모델 이름으로 대체 할 수 있습니다. 다음 표는 자세한 방법 직접 쌍과 교체를 제공합니다.
표 : 처음부터 훈련하는 데 사용되는 방법 작업 쌍.
| 방법 | 일 | {method} + {task} |
|---|---|---|
| pocket2mol | 드 노보 | 포켓 2mol + denovo |
| 그래프 BP | 드 노보 | GraphBP + Denovo |
| diffsbdd | 드 노보 | diffsbdd + denovo |
| diffbp | 드 노보 | diffbp + denovo |
| TargetDiff | 드 노보 | TargetDiff + Denovo |
| 깃발 | 드 노보 | 플래그 + 데노 보 |
| D3FG | 드 노보 | D3FG_FG + DENOVO; D3FG_LINKER + DENOVO |
| pocket2mol | 링커 디자인 | 포켓 2mol + 링커 |
| 그래프 BP | 링커 디자인 | GraphBP + 링커 |
| diffsbdd | 링커 디자인 | diffsbdd + 링커 |
| diffbp | 링커 디자인 | diffbp + 링커 |
| TargetDiff | 링커 디자인 | TargetDiff + 링커 |
| pocket2mol | 파편 성장 | 포켓 2mol + 조각 |
| 그래프 BP | 파편 성장 | GraphBP + 조각 |
| diffsbdd | 파편 성장 | diffsbdd + 단편 |
| diffbp | 파편 성장 | diffbp + 조각 |
| TargetDiff | 파편 성장 | TargetDiff + 조각 |
| pocket2mol | 발판 호핑 | 포켓 2mol + 스캐 폴드 |
| 그래프 BP | 발판 호핑 | GraphBP + 스캐 폴드 |
| diffsbdd | 발판 호핑 | diffsbdd + 스캐 폴드 |
| diffbp | 발판 호핑 | diffbp + 스캐 폴드 |
| TargetDiff | 발판 호핑 | TargetDiff + 스캐 폴드 |
| pocket2mol | 사이드 체인 장식 | 포켓 2mol + 사이드 체인 |
| 그래프 BP | 사이드 체인 장식 | GraphBP + 사이드 체인 |
| diffsbdd | 사이드 체인 장식 | diffsbdd + seedechain |
| diffbp | 사이드 체인 장식 | diffbp + sidechain |
| TargetDiff | 사이드 체인 장식 | TargetDiff + SeedeCain |
D3FG 및 플래그는 확장 작업과 호환되지 않으며 D3FG는 2 단계 훈련 전략을 사용하므로 D3FG를 직접 훈련하려면 다음을 수행해야합니다.
python train.py --config ./configs/denovo/train/d3fg_fg.yml --logdir ./logs/denovo/d3fg_fg
python train.py --config ./configs/denovo/train/d3fg_linker.yml --logdir ./logs/denovo/d3fg_linker
TODO : 사전에 사전 모델이있는 테이블
필요한 사기가 필요한 .pt 체크 포인트를 다운로드하여 ./logs/ 에 복사하면 다음 디렉토리 구조로 이어집니다.
# TODO
- CBGBench
- logs
- denovo
- d3fg_fg
- pretrain
- checkpoints
- 951000.pt
- d3fg_linker
- pretrain
- checkpoints
- 4840000.pt
- diffbp
- pretrain
- checkpoints
- 4848000.pt
...
- frag
- diffbp
- pretrain
- checkpoints
- 476000.pt
...
...
모델이 훈련되면 테스트 포켓에 다음과 같이 샘플을 그릴 수 있습니다.
bash generate.sh --method {method} --task {task} --tag {tag} --checkpoint {ckpt_number}
명령에서 {method} 및 {task} 쌍은 메소드 작업 쌍의 이전 테이블에서 찾을 수 있습니다. {TAG}는 사용하는 체크 포인트에 따라 selftrain 또는 pretrain 으로 교체해야합니다. -checkpoint 매개 변수가 숫자없이 제공되면 최신 .pt 파일을 자동으로 찾습니다. 숫자가 제공되면 지정된 체크 포인트 파일을 사용합니다.
예를 들어, 자체 훈련 된 TargetDiff 모델을 사용하여 De Novo Design의 테스트 세트에서 샘플을 생성하려면 다음을 실행할 수 있습니다.
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint
또는 100000-th의 검문소를 테스트하려는 경우.
bash generate.sh --method targetdiff --task denovo --tag selftrain --checkpoint 100000
D3FG 2 단계 생성 전략을 사용하므로 명령은
bash generate.sh --method d3fg_fg --task denovo --tag {tag} --checkpoint # generate the functional groups
bash generate.sh --method d3fg_linker --task denovo --tag {tag} --checkpoint # generate the linkers
다음을 실행하십시오.
cd evaluate_scripts
bash evaluate.sh --method {method} --task {task} --tag {tag}
예를 들어, 직접 TargetDiff를 훈련 한 경우
bash evaluate.sh --method targetdiff --task denovo --tag selftrain
bash evaluate.sh --method targetdiff --task frag --tag selftrain
bash evaluate.sh --method targetdiff --task linker --tag selftrain
bash evaluate.sh --method targetdiff --task scaffold --tag selftrain
bash evaluate.sh --method targetdiff --task sidechain --tag selftrain
또는 사전 치료 된 모델을 다운로드 한 경우 실행하십시오
cd evaluate_scripts
bash bash evaluate.sh --method {method} --task {task} --tag pretrained
사전에 걸린 모델을 사용하여 실제 대상에서 분자를 생성하십시오.
3DSBDD, DecompDiff, Voxmol 및 Ligan과 같은 더 많은 모델을 포함하십시오.
사전에 걸린 모델을 해제하고 논문이 게시되면 타당성과 재현성을 테스트하십시오.
코드베이스는 Haitao Lin에 의해 초기화되며 CBGBENCH V1은 동기 부여입니다. 코드 저장소의 후속 실행은 Guojiang Zhao와 그의 DP 기술 팀이 관리합니다.
저장소가 연구 나 프로젝트에 도움이된다면 벤치 마크 논문을 참조로 인용하십시오.
@misc{lin2024cbgbench,
title={CBGBench: Fill in the Blank of Protein-Molecule Complex Binding Graph},
author={Haitao Lin and Guojiang Zhao and Odin Zhang and Yufei Huang and Lirong Wu and Zicheng Liu and Siyuan Li and Cheng Tan and Zhifeng Gao and Stan Z. Li},
year={2024},
eprint={2406.10840},
archivePrefix={arXiv},
}
그리고 Diffbp와 d3fg의 이전 작품 :
@misc{lin2024diffbpgenerativediffusion3d,
title={DiffBP: Generative Diffusion of 3D Molecules for Target Protein Binding},
author={Haitao Lin and Yufei Huang and Odin Zhang and Siqi Ma and Meng Liu and Xuanjing Li and Lirong Wu and Jishui Wang and Tingjun Hou and Stan Z. Li},
year={2024},
eprint={2211.11214},
archivePrefix={arXiv},
primaryClass={q-bio.BM},
url={https://arxiv.org/abs/2211.11214},
}
@inproceedings{
lin2023functionalgroupbased,
title={Functional-Group-Based Diffusion for Pocket-Specific Molecule Generation and Elaboration},
author={Haitao Lin and Yufei Huang and Odin Zhang and Yunfan Liu and Lirong Wu and Siyuan Li and Zhiyuan Chen and Stan Z. Li},
booktitle={Thirty-seventh Conference on Neural Information Processing Systems},
year={2023},
url={https://openreview.net/forum?id=lRG11M91dx}
}
이 프로젝트는 GPL-3.0 라이센스의 조건에 따라 라이센스가 부여됩니다.